
Wykład:
Genomika a proteomika
Patofizjologia
III rok Wydziału Lekarskiego
Rok akademicki 2005/2006
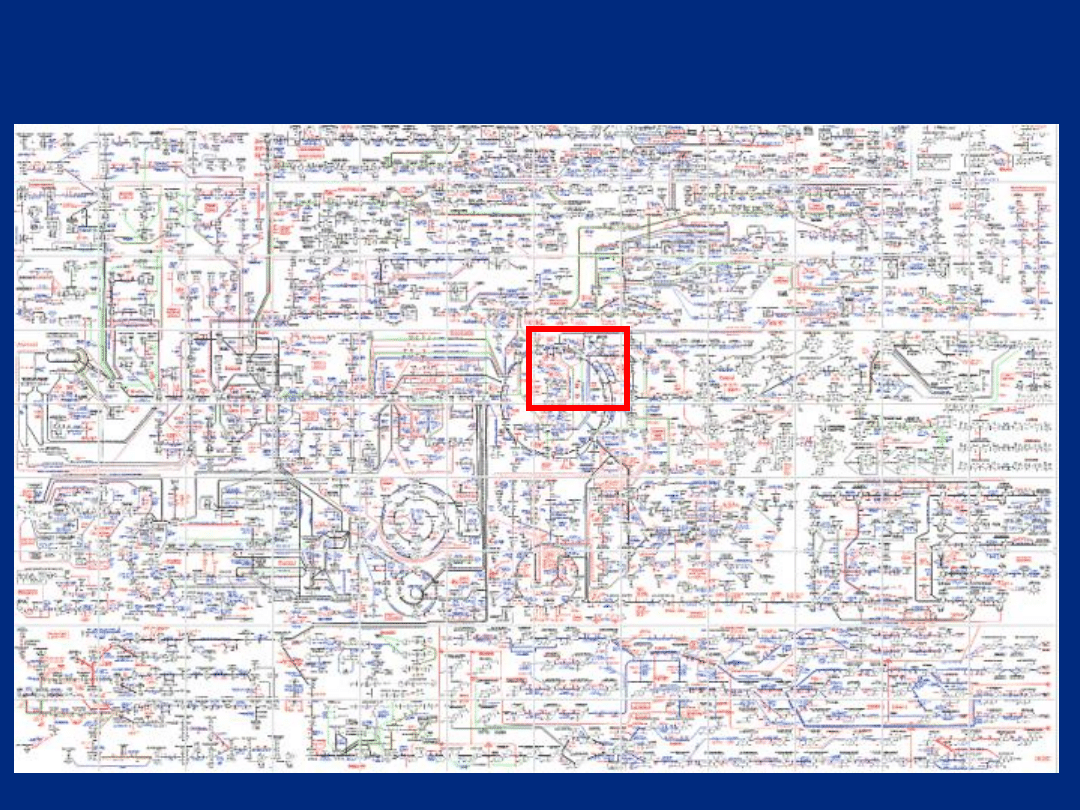
Dotychczasowe metody analizy nie
Dotychczasowe metody analizy nie
pozwalają ocenić całościowo stanu komórki
pozwalają ocenić całościowo stanu komórki
Źródło:
Źródło:
Wykład B. Jarząb;
Wykład B. Jarząb;
h
h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
Stan komórki jest wypadkową zachodzących
Stan komórki jest wypadkową zachodzących
w niej złożonych procesów metabolicznych
w niej złożonych procesów metabolicznych
Źródło: Wykład B. Jarząb;
http://www.genomika.pl/prezentacje/technologia_pliki/frame.htm

•
Geny funkcjonują w sieci wzajemnych zależności
Geny funkcjonują w sieci wzajemnych zależności
•
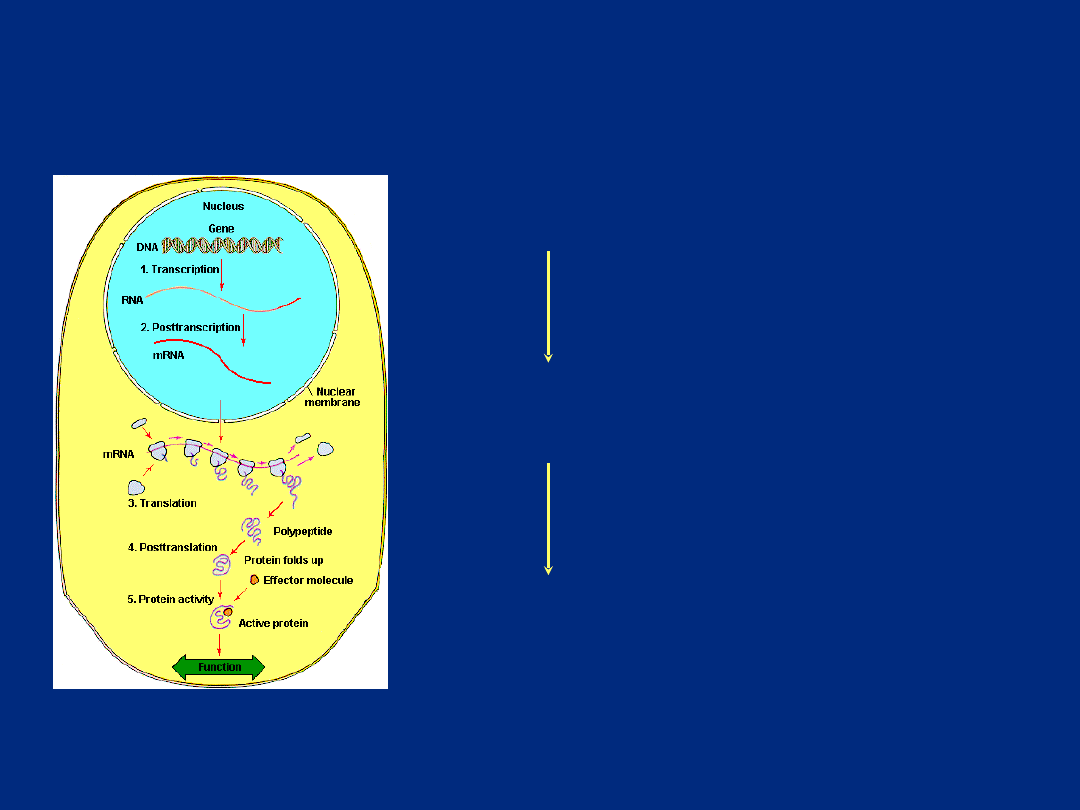
Aktywacja genu w komórce jest równoznaczna z syntezą
Aktywacja genu w komórce jest równoznaczna z syntezą
odpowiedniego mRNA (transkryptu), na matrycy którego powstaje
odpowiedniego mRNA (transkryptu), na matrycy którego powstaje
białko
białko
•
Wykrycie mRNA jest dowodem ekspresji genu, a liczba kopii mRNA
Wykrycie mRNA jest dowodem ekspresji genu, a liczba kopii mRNA
jest miarą ekspresji genu
jest miarą ekspresji genu
•
Ekspresja genów zmienia się w odpowiedzi na sygnały zewnętrzne
Ekspresja genów zmienia się w odpowiedzi na sygnały zewnętrzne
i wewnętrzne
i wewnętrzne
•
Tradycyjna biologia molekularna zakłada badanie jednego genu
Tradycyjna biologia molekularna zakłada badanie jednego genu
w jednym doświadczeniu, co uniemożliwia uzyskanie obrazu
w jednym doświadczeniu, co uniemożliwia uzyskanie obrazu
całościowego.
całościowego.
Nowy rozdział w biologii
Nowy rozdział w biologii
molekularnej
molekularnej
Źródło: Wykład B. Jarząb;
http://www.genomika.pl/prezentacje/technologia_pliki/frame.htm

genomika
genom
proteomi
ka
proteom
degradom
transkrypt
om
interakto
m
fenom
Genomika a proteomika

HUGO
Human Genome
Organisation
(1988)
HUPO
Human Proteome
Organisation
(2001)

•
Analiza northern-blotting
Analiza northern-blotting
–
czuła, ale półilościowa
czuła, ale półilościowa
–
pracochłonna, tylko kilka genów równocześnie
pracochłonna, tylko kilka genów równocześnie
•
Q-PCR (TaqMan)
Q-PCR (TaqMan)
–
ilościowa analiza metodą PCR w czasie rzeczywistym
ilościowa analiza metodą PCR w czasie rzeczywistym
–
drogie sondy, kilka-kilkanaście genów na raz
drogie sondy, kilka-kilkanaście genów na raz
•
Differential Display
Differential Display
(porównanie ekspresji losowo namnożonych fragmentów RNA)
(porównanie ekspresji losowo namnożonych fragmentów RNA)
–
analizuje wiele genów
analizuje wiele genów
–
ryzyko wyników fałszywie dodatnich
ryzyko wyników fałszywie dodatnich
•
Analiza dot blot (macroarrays)
Analiza dot blot (macroarrays)
–
ograniczona czułość
ograniczona czułość
Wszystkie metody oparte są na hybrydyzacji badanego mRNA
Wszystkie metody oparte są na hybrydyzacji badanego mRNA
z sondą o znanej sekwencji
z sondą o znanej sekwencji
•
SAGE (sekwencyjna analiza ekspresji genów)
SAGE (sekwencyjna analiza ekspresji genów)
–
czuła, analizuje wiele genów, również o nieznanej sekwencji
czuła, analizuje wiele genów, również o nieznanej sekwencji
–
wymaga znacznych umiejętności technicznych
wymaga znacznych umiejętności technicznych
Jak dotychczas badano ekspresję genów?
Jak dotychczas badano ekspresję genów?
Źródło: Wykład B. Jarząb;
http://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
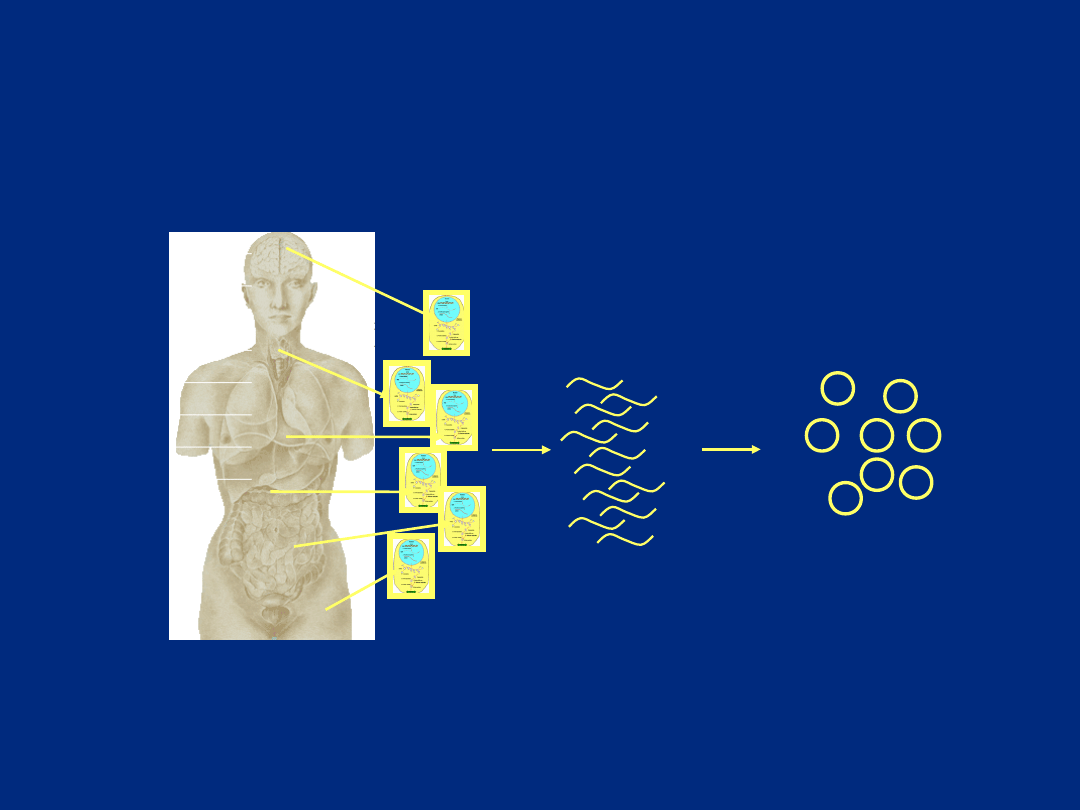
Chcemy badać komórkę poprzez jednoczesną
Chcemy badać komórkę poprzez jednoczesną
analizę
analizę
stanu wszystkich jej elementów (genów lub białek)
stanu wszystkich jej elementów (genów lub białek)
GENOMIKA
GENOMIKA
PROTEOMIKA
PROTEOMIKA
„
„
TRANSKRYPTOMIKA”
TRANSKRYPTOMIKA”
geny (DNA)
geny (DNA)
transkrypty genów
transkrypty genów
(mRNA)
(mRNA)
białka
białka
Źródło:
Źródło:
Wykład B. Jarząb;
Wykład B. Jarząb;
h
h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
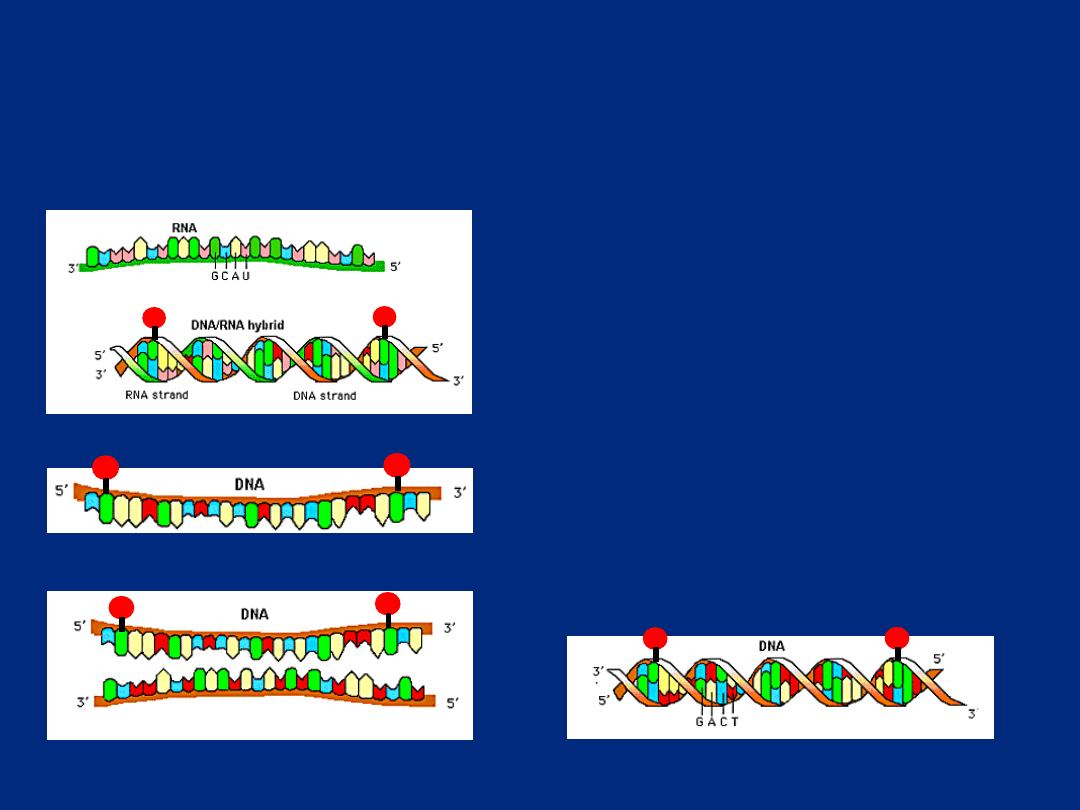
Od mRNA do cDNA
Od mRNA do cDNA
Komórki z różnych tkanek
Komórki z różnych tkanek
mRNA wyizolowane
mRNA wyizolowane
z komórek
z komórek
klony cDNA otrzymane
klony cDNA otrzymane
z cząsteczek mRNA
z cząsteczek mRNA
Źródło:
Źródło:
Wykład B. Jarząb; h
Wykład B. Jarząb; h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
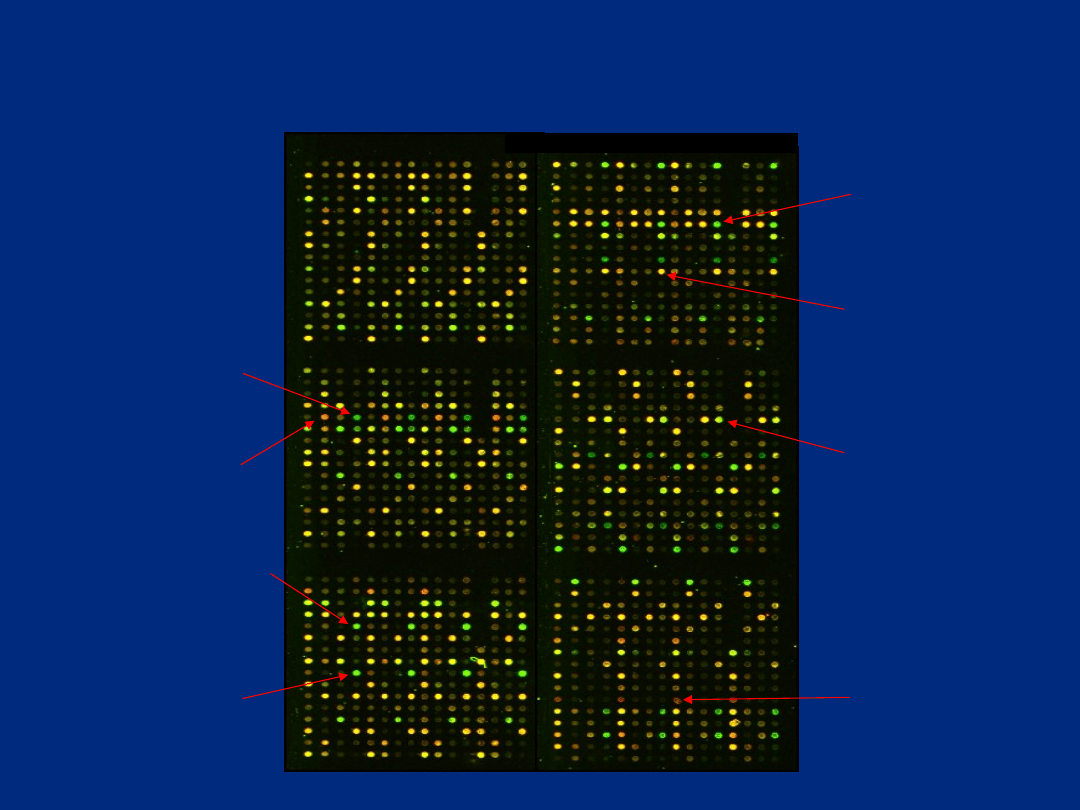
Struktura mikromacierzy; określona liczba sond naniesionych
Struktura mikromacierzy; określona liczba sond naniesionych
w sposób uporządkowany; badanie wielu genów równocześnie
w sposób uporządkowany; badanie wielu genów równocześnie
GST mu
GST mu
CYP1A1
CYP1A1
Transferrin
Transferrin
Albumin
Albumin
CYP3A
CYP3A
1
1
-Acid Glycoprotein
-Acid Glycoprotein
Amyloid A
Amyloid A
CYP1A2
CYP1A2
Źródło:
Źródło:
Wykład B. Jarząb; h
Wykład B. Jarząb; h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
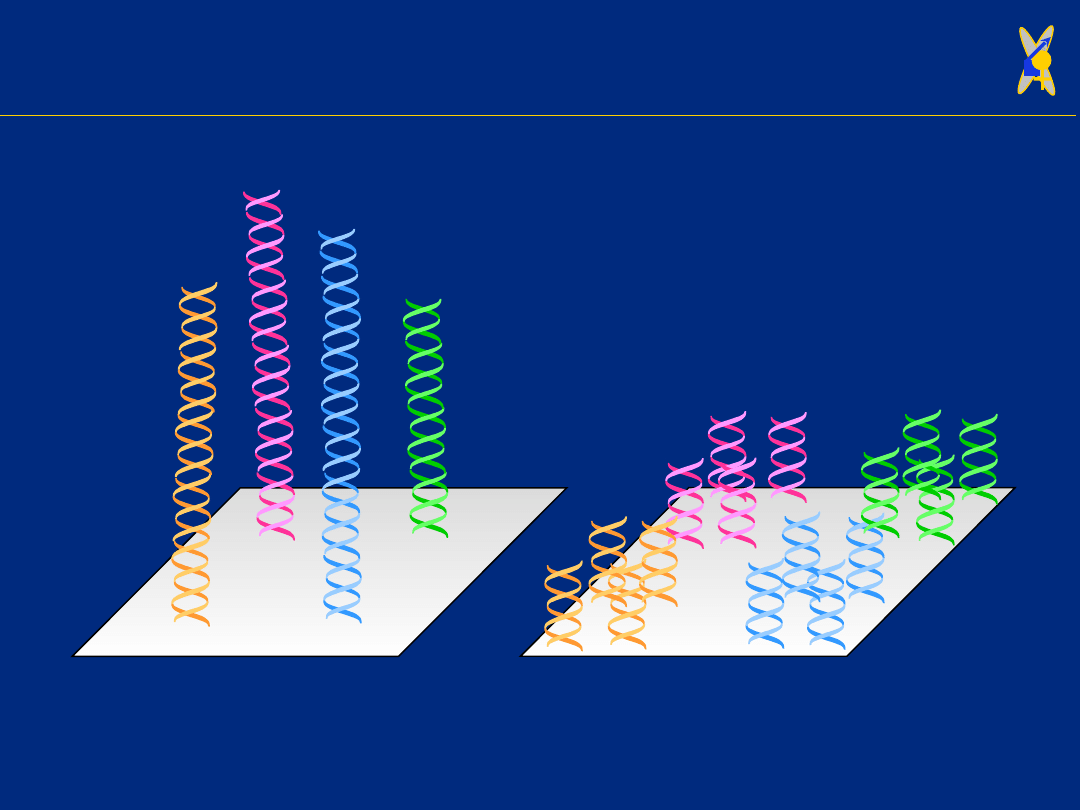
Dwa główne typy mikromacierzy DNA:
Dwa główne typy mikromacierzy DNA:
cDNA i oligonukleotydowe
cDNA i oligonukleotydowe
macierz cDNA
macierz cDNA
macierz oligonukleotydowa
macierz oligonukleotydowa
Źródło: Wykład B. Jarząb;
http://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
Źródło:
http://microarray.republika.pl/index.htm
l
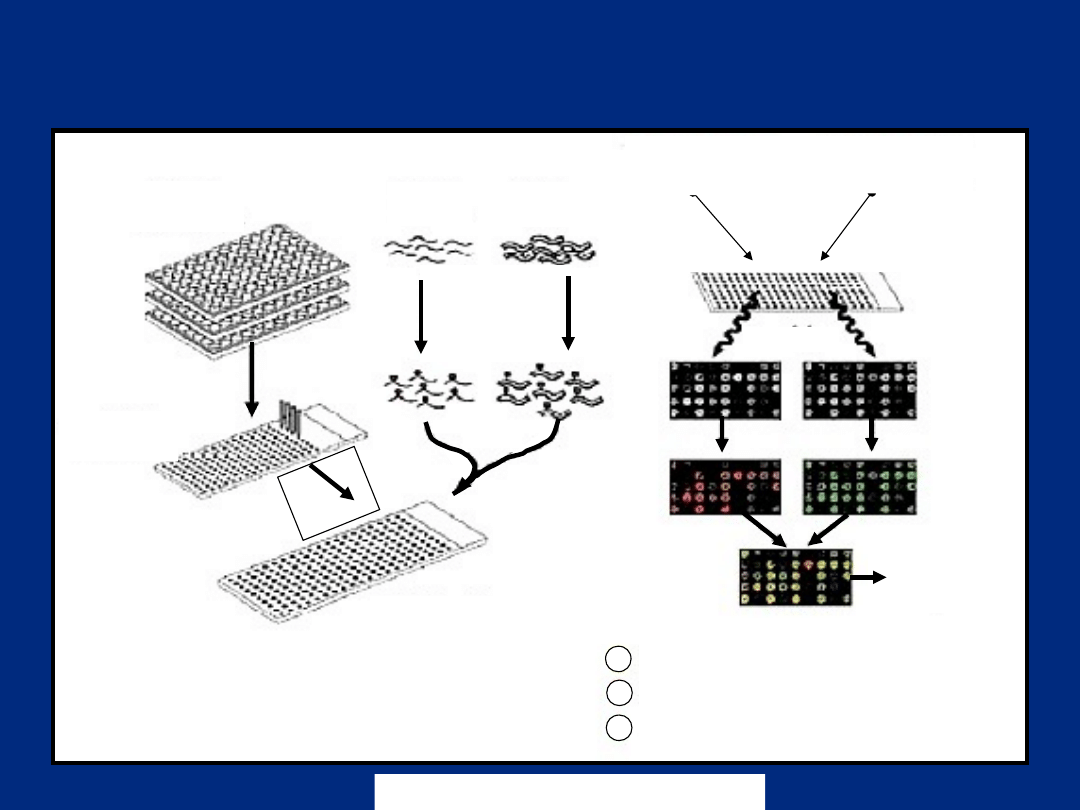
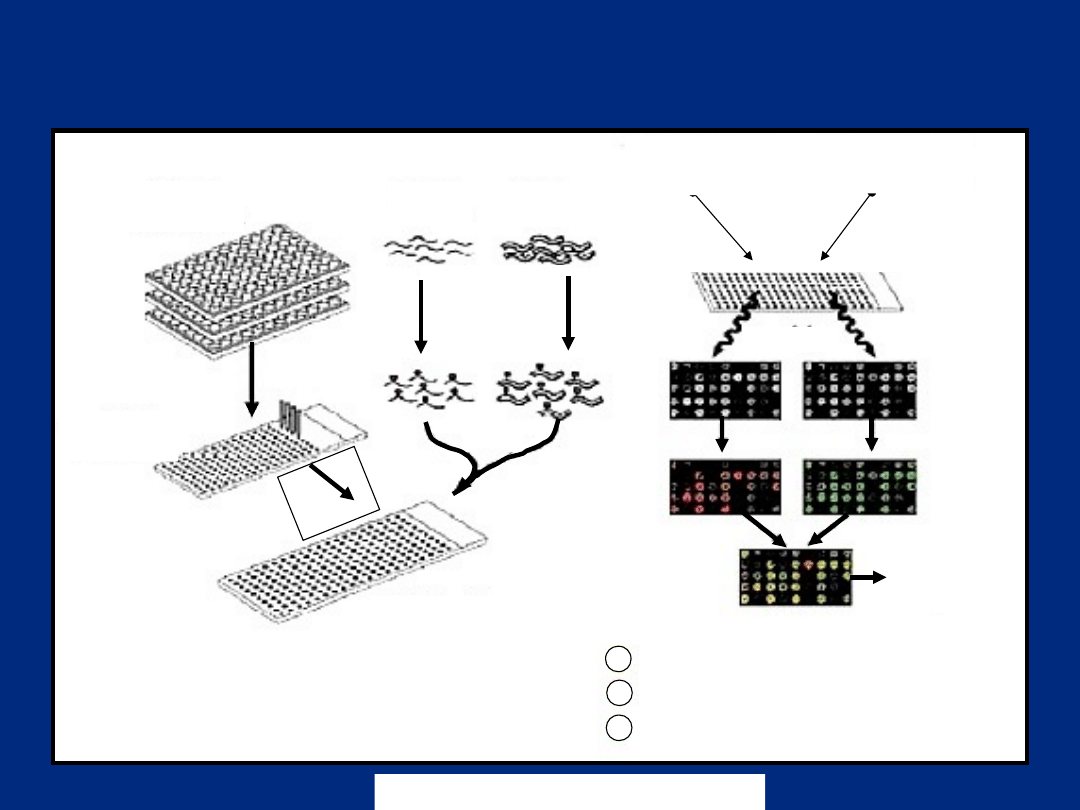
Schemat badania
Schemat badania
z wykorzystaniem mikromacierzy
z wykorzystaniem mikromacierzy
biblioteka
klonów DNA
znalezienie
„plamek”
próbka
testowa
próbka
odniesienia
odwrotna
transkrypcja
znakowanie
barwnikiem
hybrydyzacj
a
pobudzenie
pobudzenie
laser 1
laser 2
emisja
analiza
komputerowa
poziom ekspresji niezmieniony
poziom ekspresji podwyższony
poziom ekspresji obniżony
Cy5: ~650
nm
Cy3: ~650
nm
Znakowanie fluorochromem i
Znakowanie fluorochromem i
hybrydyzacja
hybrydyzacja
po izolacji mRNA podlega odwrotnej
po izolacji mRNA podlega odwrotnej
transkrypcji, w czasie której jest
transkrypcji, w czasie której jest
znakowane fluorochromem
znakowane fluorochromem
znakowana nić cDNA jest
znakowana nić cDNA jest
hybrydyzowana do DNA
hybrydyzowana do DNA
znajdującego się na mikromacierzy
znajdującego się na mikromacierzy
detekcja sygnału fluoroscencyjnego
detekcja sygnału fluoroscencyjnego
umożliwia ocenę obecności danego
umożliwia ocenę obecności danego
transkryptu w pierwotnej próbce
transkryptu w pierwotnej próbce
Źródło:
Źródło:
Wykład B. Jarząb;
Wykład B. Jarząb;
h
h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
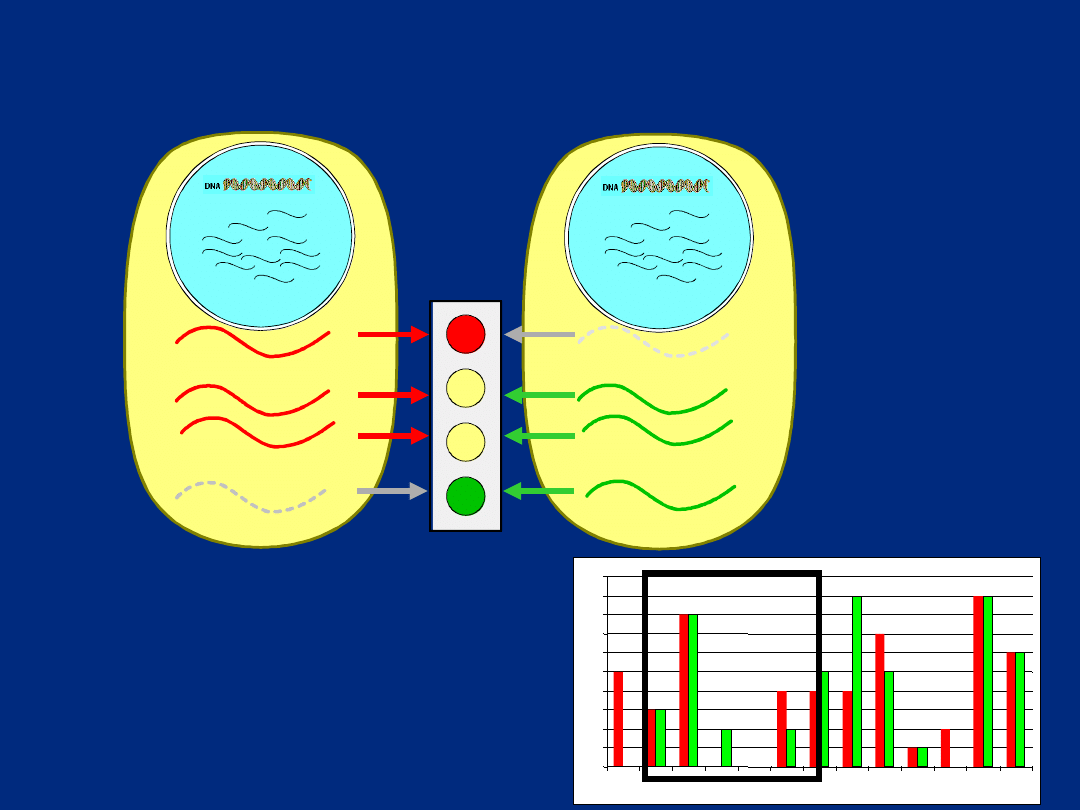
Macierze cDNA:
Macierze cDNA:
Porównawcza analiza ekspresji
Porównawcza analiza ekspresji
genów
genów
0
1
2
3
4
5
6
7
8
9
10
1
2
3
4
5
6
7
8
9
10
11
12
13
Zdrowa komórka
Nowotwór
cDNA z obydwu porównywanych
cDNA z obydwu porównywanych
typów tkanek
typów tkanek
z
z
nakujemy dwoma
nakujemy dwoma
różnymi fluorochromami
różnymi fluorochromami
i hybry
i hybry
-
-
dyzujemy wspólnie na jednej
dyzujemy wspólnie na jednej
sondzie
sondzie
Cy5
Cy3
Źródło: Wykład B. Jarząb;
http://www.genomika.pl/prezentacje/technologia_pliki/f
rame.htm

macierze
macierze
oligonukleotydowe
oligonukleotydowe
macierze cDNA
macierze cDNA
20-60 zasad/sekwencję
20-60 zasad/sekwencję
>100 zasad/sekwencję
>100 zasad/sekwencję
syntetyzowane na
syntetyzowane na
podłożu
podłożu
nakładane na podłoże
nakładane na podłoże
wiele sekwencji/gen
wiele sekwencji/gen
1 sekwencja/gen
1 sekwencja/gen
pojedyncze
pojedyncze
znakowanie
znakowanie
podwójne znakowanie
podwójne znakowanie
pomiar bezwzględny
pomiar bezwzględny
analiza porównawcza
analiza porównawcza
Mikromacierze DNA: oligo czy cDNA?
Mikromacierze DNA: oligo czy cDNA?
Źródło:
Źródło:
Wykład B. Jarząb;
Wykład B. Jarząb;
h
h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm

Technologia bioczujników DNA
Technologia bioczujników DNA
GeneChip
GeneChip
®
®
bioczujnik
bioczujnik
stacja
stacja
hybrydyzacji
hybrydyzacji
skaner
skaner
oprogramowanie
oprogramowanie
do analizy danych
do analizy danych
oprogramowanie
oprogramowanie
do analizy danych
do analizy danych
Źródło:
Źródło:
Wykład B. Jarząb;
Wykład B. Jarząb;
h
h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
Źródło:
http://microarray.republika.pl/index.htm
l
Schemat badania
Schemat badania
z wykorzystaniem mikromacierzy
z wykorzystaniem mikromacierzy
biblioteka
klonów DNA
znalezienie
„plamek”
próbka
testowa
próbka
odniesienia
odwrotna
transkrypcja
znakowanie
barwnikiem
hybrydyzacj
a
pobudzenie
pobudzenie
laser 1
laser 2
emisja
analiza
komputerowa
poziom ekspresji niezmieniony
poziom ekspresji podwyższony
poziom ekspresji obniżony
Cy5: ~650
nm
Cy3: ~650
nm
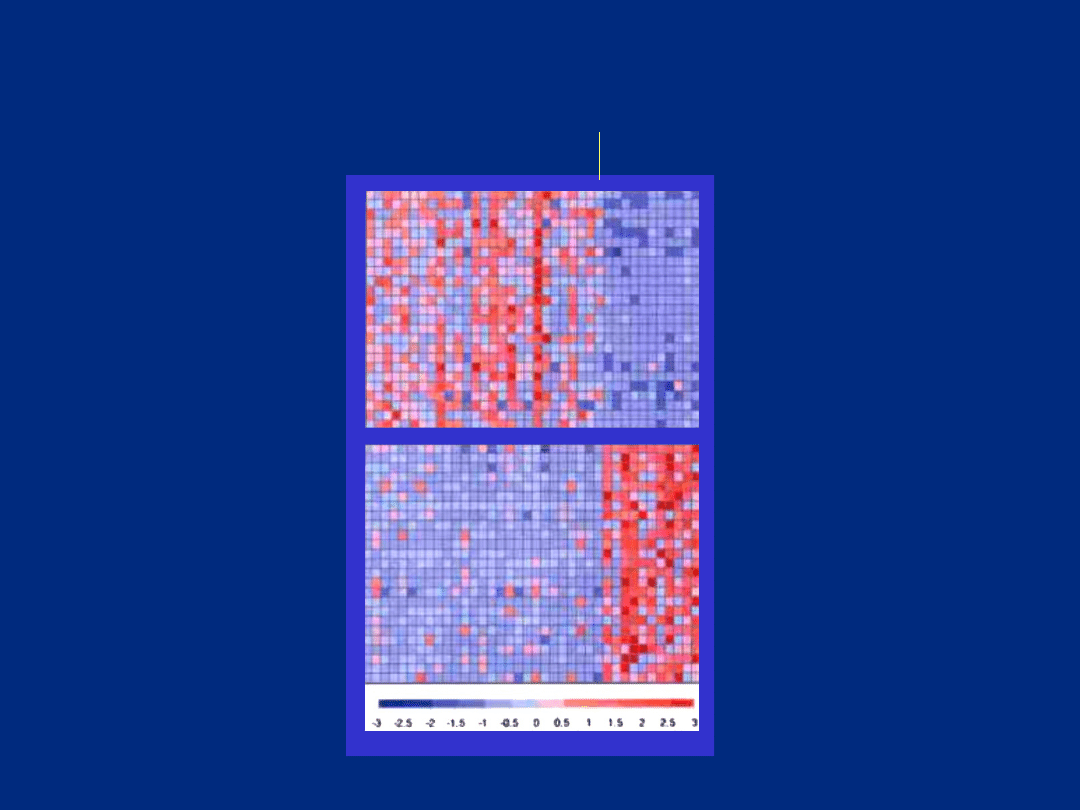
niska
wysoka
Różnice w ekspresji wybranej grupy genów między ostrą
Różnice w ekspresji wybranej grupy genów między ostrą
białaczką szpikową (AML), a ostrą białaczką limfatyczną (ALL)
białaczką szpikową (AML), a ostrą białaczką limfatyczną (ALL)
ALL
ALL
AML
ekspresja
Źródło: Wykład B. Jarząb;
http://www.genomika.pl/prezentacje/technologia_pliki/frame.htm

Źródło: www.hmds.org.uk/fish_cgh.html
Porównawcza hybrydyzacja genomowa
Porównawcza hybrydyzacja genomowa
Comparative genome hybridization (CGH)
Comparative genome hybridization (CGH)
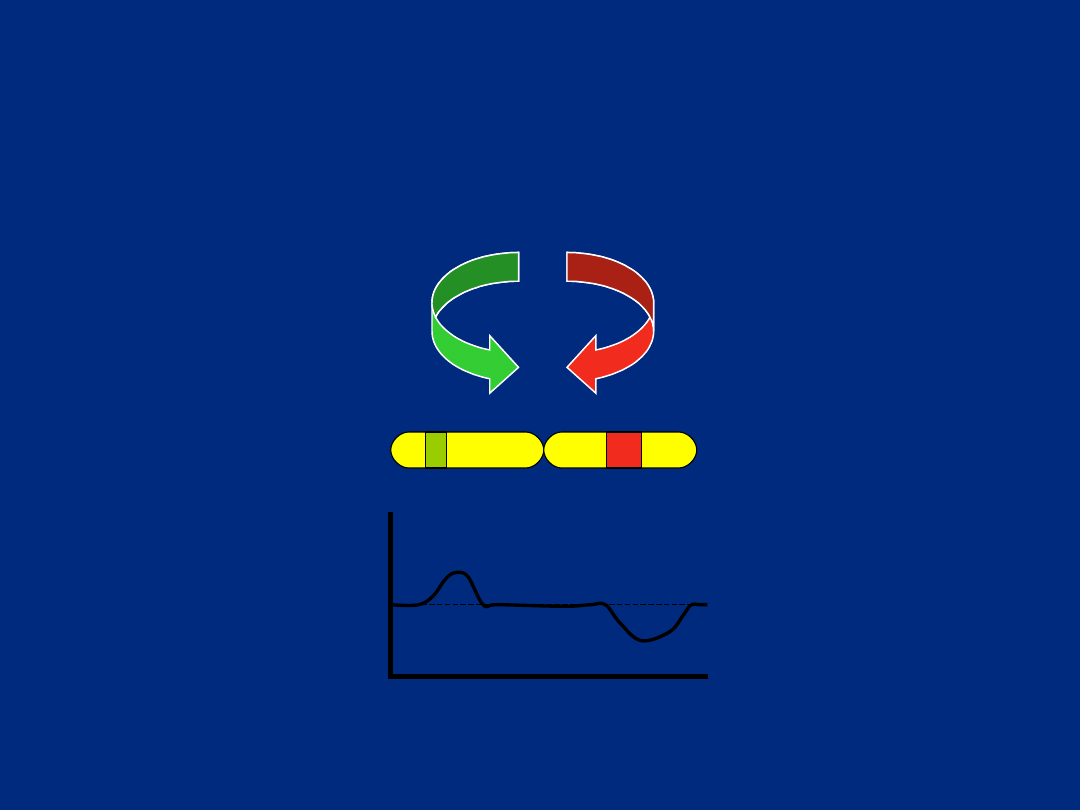
R
a
ti
o
Pozycja na chromosomie
Genomowe DNA
pacjenta
Genomowe DNA
referencyjne
Cot-1 DNA
addycja
delecja
Porównawcza hybrydyzacja genomowa
Porównawcza hybrydyzacja genomowa
Comparative genome hybridization (CGH)
Comparative genome hybridization (CGH)
Źródło: B. Nowakowska; Z-d Genetyki IMiDz w Warszawie

1.
Czułość metody HR-CGH
Czułość metody HR-CGH
~ 3-
~ 3-
5
5
Mb
Mb
3. Rozdzielczość metody zależy od wielkości
3. Rozdzielczość metody zależy od wielkości
użytych klonów oraz odległości miedzy nimi w
użytych klonów oraz odległości miedzy nimi w
genomie –
genomie –
rozdzielczość
rozdzielczość
1M
1M
pz
pz
wymaga użycia
wymaga użycia
3,500
3,500
klonów
klonów
2. Zwiększenie rozdzielczości poprzez zastąpienie
2. Zwiększenie rozdzielczości poprzez zastąpienie
chromosomów metafazowych klonami
chromosomów metafazowych klonami
BAC
BAC
oraz
oraz
PAC
PAC
CGH do mikromacierzy
CGH do mikromacierzy

CGH do mikromacierzy
CGH do mikromacierzy
Genomowe
DNA pacjenta
Genomowe
DNA referencyjne
R
a
ti
o
Pozycja klonu
Cot-1 DNA

Rodzaje i zastosowanie CGH do
Rodzaje i zastosowanie CGH do
mikromacierzy
mikromacierzy
•
Mikromacierz skonstruowana dla określonego
Mikromacierz skonstruowana dla określonego
regionu chromosomu
regionu chromosomu
- region 1p36 - Yu W i wsp.,2003,
- region 1p36 - Yu W i wsp.,2003,
- region 15q – Locke i wsp.,2004,
- region 15q – Locke i wsp.,2004,
- region 18q – Veltman JA i wsp., 2004,
- region 18q – Veltman JA i wsp., 2004,
- region 17p – Shaw CJ i wsp.,2004
- region 17p – Shaw CJ i wsp.,2004
•
Mikromacierz kliniczna, zawiera klony bakte-ryjne
Mikromacierz kliniczna, zawiera klony bakte-ryjne
specyficzne dla regionów krytycznych zespołów
specyficzne dla regionów krytycznych zespołów
genetycznych oraz regiony subtelo-merowe
genetycznych oraz regiony subtelo-merowe
(Cheung SW i wsp.,2005, Shaffer L i wsp., 1999)
(Cheung SW i wsp.,2005, Shaffer L i wsp., 1999)
•
Mikromacierz sporządzona dla całego
Mikromacierz sporządzona dla całego
genomu
genomu
(Li Ji wsp.,2003, Cai WW i wsp.,2002 )
(Li Ji wsp.,2003, Cai WW i wsp.,2002 )

•
genotypowanie – np. porównawcza hybrydyzacja (matrix CGH),
genotypowanie – np. porównawcza hybrydyzacja (matrix CGH),
detekcja mutacji, ocena polimorfizmów
detekcja mutacji, ocena polimorfizmów
•
mapowanie
mapowanie
•
badanie profilu ekspresji genów („transcriptomics”) – możliwa ocena
badanie profilu ekspresji genów („transcriptomics”) – możliwa ocena
ilościowa!
ilościowa!
–
różnice w ekspresji genów pomiędzy tkankami
różnice w ekspresji genów pomiędzy tkankami
–
profil ekspresji genów podczas rozwoju
profil ekspresji genów podczas rozwoju
–
ekspresja genów w zdrowiu i chorobie (np. porównanie nowotworu i
ekspresja genów w zdrowiu i chorobie (np. porównanie nowotworu i
tkanki zdrowej)
tkanki zdrowej)
–
wpływ leków na procesy zachodzące w komórkach
wpływ leków na procesy zachodzące w komórkach
•
badanie profilu ekspresji przez analizę białek komórkowych
badanie profilu ekspresji przez analizę białek komórkowych
(proteomika)
(proteomika)
Zastosowania mikromacierzy: początek
Zastosowania mikromacierzy: początek
nowej ery w biologii molekularnej?
nowej ery w biologii molekularnej?
Źródło:
Źródło:
Wykład B. Jarząb; h
Wykład B. Jarząb; h
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
ttp://www.genomika.pl/prezentacje/technologia_pliki/frame.htm
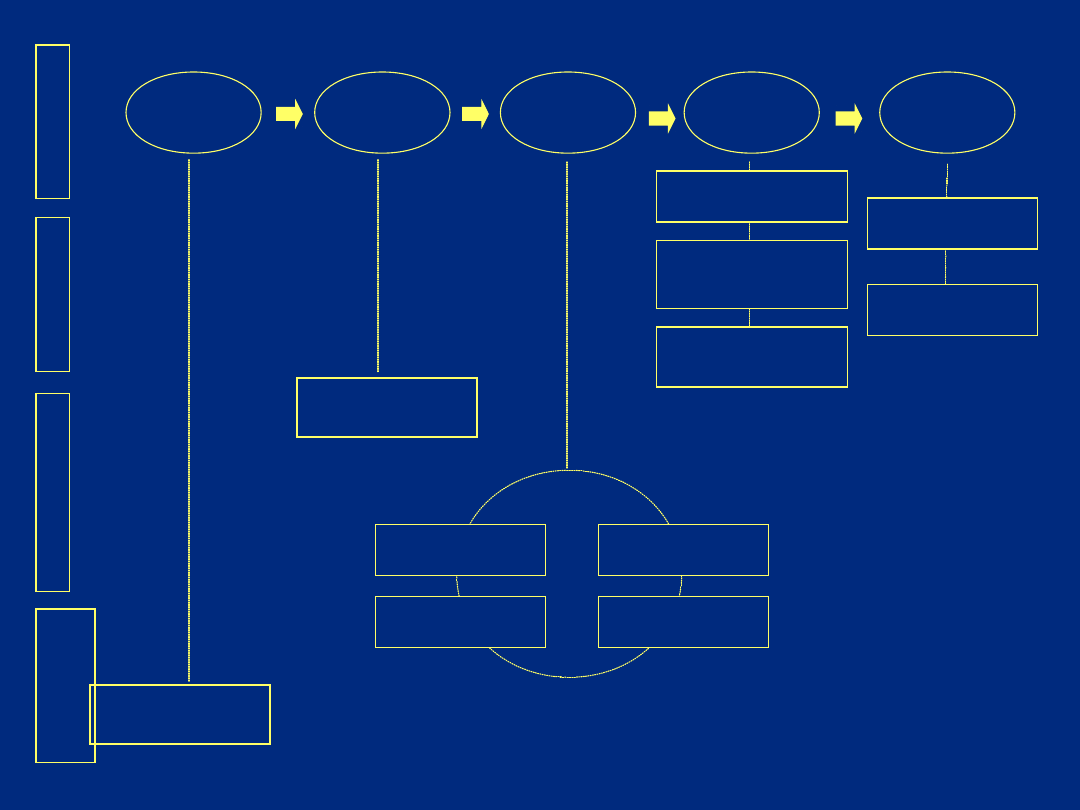
genomow
e
DNA
mRNA
produkty
białkowe
białka
funkcjonal
ne
system
biologicz
ny
sekwencjonow
anie
profil
ekspresji
badania
struktury
profil
ilościowy
białek
mapa
sprzężeń
białek
(katalog)
mapa
sprzężeń
białek
(dynamiczna)
profil
aktywności
modyfikacje
post-
translacyjne;
zmiany
aktywności
lokalizacja
subkomórkowa
integracja
danych
symulacja
systemu
te
c
h
n
o
lo
g
ia
ro
zw
ó
j
p
ro
to
ty
p
za
a
w
a
n
so
w
a
n
ie

W znaczeniu absolutnym
proteom jest nieosiągalny
podobnie jak horyzont
Jest zjawiskiem
dynamicznym, a nie
statycznym

„
Proteins are central to our
understanding of cellular function
and disease processes, and
without a concerted effort in
proteomics, the fruits of
genomics
will go unrealized”
Ian Humphery-Smith
University of Utrecht
współzałożyciel HUPO
(2001)

Modyfikacje potranslacyjne
fosforylacj
a
glikozylacj
a
sulfatacja
metylacja
acetylacja
aktywność
stabilność
lokalizacja
przemiana
(metaboliz
m)
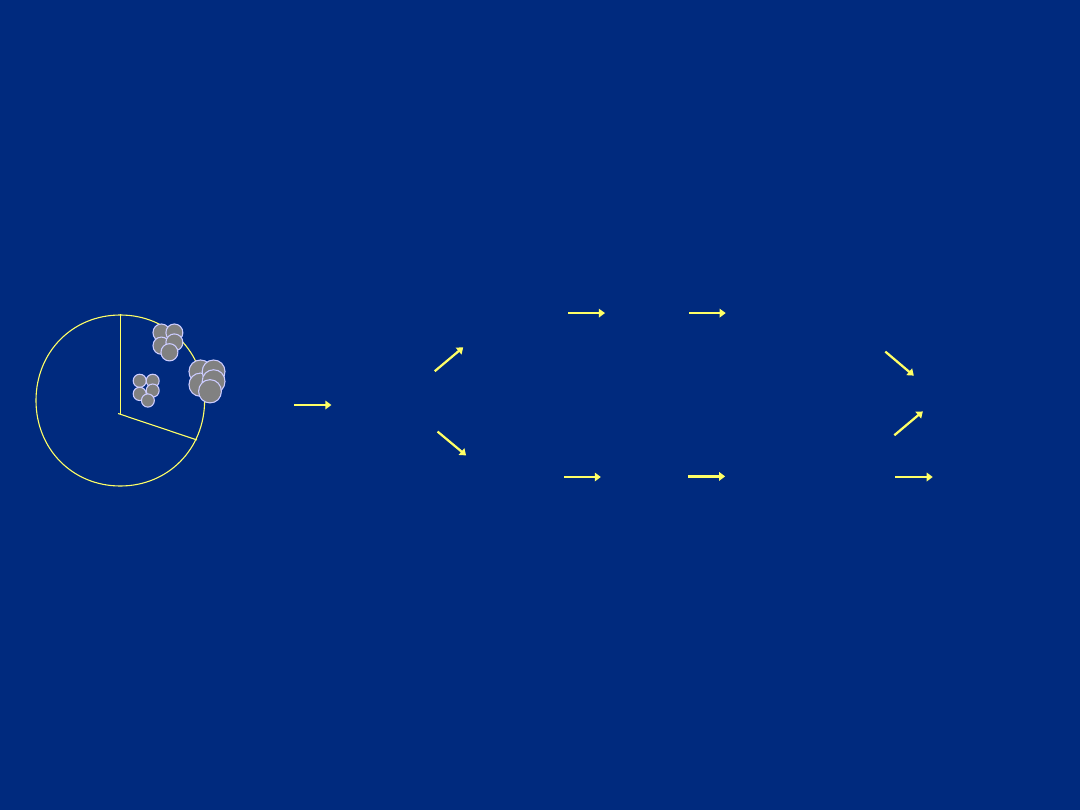
ekstra
kt
wzbogacen
ie
analiza
ilościowa (2DE)
trawienie
trawienie
rozdział
spektrometryczn
a analiza mas
cząst.
spektrometryczn
a analiza mas
cząst.
identyfikac
ja
analiza
ilościowa

Zadania HUPO
Działania na rzecz powszechnego
dostępu drogą elektroniczną do
wyników badań
Odstąpienie od zasady patentowania
wyników prac badawczych zarówno w
ośrodkach akademickich jak i
przemysłowych
Właściwa koordynacja badań w
zakresie merytorycznym i finansowym

Zadania HUPO
Opracowanie definicji proteomu w plazmie
Występowanie z propozycjami zakresu badań w
określonych typach komórek
Działania na rzecz utworzenia konsorcjum
mającego na celu wytworzenie przeciwciał dla
wszystkich białek człowieka
Skatalogowanie pierwotnej struktury
wszystkich białek
Mapowanie wszystkich organelli i uzyskanie ich
w postaci oczyszczonej (sub-proteomika)

Zadania HUPO
Opracowanie map interakcji między białkami w
modelowych organizmach
Integracja badań w zakresie proteomiki i
genomiki
Rozwój metod informatycznych i budowa
odpowiedniej infrastruktury
Information must obviously be presented in a
form that can be processed by the human user
(Nature, March 2003)

Efekty HUPO
Identyfikacja kompleksu-1 w stwardnieniu guzowatym
jako fizjologicznie występującego substratu białkowej kinazy B
(PKB)
dzięki integracji danych z wykorzystaniem algorytmu ScanSite
tylko na podstawie metod elektroforetycznych z uwzględnieniem
ilości fragmentów sekwencji specyficznych dla PKB
Badania nad Neurokininą B (NKB)
w stanach przedrzucawkowych wzrost poziomu NKB
W badaniach cytotrofoblastu wzrost transkrypcji RZR alfa,
obniżona transkrypcja c-abl, CD40 oraz lekkiego łańcucha dyneiny
cytoplazmatycznej (genomika)
Obniżony poziom 21 białek NKB zależnych, związanych klinicznie
ze stanem przedrzucawkowym (m. in. thoredoxin – udział w
obronie przed stresem oksydacyjnym; annexin II –
wewnątrznaczyniowe procesy trombolityczne; białko wiążące
fosfanetanolaminę/inhibitor kinazy Raf – odpowiedź zapalna)
(proteomika)

Efekty HUPO
Badania nad proteomem powierzchni komórki
Wykrycie szeregu nowych białek na powierzchni komórek
nowotworowych
Badania „in situ” w tkankach nowotworowych
z użyciem macierzy indukowanej laserowo
(matrix-assisted laser desorption/ionization; MALDI)
Badania z użyciem przeciwciał dla określonych typów białek –
choroby autoimmunologiczne, w tym np. lupus erythematosus

Proteomika – badania
proteomu
• Skład wszystkich białek w komórce
• Skład wszystkich izoform i zmodyfikowanych
postaci białek
• Interakcje między białkami
• Opis struktury białek
• Tworzenie przez białka złożonych struktur i
kompleksów
• Pełny opis funkcjonowania komórki

Proteomika – badania
proteomu
Two dimensional polyacrylamide gel
electrophoresis – 2D PAGE
np. profile białkowe w różnicowaniu podtypów
białaczek
Elektroforeza w gradiencie pH (immobilized pH
gradient – IPG)
stabilizacja gradientu pH w podłożu
acrylamidowym
Spektrometria w oparciu o masę molekularną
białek
i ich fragmentów (mass spectrometry)

Proteomika – badania
proteomu
Badania układów białek z użyciem białek
rekombinowanych lub czynników (znaczników)
wchodzących w reakcje z białkami – przeciwciała,
peptydy, mikrocząsteczki
Lokalizacja białek metodami fluorescencyjnymi
GFP – green fluorescent protein
Fluorescencyjny rezonans przenoszenia energii
FRET – fluorescence resonance energy transfer
FlexGene Consortium - kolekcja cDNA
niezbędna dla celów identyfikacji białek;
http://www.hip.harvard.edu

Proteomika – badania
proteomu
Badania białek nieaktywnych, kompleksów
białek lub megakompleksów na skalę komórki
Krystalografia z użyciem promieni X
Magnetyczny rezonans jądrowy (NMR)
Mikroskopia elektronowa
Tomografia elektronowa
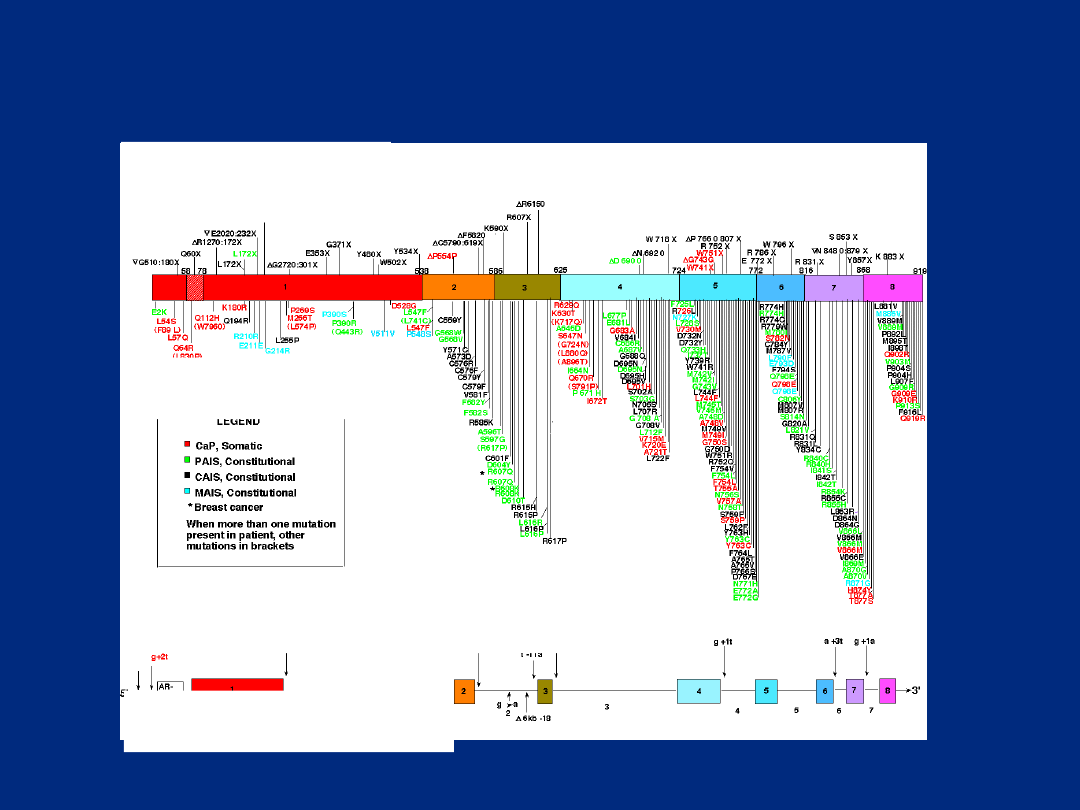
Źródło: http://ww2.mcgill.ca/androgendb
Źródło: http://ww2.mcgill.ca/androgendb
Premature
termination
mutations, or 1-4 bp
Aa substitution
mutations
Location of splicing and untranslated
mutations
Location of intron
mutations
ZESPÓŁ NIEWRAŻLIWOŚCI NA ANDROGENY
ZESPÓŁ NIEWRAŻLIWOŚCI NA ANDROGENY
Mutacje genu receptora androgenowego
Mutacje genu receptora androgenowego

locus
symbol genu
Gen kandydujący
cM
AR
D15S519
D15S520
D10S192
CYP19
D17S934
HSD17B2
D9S1809
D1S514
D8S1821
AR
CYP11A
CYP11A
CYP17
CYP19
HSD17B1
HSD17B2
HSD17B3
HSD3B1+2
STAR
Androgen receptor
CYP11A-cytochrome P450 side-chain cleavage enzyme
CYP11A-cytochrome P450 side-chain cleavage enzyme
CYP17-cytochrome P450 17-hydroxylase/17,20-desmolase
CYP19-cytochromearomatase
17-hydroxysteroid dehydrogenase, type I
17-hydroxysteroid dehydrogenase, type II
17-hydroxysteroid dehydrogenase, type III
3-hydroxysteroid dehydrogenase, type I and II
Steroidogenic acute regulatory protein
0
0
0
< 1
0
<2
0
<1
<1
<2
Geny „kandydujące” w etiopatogenezie PCOS
Geny „kandydujące” w etiopatogenezie PCOS
wg : Urbanek i wsp., PNAS, 96, 8573, 1999
wg : Urbanek i wsp., PNAS, 96, 8573, 1999
Lokalizacja
chromosomowa
w zakresie działania
hormonów steroidowych:
Xq11.2
15q23-24
15q23-24
10q24.3
15q21
17q11-21
16q24.2
9q22
1p31.1
8p11.2

locus
symbol genu
Gen kandydujący
cM
D12S347
D2S2335
D3S1298
D5S474
D5S623
D5S822
D2S163
INHBA
D12S347
D2S2335
D3S1298
D5S474
D5S623
D5S822
ACTR1
ACTR2A
ACTR2B
FS
FS
FS
INHA
INHBA
INHBB
INHC
SHBG
LHCGR
FSHR
MADH4
Activin receptor 1
Activin receptor 2A
Activin receptor 2B
Follistatin
Follistatin
Follistatin
Inhibin A
Inhibin - A
Inhibin - B
Inhibin C
Sex hormone binding globulin
Luteinizing hormone/choriogonadotropin receptor
Follicle-stimulating hormone receptor
Mothers against decapentaplegic homolog 4
< 1
< 1
< 1
< 2
< 0,5
< 1
< 1
0
2
< 1
< 1
< 2
< 2
< 1
Lokalizacja
chromosomowa
w zakresie działania gonadotropin:
12q13.12
2q22.2
3p22.2
5p14
5p14
5p14
2q33.34
7p13-15
2cen-2q13
12q13
17p13.2
2p21
2p21
18q21
Geny „kandydujące” w etiopatogenezie PCOS
Geny „kandydujące” w etiopatogenezie PCOS
wg : Urbanek i wsp., PNAS, 96, 8573, 1999
wg : Urbanek i wsp., PNAS, 96, 8573, 1999

locus
symbol genu
Gen kandydujący
cM
IGF1
IGF1R
D7S519
HphI site
INSR
D19S216
D19S905
D19S884
D19S922
D19S391
D19S865
D19S906
D19S840
D19S212
D19S410
IRS1
D3S1263
IGF1
IGF1R
IGFBPI1+3
INS VNTR
INSR
INSR
INSR
INSR
INSR
INSR
INSR
INSR
INSR
INSL3
INSL3
IRS1
PPARG
Insulin-like growth factor I
Insulin-like growth factor I receptor
Insulin-like growth factor binding protein 1 + 3
Insulin gene VNTR
Insulin receptor
Insulin receptor
Insulin receptor
Insulin receptor
Insulin receptor
Insulin receptor
Insulin receptor
Insulin receptor
Insulin receptor
Leydig insulin-like protein 3
Leydig insulin-like protein 3
Insulin receptor substrate 1
Peroxisome proliferator-activated receptor-gamma
0
0
1
0
0
4.2
0
1.2
1.2
3.6
7.2
11
14
< 1
< 1
0
< 0.2
Lokalizacja
chromosomowa
w zakresie działania insuliny:
12q22-23
15q25-26
7p13-7p12
11p15.5
19p13.3
19p13.3
19p13.3
19p13.3
19p13.3
19p13.3
19p13.3
19p13.3
19p13.3
19p13.1
19p13.1
2q36-37
3p25-24.2
Geny „kandydujące” w etiopatogenezie PCOS
Geny „kandydujące” w etiopatogenezie PCOS
wg : Urbanek i wsp., PNAS, 96, 8573, 1999
wg : Urbanek i wsp., PNAS, 96, 8573, 1999

locus
symbol
genu
Gen kandydujący
cM
D18S564
D7S1875
D1S198
D2S131
D11S911
MC4R
OB
OBR
POMC
UCP2 + 3
Melanocortin 4 receptor
Leptin
Leptin receptor
Pro-opiomelanocortin
Uncoupling protein 2 + 3
< 3
0.2
0.5
< 1
< 4
Lokalizacja
chromosomow
a
otyłość i regulacja energetyczna:
12q13.12
2q22.2
3p22.2
5p14
5p14
Geny „kandydujące” w etiopatogenezie PCOS
Geny „kandydujące” w etiopatogenezie PCOS
wg : Urbanek i wsp., PNAS, 96, 8573, 1999
wg : Urbanek i wsp., PNAS, 96, 8573, 1999

Stosowane techniki badań polimorfizmów DNA
Stosowane techniki badań polimorfizmów DNA
VNTR minisatellite - variable number tandem repeats
VNTR minisatellite - variable number tandem repeats
SSLP (STRP) - single sequence length polymorphism (short
SSLP (STRP) - single sequence length polymorphism (short
tandem repeat polymorphism)
tandem repeat polymorphism)
RFLP - restriction fragment length polymorphism
RFLP - restriction fragment length polymorphism

Geny kandydujące („kandydaci”)
Geny kandydujące („kandydaci”)
analizowane w PCOS
analizowane w PCOS
gen
gen
lokalizacja
lokalizacja
marker
marker
wartość P
wartość P
follistatin
follistatin
5p14
5p14
D5S474
D5S474
D5S623
D5S623
D5S822
D5S822
0.8871
0.8871
0.2437
0.2437
0.1215
0.1215
aromataz
aromataz
a
a
15q21
15q21
CYP19
CYP19
0.785
0.785
CYP17a
CYP17a
10q24.
10q24.
3
3
D10S192
D10S192
0.7663
0.7663
receptor
receptor
insulinowy
insulinowy
19p13.3
19p13.3
D19S884
D19S884
D19S922
D19S922
0.006
0.006
*
*
0.2848
0.2848
wg Tucci i wsp.: J. Clin. Endocrinol. Metab., 86, 446, 2001
wg Tucci i wsp.: J. Clin. Endocrinol. Metab., 86, 446, 2001

Markery z najbliższego otoczenia genu dla INSR
Markery z najbliższego otoczenia genu dla INSR
w badaniach u pacjentek z PCOS
w badaniach u pacjentek z PCOS
lokalizacja (cM)
lokalizacja (cM)
marker
marker
wartość P
wartość P
20.0
20.0
23.0
23.0
25.2
25.2
25.2
25.2
25.2
25.2
25.2
25.2
25.2
25.2
26.4
26.4
27.1
27.1
27.2
27.2
0.891
0.891
0.092
0.092
0.150
0.150
0.116
0.116
0.785
0.785
0.475
0.475
0.750
0.750
0.006
0.006
0.561
0.561
0.178
0.178
D19S216
D19S216
D19S869
D19S869
INSR
INSR
D19S406
D19S406
D19S567
D19S567
D19S873
D19S873
D19S905
D19S905
D19S884
D19S884
D19S912
D19S912
D19S922
D19S922
wg Tucci i wsp.: J. Clin. Endocrinol. Metab., 86, 446, 2001
wg Tucci i wsp.: J. Clin. Endocrinol. Metab., 86, 446, 2001

Markery z najbliższego otoczenia genu dla INSR
Markery z najbliższego otoczenia genu dla INSR
w badaniach u pacjentek z PCOS
w badaniach u pacjentek z PCOS
pter
pter
cen
cen
region 19p13.3
region 19p13.3
INSR
INSR
D19S1186
D19S1186
D19S905
D19S905
Resistin
Resistin
D19S1190
D19S1190
D19S1183
D19S1183
D19S912
D19S912
D19S922
D19S922
D19S884
D19S884
<
<
<
<

Wykład:
Wieloczynnikowe uwarunkowania
chorób nowotworowych
Patofizjologia
III rok Wydziału Lekarskiego
Rok akademicki 2005/2006
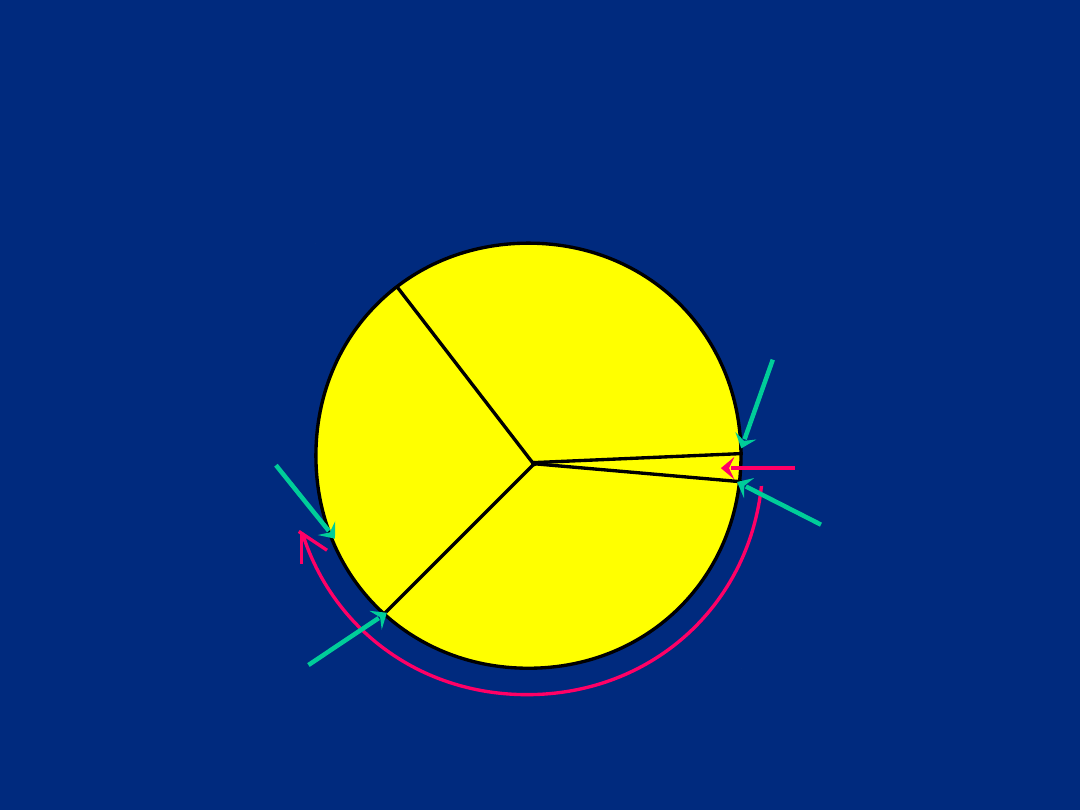
G
1
G
2
S
M (mitoza);
MPF
punkt kontrolny G1/S
białka P53 (TP53);
PRB (P110) – gen RB1
zatrzymanie G1; reparacja
DNA
punkt kontrolny
G2/M
białka P53 (TP53);
PRB (P110) – gen
RB1
kontrola
kondensacji
i segregacji
materiału
genetycznego
Cykl komórkowy
fosforylacja
białek
fosforylacja
białek

Cykl komórkowy
P53 (17p13.1) - „strażnik genomu”
Wzrost poziomu w miarę starzenia się komórki –
zatrzymanie cyklu komórkowego
Zniesienie tego efektu przez onkogenne białka
wirusowe:
antygen T wirusa SV40
białko E1A adenowirusa
białka E6, E7 brodawczaka ludzkiego
Biologicznie czynny w postaci ufosforylowanego
homotetrameru;
Zachowuje się jak czynnik transkrypcyjny
W formie zmutowanej lub z ww. v-onc nieaktywne
heterotetramery
Regulatorem P53 jest MDM2 (12q13-14)

Cykl komórkowy
MPF - M-phase Promoting Factor
Cyklina B składnikiem kluczowym
– Wysoki poziom MPF prowadzi do:
kondensacji chromosomów (rola histonu
H1)
uszkodzenia otoczki jądrowej
tworzenia (montowania) wrzeciona
– Niski poziom MPF prowadzi do:
segregacji chromosomów
dekondensacji chromosomów
odtwarzania otoczki jądrowej
replikacji DNA
podwojenia centromeru

Cykl komórkowy
Geny mutatorowe
odpowiedzialne za naprawę zmian w
całym genomie
poreplikacyjny system naprawy
błędnie sparowanych zasad
MSH2, MLH1,
PMS1, PMS2

Online Mendelian Inheritance In Man
(OMIM)
www.ncbi.nlm.nih.gov/Omim

Cykl komórkowy
fosforylacja białek fazy S → synteza DNA
kinazy zależne od cyklu komórkowego (CDK)
cykliny (białka regulatorowe)
aktywacja (fosforylacja) kompleksu cyklina/CDK
przez kinazę CAK (cdk activating kinase; CDK7)
lub przez kinazy białkowe typu wee-1/mik-1
poprzedzająca ten proces aktywacja CAK przez cyklinę H
enzymy INK uniemożliwiające fosforylację kompleksu cyklina/CDK
enzymy INK uniemożliwiające fosforylację kompleksu cyklina/CDK
lub prowadzące do dysocjacji kompleksu
lub prowadzące do dysocjacji kompleksu

Cykl komórkowy
Cykl komórkowy
Apoptoza
– w przebiegu prawidłowego rozwoju
– w prawidłowym obrocie komórki
– w atrofii indukowanej cytokinami np. przez
czynnik martwicy nowotworów (TNF – tumor
necrosis factor)
– w wirusowej indukcji procesu np. w AIDS
– w niektórych chorobach
neurodegeneracyjnych
jony wapniowe indukują apoptozę
(endonukleazy, tkankowa transglutaminaza)
jony cynku są inhibitorem apoptozy

Cykl komórkowy
Apoptoza
– kondensacja chromatyny
– rozpad jądra
– tworzenie pęcherzykowatych uwypukleń błony
jądrowej
– fragmentacja komórki na drobne ciałaka
apoptotyczne
utrata kwasów sjalowych – końcowych cukrów
w glikoproteinach i w glikolipidach błony
komórki apoptotycznej – komórki stają się
„smaczniejsze” dla makrofagów
receptory witronektynowe na powierzchni
komórki przyciągają makrofagi

Nowotwór
Nowotwór
to:
to:
•
niekontrolowany
rozplem
(namnażanie,
proliferacja)
populacji
komórek
przekraczający potrzeby organizmu
lub
•
zaburzenia równowagi między tempem
podziałów komórkowych, a szybkością
utraty komórek
albo
• zaburzenia procesów kontroli wzrostu,
różnicowania, dojrzewania, lokalizacji i
śmierci komórek organizmu
– cechy transformacji nowotworowej

Rak zaczyna powstawać wówczas, gdy
komórka wyłamuje się spod kontroli
mechanizmów decydujących o jej
podziałach i lokalizacji
Robert A. Weinberg

Genom
30000 genów
20000 (?) genów
aktywnych w danym typie komórek
większość to geny metabolizmu podstawowego =
houssekeeping gene
replikacja i reparacja DNA; budowa organelli komórkowych i innych
struktur komórkowych i tkankowych
> 1000 genów
uczestnictwo w torach mutacyjnych
kontrola wzrostu, różnicowania, śmierci komórki;
replikacja i reparacja DNA; transmisja sygnałów;
aktywacja innych genów

Genotyp a nowotwór
Protoonkogeny (c-onkogeny)
- prawidłowe elementy genomu człowieka biorące udział w
przebiegających fizjologicznie procesach rozwojowych;
przykłady:
c-src – cytoplazmatyczna kinaza białkowa (fosforylacja aminokwasów w
obrębie określonych białek docelowych)
c-erbB – receptor naskórkowego czynnika wzrostu; aktywność tyrozynowej
kinazy białkowej)
Int-2 – związek z genem czynnika wzrostu fibroblastów
c-jun – czynnik transkrypcyjny regulujący ekspresję genów
c-ras – produkt wiąże GTP; istotne dla kaskady sygnałów z
powierzchniowych receptorów komórki do jądra
Onkogeny
- do ujawnienia potencjału onkogennego protoonkogenu
niezbędna jest jego aktywacja
- uszkodzenie onkogenu, jego niewłaściwa lub nadmierna
ekspresja może prowadzić do transformacji nowotworowej

Genotyp a nowotwór
Onkogeny
- do ujawnienia potencjału onkogennego protoonkogenu
niezbędna jest jego aktywacja
- wybrane mechanizmy:
mutacja punktowa (przykład: mutacje c-ras)
aktywacja poprzez łączenia strukturalne (przykład: c-
abl; bcr)
amplifikacja genu; także insercje
małe unikatowe chromosomy (double minutes) lub
regiony o wielu kopiach onkogenu w zatartej strukturze
chromosomu (HSR)
poddanie protoonkogenu kontroli bardziej aktywnego
promotora (np. retrowirusowego)
poddanie protoonkogenu kontroli sekwencji
wzmacniającej (np. translokacja c-myc z chromosomu 8
na 14 w pobliżu locus immmunoglobulin w chłoniaku
Burkitta)

Genotyp a nowotwór
Onkogeny
Wybrane onkogeny i ich funkcja
Nazwa onkogenu
Funkcja produktu onkogenu
src, yes, fgr, abl, fps, kit, sea,
ros
raf, mos
Kinaza białkowa
tyrozyny
seryny/treoniny
sis
int
Czynniki wzrostu
PDGF
FGF
erb-B
fms
erb-A
mas
Receptory czynników wzrostu
EGF
M-CSF
hormonu tarczycy
angiotenzyny
Ha-ras, Ki-ras, N-ras
Białka wiążące GTP
myb, myc, fos, jun, ets, rel, ski
Czynniki transkrypcyjne
bcl-2
Inne, np. białko błony
mitochondrialnej i jądrowej
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
Genotyp a nowotwór
Geny supresorowe transformacji nowotworowej (antyonkogeny)
Gen
supresorow
y
Lokalizacja
chromosomo
wa
Funkcja produktu genu
P53
17p13.1
czynnik transkrypcyjny, kontroler
prawidłowego przebiegu proliferacji,
„strażnik genomu”
RB1
13q14
kontroler prawidłowego przebiegu
proliferacji, czynnik transkrypcyjny,
APC
5q21
? (cząsteczka adhezyjna)
WT1
11p13
? (czynnik transkrypcyjny)
NF1
17q11
cytoplazmatyczne białko aktywujące
GTP
NF2
22q
białko cytoszkieletu
VHL
3p25
? (inhibitor elongacji transkrypcji)
MTS1
9p21
inhibitor kinazy CDK4 i 6
BRCA1
17q21
czynnik transkrypcyjny,
BRCA2
13q12-13
czynnik transkrypcyjny,
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
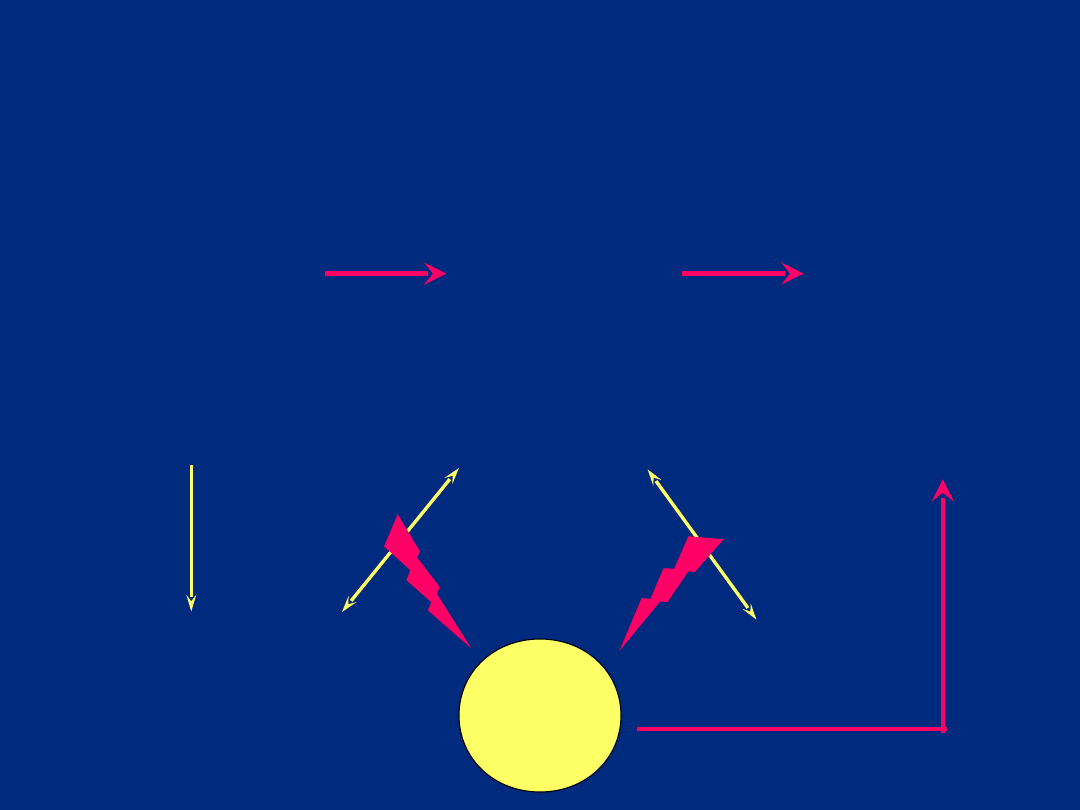
Etapy karcinogenezy
Etap I
PRE –
INICJACJA
(inicjacja)
ekspozycja na
karcinogeny
chemiczne, fizyczne i
biologiczne
Etap II
INICJACJA
(promocja)
nagromadzenie
mutacji
prowadzących do
transformacji
Etap III
PROGRESJ
A
(progresja)
selekcja klonalna;
nabycie zdolności
do
przerzutowania
carcinoma
„in situ”
naprawa
DNA
apoptoz
a

Etapy karcinogenezy
PRE – INICJACJA
–
ekspozycja na karcinogeny chemiczne, fizyczne i
biologiczne
–
predyspozycje genetyczne w metabolizowaniu i
usuwaniu czynników rakotwórczych
= czas
ekspozycji komórki na działanie czynników
uszkadzających lub ich metabolitów
–
predyspozycje genetyczne związane z reparacją
DNA (polimorfizm genów kodujących enzymy
systemu reparacyjnego)
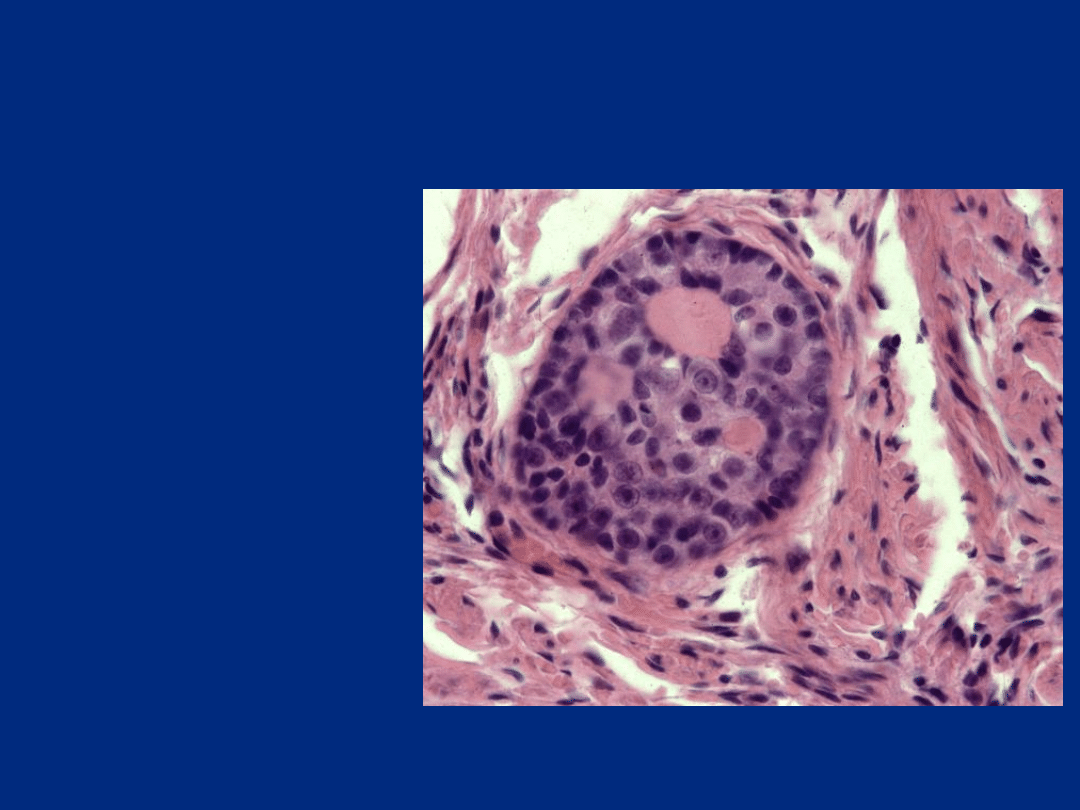
CZYSTA DYSGENEZJA GONAD
CZYSTA DYSGENEZJA GONAD
(zespół Swyera; MIM 306100)
(zespół Swyera; MIM 306100)
Gonadoblastoma „in
situ”
u kobiety 46,XY
Etapy karcinogenezy
Pre-inicjacja i inicjacja
(promocja)
Czynniki inicjujące:
(karcinogeny i
prokarcinogeny)
Czynniki wspierające:
(promotory i
kokarcinogeny)
Są samodzielnie rakotwórcze
Są mutagenami
Wiążą się kowalencyjnie z
DNA
Wystarczy pojedyncza
ekspozycja
Działanie zależne od dawki i
kumulatywne
Nie ma dawki progowej
Ekspozycja na karcinogen
musi poprzedzać działanie
promotora
Samodzielnie nie są
rakotwórcze
Nie wykazują działania
mutagennego
Nie wiążą się z DNA
Konieczna długotrwała,
powtarzana ekspozycja
Działanie odwracalne i
niekumulatywne
Istnieją dawki progowe
Ekspozycja musi być po
zadziałaniu karcinogenu
Na podstawie: Patofizjologia. Red. S. Maśliński, J. Ryżewski, PZWL 2000
Na podstawie: Patofizjologia. Red. S. Maśliński, J. Ryżewski, PZWL 2000

Etapy karcinogenezy
INICJACJA
– nagromadzenie
mutacji
prowadzących
do
transformacji
– stała ekspozycja na karcinogeny i błędy naprawy
DNA
– błędy w procesie replikacji DNA (np. gen MST1 w
dziedzicznym raku jelita grubego - brak
identyfikacji źle sparowanych zasad)
– tory mutacyjne; różne znaczenie poszczególnych
mutacji; uszkodzenia genu P53 w ponad 50%
wszystkich
typów
komórek
nowotworowych
(odziedziczenie mutacji P53 to ryzyko zespołu Li-
Fraumeni przed 40 rż)
– skrócenie czasu transformacji, gdy jedna z mutacji
toru jest odziedziczona (np. gen RB i ryzyko
siatkówczaka)
– nieefektywna
apoptoza
lub
procesy
immunologiczne

Etapy karcinogenezy
WYBRANE ZMIANY TOWARZYSZĄCE
TRANSFORMACJI NOWOTWOROWEJ
– Zmiany w błonach komórkowych
Zwiększenie transportu metabolitów
Zwiększenie tworzenia pęcherzyków błonowych
Wzrost ruchliwości białek błonowych
– Zmiany adhezyjne
Zmniejszenie adhezji powierzchniowej
Nieprawidłowa organizacja filamentów aktynowych
Zanik zewnętrznych struktur fibronektynowych
Nadprodukcja aktywatora plazminogenu
– Zmiany we wzroście i podziałach komórek
Zmniejszenie zapotrzebowania na czynniki wzrostowe
Nabycie
zdolności
do
nieograniczonej
proliferacji
(nieśmiertelność)
Nabycie zdolności do wzrostu w zawiesinie
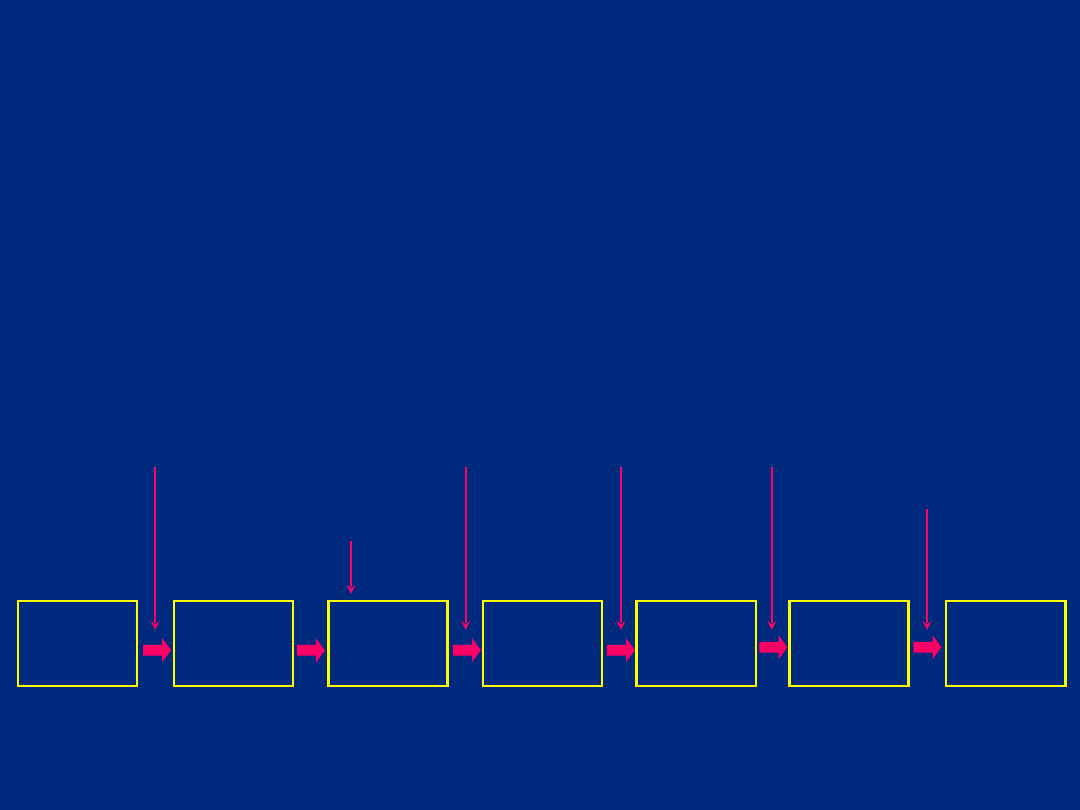
Tor mutacyjny prowadzący do raka jelita grubego
nabłone
k
hiperplaz
ja
nabłonka
wczesny
gruczola
k
pośredni
gruczola
k
późny
gruczola
k
przerzut
y raka
rak
5q
utrata
APC
12p
aktywac
ja
Ki-RAS
18q
utrata
DCC
17p
utrata
P53
Inne
zmiany
hipometylacj
a DNA
Etapy karcinogenezy

Przyjmując pogląd o
monoklonalnym pochodzeniu
nowotworów należy uznać, że
aby pojedyncza komórka mogła
ulec pełnej transformacji musi
mieć utrwalone mutacje w co
najmniej kilku, a nawet
kilkunastu genach

Guz nowotworowy stanowi jednak często
strukturę złożoną (heterogenną) z komórek
różnego
pochodzenia,
prawidłowych
i
stransformowanych (pleoclonal origin):
– zdolnych lub niezdolnych do podziału;
– proliferujących (clonogenic and stem
cells);
wznowy po leczeniu, hodowla „in vitro”,
retransplantacja
–
hybryd komórek
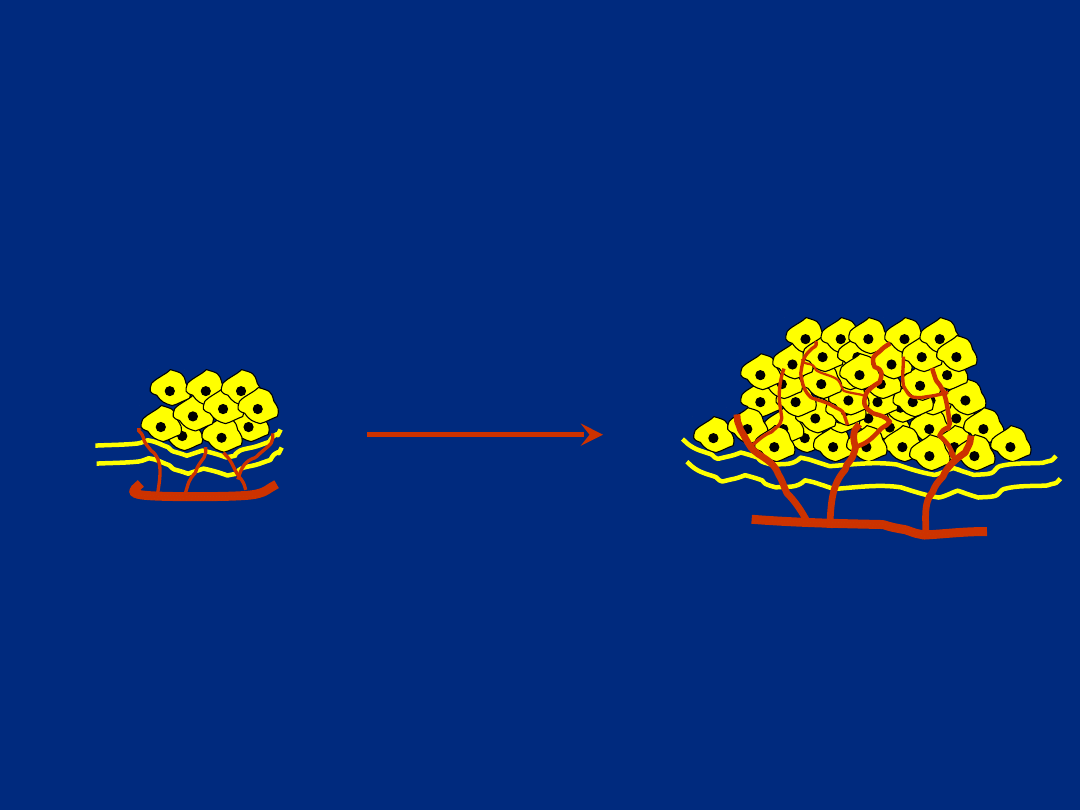
Angiogenez
a
VEGF, TAF, EAF,
cytokiny, kolagenazy,
proteazy i szereg
innych
VEGF – vascular endothelial
growth factor
TAF – tumor angiogenesis factor
EAF – epithelial angiogenesis
factor
Carcinoma
„in situ”
Rozrost
nowotworo
wy
unaczynion
y
Etapy karcinogenezy

Etapy karcinogenezy
PROGRESJA
– selekcja klonalna; nabycie zdolności do
przerzutowania
– angiogeneza,; substancje odżywcze; przewaga
namnażania nad śmiercią komórek
– pogłębiająca się niestabilność genomu; wzrost
liczby mutacji; specyficzne dla typu nowotworu i
niespecyficzne aberracje chromosomowe
– penetracja
naczyń
krwionośnych
i/lub
limfatycznych
– w klonach zyskujących przewagę aktywacja genów
ułatwiających szybki wzrost i tworzenie nacieków
inwazyjnych
– poza zaburzeniami proliferacji także zaburzenia
adhezji
z aktywacją enzymów proteolitycznych (np.
kolagenazy IV)

Etapy karcinogenezy
PRZERZUTOWANIE
– Enzymy
aktywatory plazminogenu; plazmina;
rozpuszczanie fibryny i fibrynogenu
metaloproteinazy, katepsyny, glikozydazy trawiące
proteoglikany, różne typy kolagenu (zwłaszcza
kolagenaza IV), elastynę, fibronektynę
zaburzenia krzepnięcia z aktywacją płytek;
mediatory; penetracja komórek do naczyń;
przerzuty
– Adhezyny
integryny, nadrodzina Ig, kaderyny-E, slektyny;
zaburzenia przylegania do macierzy komórkowej
(ECM-extracellular matrix) i oddziaływań
międzykomórkowych

Etapy karcinogenezy
PRZERZUTOWANIE
– Zaangażowane geny:
rodzina genów NM23; niższe poziomy białek
NM23
– większa inwazyjność; kodowanie
kinazy
NDP
– forylacja GDP do GTP
gen dla kadheryny-E (uwomoruliny) – białko
łączące fragmenty aktynowe cytoszkieletu
geny dla
receptorów laminin, integryn
i cząsteczki
CD44
w zakresie prawidłowego funkcjonowania
ECM; CD44 – transmembranowy glikoproteid na
powierzchni limfocytów, fibroblastów, komórek
nabłonka – oddziaływania komórka/komórka i
komórka/ECM - duża zmienność CD44 poprzez
potranslacyjną modyfikację lub alternatywne
składanie genu
CZYSTA DYSGENEZJA GONAD
CZYSTA DYSGENEZJA GONAD
(zespół Swyera; MIM 306100)
(zespół Swyera; MIM 306100)
CZYSTA DYSGENEZJA GONAD
CZYSTA DYSGENEZJA GONAD
(zespół Swyera; MIM 306100)
(zespół Swyera; MIM 306100)
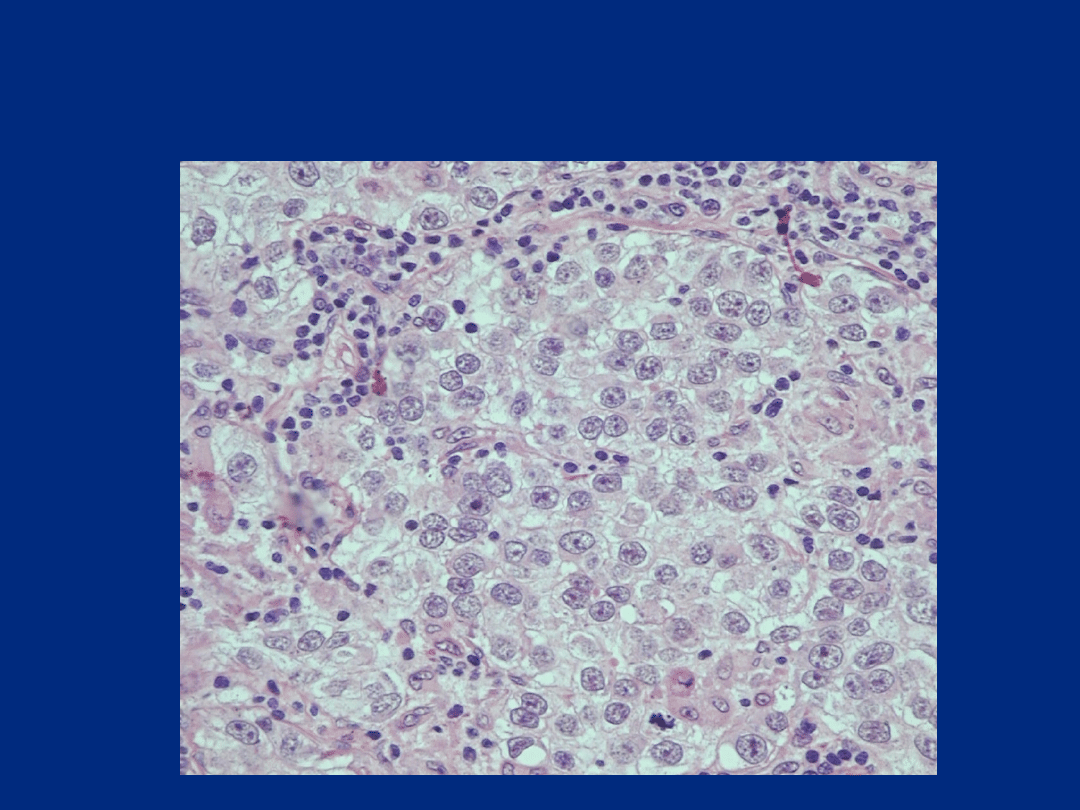
CZYSTA DYSGENEZJA GONAD
CZYSTA DYSGENEZJA GONAD
(zespół Swyera; MIM 306100)
(zespół Swyera; MIM 306100)
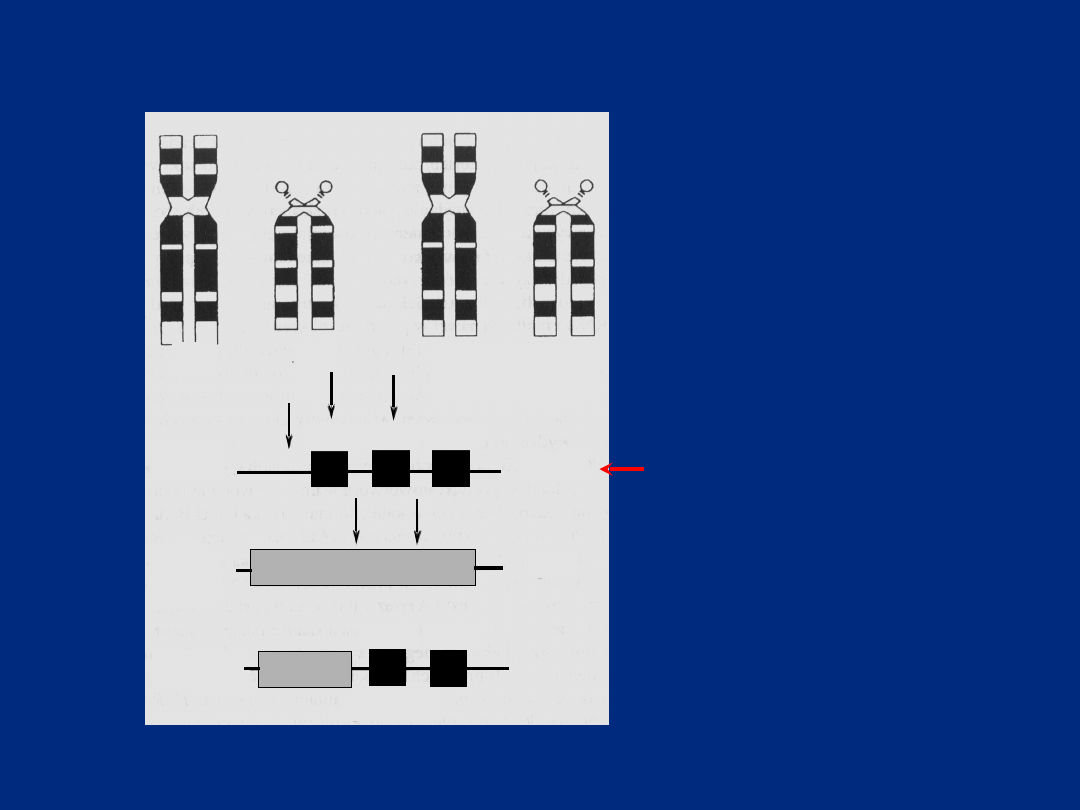
8
1
4
t(8;14)
(q24;q32)
chr
14
chr 8
ekson
1
ekson
3
ekson
2
ekson
2
ekson
3
t(8;14)
IgH-
MYC
IgH
c-
MYC
Aberracje chromosomowe w etiopatogenezie
nowotworów
Chłoniak
Burkitt’a
myelocytomatosis
oncogene1

ISCN (1995)
An International System for Human
Cytogenetic Nomenclature
Mitelman F. (wyd.): S. Karger, Basel,
1995.

chr 9
chr 22
chr
9
chr
22
ABL
BCR
ABL-
BCR
t(9;2
2)
9q34
.1
chromosom filadelfia (Ph’)
Abelson murine leukemia
viral oncogene homolog 1
break point cluster
region
22q1
1
Aberracje chromosomowe w etiopatogenezie
nowotworów
CML –
przewlekła
białaczka
szpikowa

Przykłady swoistych aberracji chromosomowych w
komórkach nowotworowych układu
krwiotwórczego
Choroba
Rodzaj aberracji
chromosomowej
Zaangażowa
ne geny
Ostra białaczka
limfoblastyczna (ALL)
t(1;19)(q23;p13)
t(4;11)(q21;q23)
PBX; EZA
AF4; MLL
Ostra białaczka szpikowa
(AML)
M2
M3
M4
M5
t(8;21)(q22;q22)
t(15;17)(q22;q11-21)
inv(16)(p13q22)
t(9;11)(p21;q23)
ETO; AML1
PML; RARA
MYH11
AF9;MLL
Mielodysplazja (MDS)
del(5q),-
7,+8,del(20q),-Y
Chłoniak złośliwy
t(14;18)(q32;q21)
BCL2
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
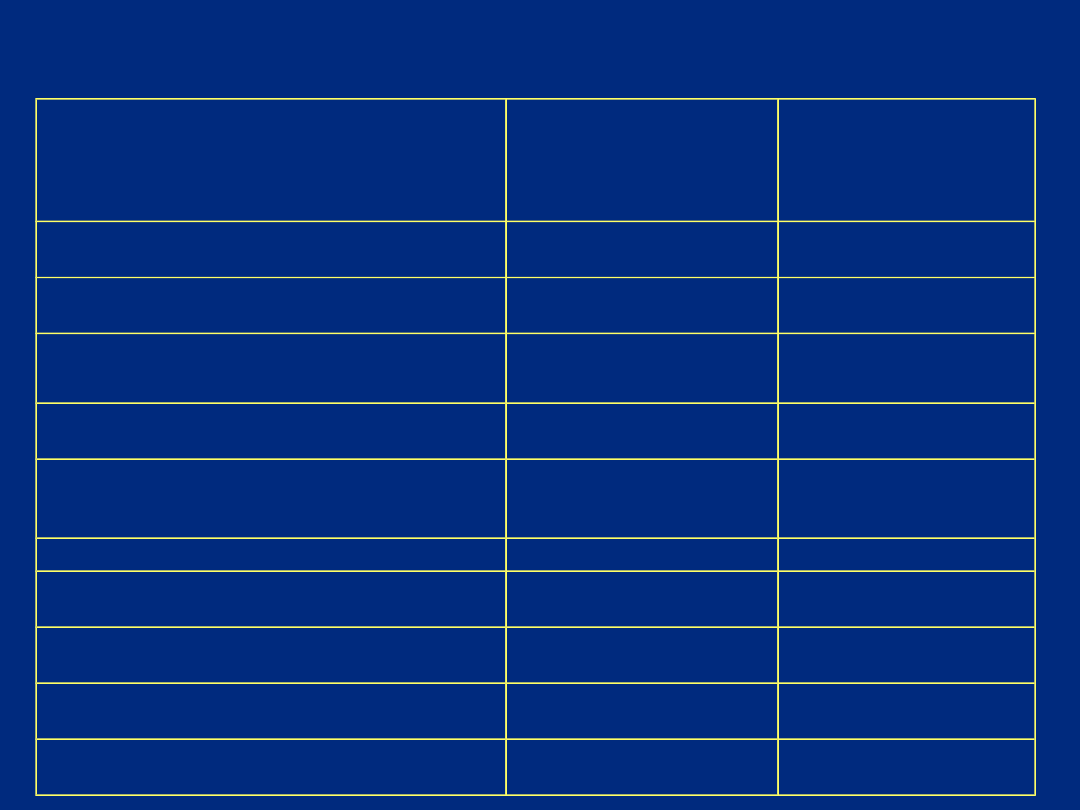
Przykłady aberracji chromosomowych typowych
dla komórek niektórych rodzajów złośliwych
guzów nowotworowych
Rodzaj nowotworu
Typ aberracji
chromosomowej
Geny fuzyjne
powstałe w
wyniku
translokacji
Tłuszczakomięsak śluzowaty
(liposarcoma myxoides)
t(12;16)
(q13.3;p11.2)
CHOP-TLS (FUS)
Tłuszczakomięsak
wysokozróżnicowany
+r(12)
Mięśniakomięsak prążkowano-
komórkowy pęcherzykowy
(rhabdomyosarcoma alveolare)
t(2;13)(q35-37;p14)
t(1;13)(p36;p14)
PAX3-FKHR
PAX7-FKHR
Mięsak jasnokomórkowy (sarcoma
clarocellulare)
t(12;22)(q13;q12)
ATF1-EWS
złośliwy włóniak histiocytarny
(fibrohistiocytoma malignum)
add(19)
(p13),r,tas,hsr,dic,d
min
Włókniakomięsak dziecięcy
+8,+11,+17,+20
Maziówczak złośliwy (synovioma
malignum)
t(X;18)(p11.2;q11.2) SSX1, SSX2-SYT
Guz Ewinga, PNET – prymitywne
nowotwory neuroektodermalne
t(11;22)(q24;q12)
FLI1-EWS
Pozaszkieletowy chrzęstniako-
mięsak śluzowaty
t(9;22)(q22;q12)
TEC-EWS
Drobnokomórkowy desmoplastyczny
guz dziecięcy jamy brzusznej
t(11;22)(p13;q12)
WT1-EWS
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
Na podstawie: Badania molekularne i cytogenetyczne w medycynie. Red. J. Bal, PWN 1998
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
- Slide 42
- Slide 43
- Slide 44
- Slide 45
- Slide 46
- Slide 47
- Slide 48
- Slide 49
- Slide 50
- Slide 51
- Slide 52
- Slide 53
- Slide 54
- Slide 55
- Slide 56
- Slide 57
- Slide 58
- Slide 59
- Slide 60
- Slide 61
- Slide 62
- Slide 63
- Slide 64
- Slide 65
- Slide 66
- Slide 67
- Slide 68
- Slide 69
- Slide 70
- Slide 71
- Slide 72
- Slide 73
- Slide 74
- Slide 75
- Slide 76
- Slide 77
- Slide 78
- Slide 79
- Slide 80
- Slide 81
- Slide 82
- Slide 83
- Slide 84
Wyszukiwarka
Podobne podstrony:
Nowotwory
Nowotworynew
Wybrane markery chorb nowotworowych
Leczenie bólu i opieka paliatywna w chorobach nowotworowych
Profilaktyka nowotworowa
nowotwory noworodek
Nowotwory złośliwe odbytnicy + TME prezentacja
Immunologia nowotworów
Markery nowotworowe 2
Leczenie uzupełniające nowotworów narządu rodnego chemioterapia, radioterapia
zmiany nowotworowe
więcej podobnych podstron