
ZMIENNOŚĆ I MUTACJE
Katedra i Zakład Biologii
Medycznej
AM w Bydgoszczy

ZMIENNOŚĆ
Zmienność-występowanie dziedzicznych
lub niedziedzicznych różnic:
▪ zmienność wewnątrzosobnicza -
pomiędzy komórkami danego organizmu
▪ zmienność osobnicza - pomiędzy
osobnikami należącymi do tej samej
populacji ,
▪ zmienność grupowa - pomiędzy
populacjami .

ZMIENNOŚĆ
1. MUTACYJNA
-
mutacje genowe
(tranzycje, transwersje,
delecje, insercje, inwersje)
-
mutacje chromosomowe
:
- strukturalne (inwersje, translokacje,
duplikacje,
delecje, izochromosomy, chromosomy
koliste)
- liczbowe-aneuploidie (monosomie,
nullisomie,
trisomie)
- euploidie (poliploidie) :
> autopoliploidie (triploidie, tetraploidie,
itd.)
> allopoloploidie (amfiploidie)

2. REKOMBINACYJNA
(rekombinacja
homologiczna,
rekombinacja zlokalizowana, rekombinacja
transpozycyjna)
3. FLUKTUACYJNA
(ciągła)
4. ALTERNATYWNA
(skokowa)
ZMIENNOŚĆ

• Zmienność fluktuacyjna (ciągła) – daje się
określić w jednostkach miary (wzrost, masa
ciała, IQ, liczba krwinek, pigmentacja włosów i
skóry)
• Zmienność alternatywna (skokowa, nieciągła) –
np. układ grupowy Rh
ZMIENNOŚĆ FENOTYPOWA

REKOMBINACJE
– procesy wymiany
fragmentów DNA między chromosomami
homologicznymi lub dwuniciowymi
helisami DNA. Rekombinacje nie prowadzą do
wytworzenia nowych alleli genów, ale do
ciągłego ich przetasowywania i powstawania
różnych kombinacji genotypów. Rekombinacje
mogą pojawiać się po mejozie, mitozie lub
koniugacji.
ZMIENNOŚĆ DZIEDZICZNA
(REKOMBINACJE I MUTACJE)

• U organizmów prokariotycznych rekombinacja
może wystąpić w wyniku :
- losowego doboru koniugantów
- rekombinacji zlokalizowanej,
transpozycyjnej,
homologicznej w czasie koniugacji
• U organizmów eukariotycznych :
- losowej segregacji chromosomów w
spermatogenezie i oogenezie
- losowego doboru rodziców
- losowego łączenia się gamet
- rekombinacji homologicznej (crossing over),
zlokalizowanej lub transpozycyjnej

• Zjawisko prowadzące do rekombinacji
genetycznej sprzężonych genów polegające na
wymianie odpowiadających sobie położeniem
odcinków między homologicznymi grupami
sprzężeń (chromosomami)
• Zachodzi w wyniku symetrycznych pęknięć i
ponownego połączenia się odcinków chromatyd
chromosomów homologicznych po ich wzajemnej
wymianie w pachytenie i/lub diplotenie mejozy
• Występuje również somatyczny , czyli mitotyczny
crossing over , który ma znaczenie w procesach
naprawy DNA. Występuje rzadko i z różną
częstością w chromosomach .
REKOMBINACJA HOMOLOGICZNA
(crossing over)
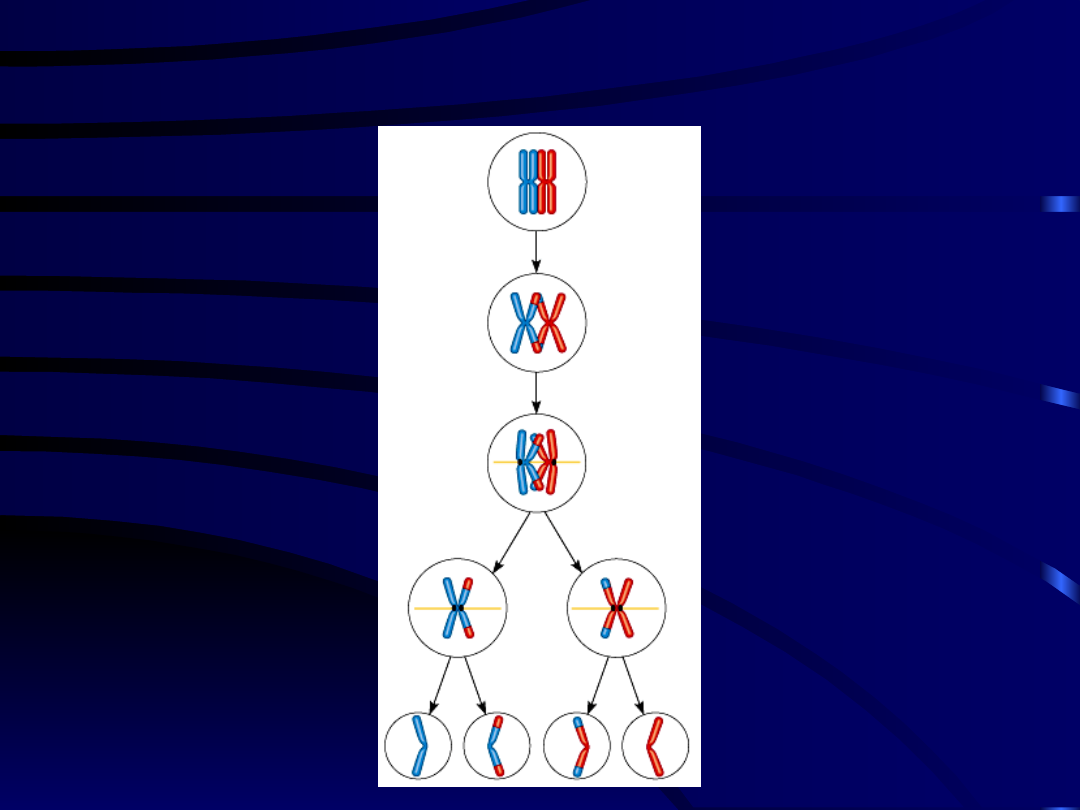
REKOMBINACJA HOMOLOGICZNA
(crossing over)

• Dotyczy wymiany niehomologicznych, ale
specyficznych fragmentów
• Jest katalizowana przez białka rozpoznające
specyficzne sekwencje zasad
• Przykładem tego typu rekombinacji jest tworzenie
przeciwciał i receptorów limfocytów T
• W komórkach bakteryjnych rekombinacja
zlokalizowana zachodzi podczas wbudowywania
plazmidów do genomu bakterii. Do przebiegu tego
procesu potrzebne są białka kodowane przez
plazmid i DNA bakterii.
REKOMBINACJA ZLOKALIZOWANA

• Zachodzi podczas wbudowywania
transpozonów w nowe miejsce genomu
• Transpozony są to ruchome elementy
genomu zawierające geny kodujące
transpozazę (enzym o aktywności nukleazy)
• Najprostszym przykładem transpozonów są
sekwencje insercyjne (IS)
• W rekombinacji transpozycyjnej wymagane
jest istnienie specyficznej sekwencji DNA w
miejscu akceptorowym dla IS. Sekwencja ta
ulega duplikacji, a IS wbudowywana jest
między podwojony fragment (podwojona
sekwencja).
REKOMBINACJA TRANSPOZYCYJNA

• Takie podwojenie sekwencji zasad
powoduje zwiększenie ilości
homologicznego materiału
genetycznego w komórce
• Rekombinacja pomiędzy podwojonymi
sekwencjami może prowadzić do
delecji, inwersji lub duplikacji genów
• Oprócz genów transpozazy transpozon
może zawierać inne geny – jest to tzw.
transpozon złożony
• U Eukaryota transpozony mają
budowę podobną do retrowirusów

• Termin „
mutacja
” wprowadził H. De Vries w
1909r
• Mutacja
jest to zmiana dziedziczna powstająca
na skutek zmiany genu w jego nowy allel
(mutacja genowa), zmiany struktury
chromosomu (mutacja chromosomowa),
zmiany liczby chromosomów (mutacja
liczbowa), bądź zwielokrotnienie haploidalnego
zestawu chromosomów (mutacja genomowa)
MUTACJE

• każda zmiana sekwencji nukleotydów w
obrębie genu , inna od sekwencji genu
wyjściowego (powstaje nowy allel genu)
• dotyczy genów kodujących białka
strukturalne i enzymatyczne
• zmiana sekwencji nukleotydów w DNA może
powstać w wyniku : tranzycji, transwersji,
delecji, insercji
MUTACJE GENOWE

• Tranzycja
– zamiana jednej zasady purynowej
na drugą purynową , lub pirymidynowej na
inną pirymidynową.
• Traswersja
– zamiana zasady purynowej na
pirymidynową lub odwrotnie.
• Delecja
– wypadnięcie pojedynczej lub większej
liczby par nukleotydów z danego genu.
• Insercja
– wstawienie pojedynczej lub większej
liczby par nukleotydów do danego genu .
MUTACJE GENOWE

• Następstwa mutacji genowych :
- zmiana sekwencji aminokwasów w
polipeptydzie kodowanym przez zmutowany
gen
- przerwanie syntezy łańcucha
polipeptydowego
• Mutacja typu zmiany sensu – na skutek
tranzycji lub transwersji dojdzie do zmiany
kodonów
• Mutacja niema – kodony będą synonimiczne –
kodujące ten sam aminokwas
• Mutacja „missens” – kodony będą różne – do
łańcucha polipeptydowego dołączony zostanie
nowy aminokwas
MUTACJE GENOWE

• Mutacje nonsensowne - powodują
powstanie nowych kodonów „stop” i
prowadzą do przerwania syntezy
białka
Wynikiem mutacji genowych u
człowieka są choroby monogenowe,
m.in.: mukowiscydoza,
fenyloketonuria, alkaptonuria,
albinizm, retinoblastoma
achondroplazja i inne

1. Aberracje chromosomowe strukturalne
• Mogą dotyczyć pojedynczej lub obu chromatyd
• Przyczyną jest przerwanie ciągłości
chromatydy lub chromosomu
ABERRACJE CHROMOSOMOWE
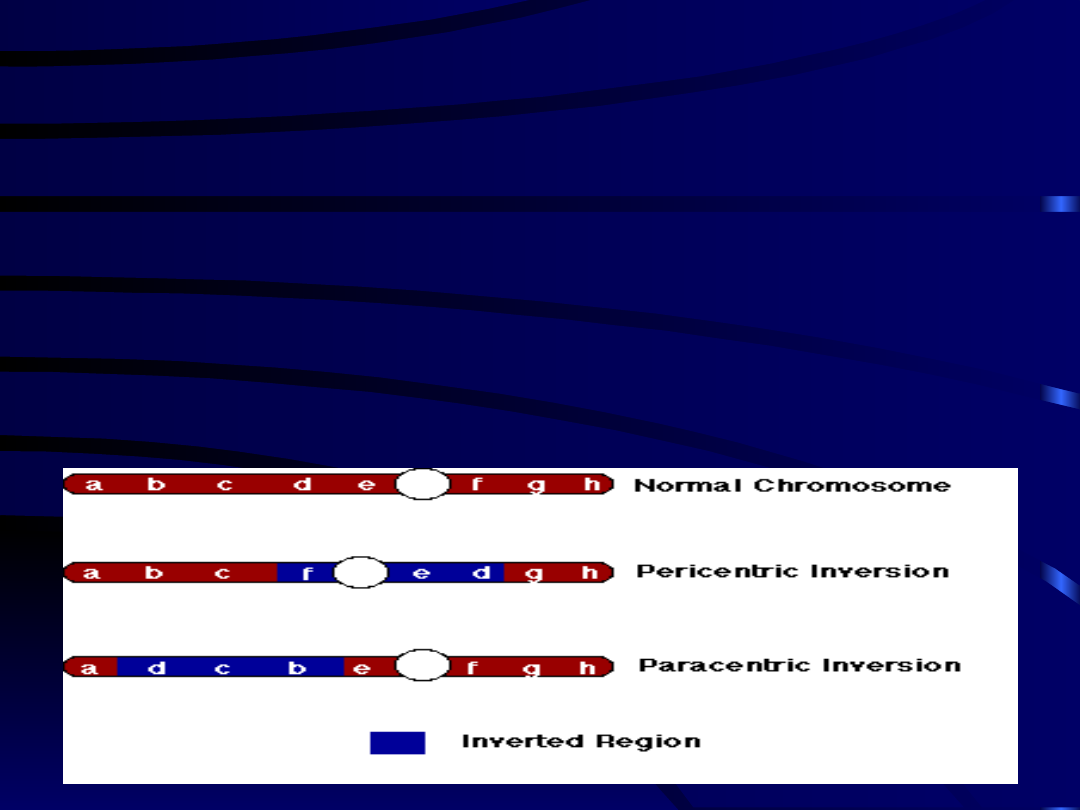
• Inwersja
– (odwrócenie fragmentu chromosomu o
180
o
) na skutek pęknięć jednego chromosomu i
połączenia wolnych jego końców w odwrotnym
kierunku .w następstwie zmiany pozycji genów w
chromosomie może dojść do zmiany ich ekspresji.
→ paracentryczna obejmuje odcinek
chromosomu bez
centromeru
→ perycentryczna obejmuje fragment z
centromerem

• Translokacja
– przemieszczenie się fragmentu
chromosomu w inne miejsce tego samego lub innego
chromosomu.
→ T. intrachromosomalna (wewnętrzna) między
homologicznymi
chromosomami
→ T. interchromosomalna (zewnętrzna) między chromosomami
niehomologicznymi
→ T. wymienna (wzajemna) wzajemna wymiana odcinków
między
chromosomami niehomologicznymi, całkowita liczba
chromosomów pozostaje nie zmieniona, a dwa spośród
nich mają
nieprawidłowe kształty .
ABERRACJE CHROMOSOMOWE
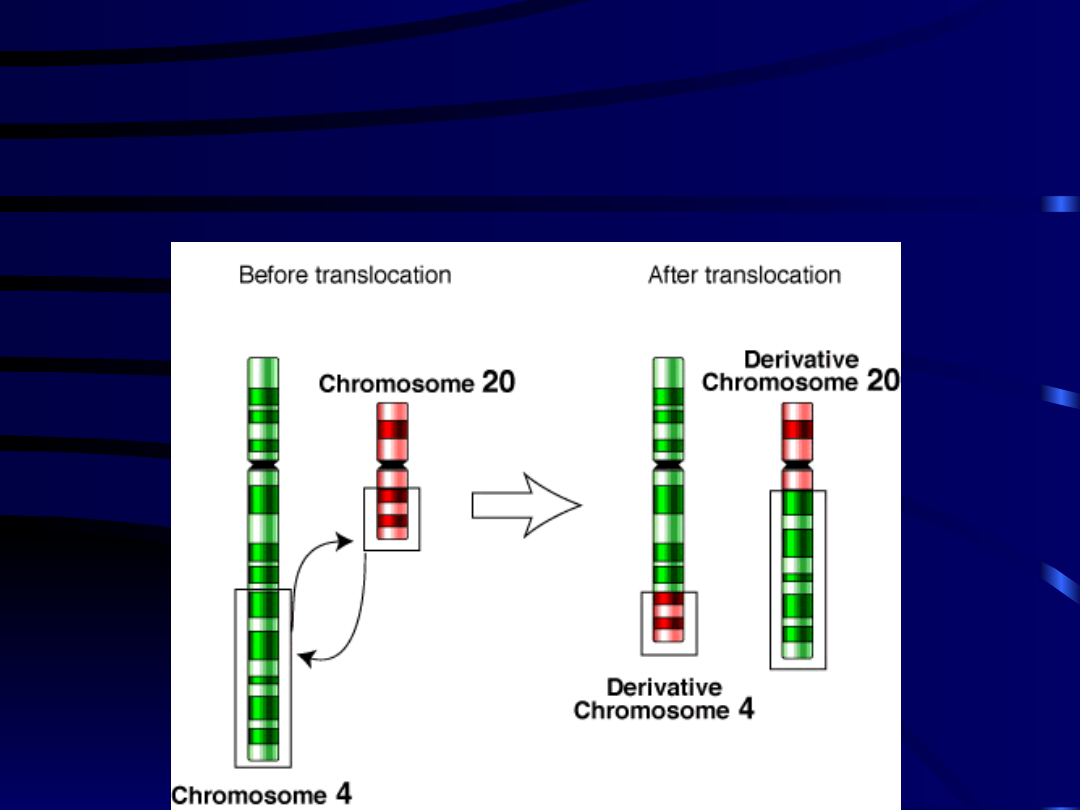
TRANSLOKACJA WYMIENNA
(WZAJEMNA)

TRANSLOKACJA ROBERTSONOWSKA (typu fuzji):
Łączą się całe lub prawie całe ramiona długie
dwóch różnych chromosomów (połączenia
centryczne). Miejscem połączenia jest rejon
centromeru. Dochodzi do utraty funkcjonalnie
nieistotnej części materiału genetycznego ( ramiona
krótkie ) .U człowieka dotyczą tylko chromosomów
akrocentrycznych.
Translokacja robertsonowska zrównoważona
-nie
zmienia się ilość materiału genetycznego, ale następuje
zmiana jego lokalizacji w genomie. Brak objawów
fenotypowych.
Translokacja robertsonowska niezrównoważona
-ilość
materiału genetycznego powiększa się o dodatkową
kopię translokowanego chromosomu. Fenotypowe
ujawnienie choroby
ABERRACJE CHROMOSOMOWE
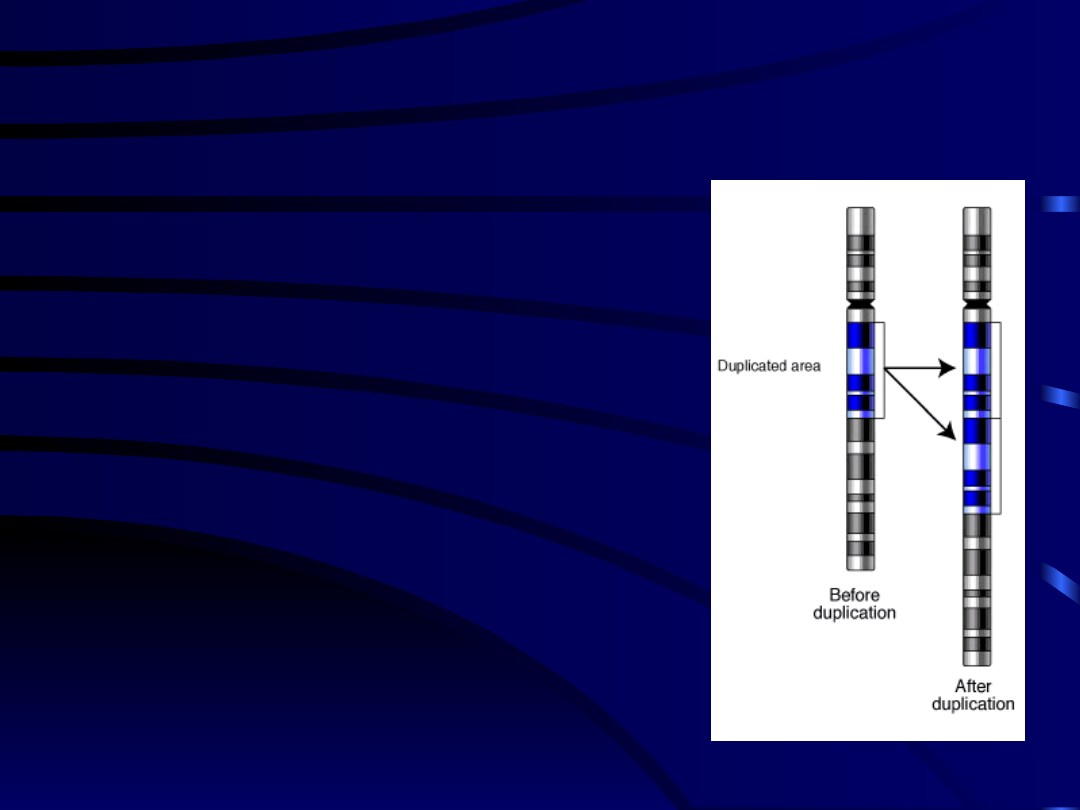
• Duplikacja
– podwojenie
tych samych odcinków
chromosomów (podwojenie
kopii genów). Podwojone
fragmenty mogą występować
jako bezpośrednie
powtórzenia (proste
powtórzenia tandemowe) lub
jako odwrócone względem
siebie powtórzenia
fragmentów chromosomów.
ABERRACJE CHROMOSOMOWE
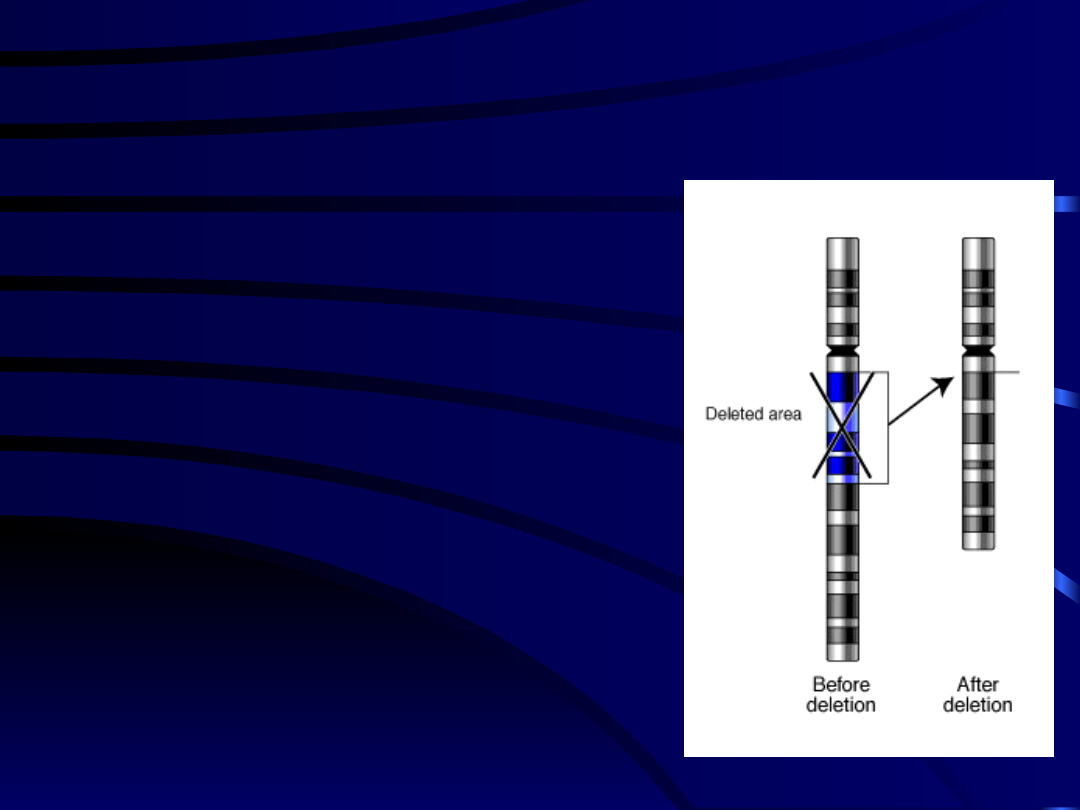
• Delecja (deficjencja)
–
utrata odcinka
chromosomu.
→ d. terminalna obejmuje
część
dystalną chromosomu
→ d. interstycjalna obejmuje
fragment środkowy
ABERRACJE CHROMOSOMOWE
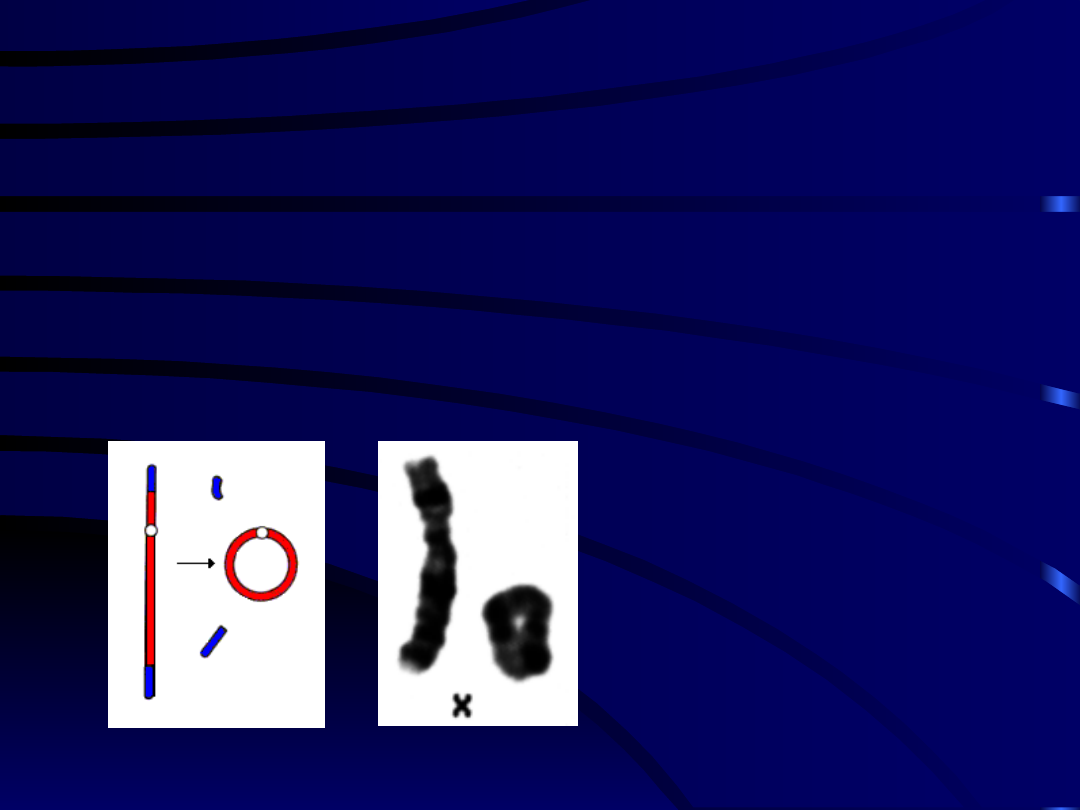
• Chromosom kolisty
– powstaje w wyniku
pęknięcia, a następnie połączenia końców
chromosomu.
U człowieka chromosomy koliste powstają
najczęściej z chromosomów 4 , 13 , 18 pary
oraz chromosomu X .
ABERRACJE CHROMOSOMOWE

• Izochromosom
– powstaje w
wyniku nieprawidłowego ,
poprzecznego podziału
centromeru chromosomu
metafazowego. Składa się on
tylko z połączonych ramion
długich lub krótkich. Powstanie
izochromosomów powoduje
ubytek genów zawartych w
utraconych ramionach i
podwojenie ich liczby w
ramionach , które utworzyły
chromosom. Powstają zarówno z
autosomów jak i chromosomu X .
ABERRACJE CHROMOSOMOWE

2. Aberracje liczbowe
• Aneuploidie
– powstają w wyniku zwiększenia
lub zmniejszenia diploidalnej liczby
chromosomów o pojedyncze chromosomy .
Powstawanie uniparentalnej disomii
(UPD)
- polega na obecności u diploidalnego
potomka pary
chromosomów pochodzących tylko od
jednego
rodzica .
ABERRACJE CHROMOSOMOWE
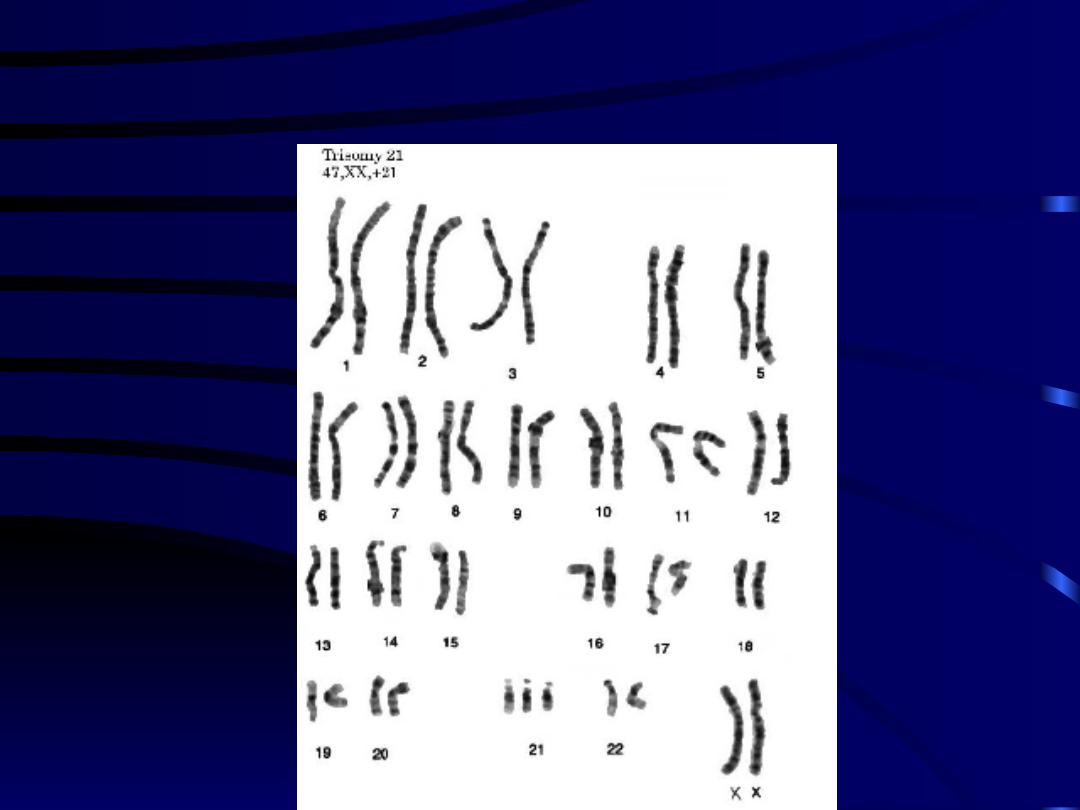
TRISOMIA 21 PARY CHROMOSOMÓW
47,XX,+21 (ZESPÓŁ DOWNA)

• Euploidie
– zwielokrotnienie całego podstawowego zespołu
chromosomów. Zapis 3n, 4n, 5n, itd.
→
autopoliploidie
- garnitur chromosomów jest
zwielokrotniony
o ten sam zestaw chromosomów.
Autopoliploidie są u człowieka letalne i prowadzą do
poronień.
np. AABB(2n) + AABB(2n) = AAAABBBB(4n)
→
allopoliploidie (amfiploidie)
zwielokrotnienie
niehomologicznych
zespołów chromosomów . Powstają najczęściej na skutek
podwojenia liczby chromosomów u mieszańców
międzygatunkowych. Ten typ aberracji nie występuje u
człowieka .
np. AABB(2n) + CCDD(2n) = AABBCCDD(4n)
ABERRACJE CHROMOSOMOWE

Mutageny
to czynniki indukujące powstawanie
mutacji znacznie ponad poziom mutacji
spontanicznych.
Mutageny mogą powodować:
• Transformację nowotworową (kancerogeneza)
• Wady rozwojowe (teratogeneza)
• Śmierć komórek (całego organizmu)
Czynniki mutagenne podzielono na:
• Fizyczne (np. UVA, UVB, promieniowanie X)
• Chemiczne (np. niektóre leki, środki konserwujące i
inne)
• Biologiczne (wirusy brodawczaka ludzkiego i inne)
CZYNNIKI MUTAGENNE

• uszkodzenia DNA, przed ich naprawą, są
rozpoznawane przez enzymy reparacyjne:
- polimerazy DNA: jądrowe (α, β, δ, ε) i
mitochondrialna (γ)
- ligazy DNA
- glikozylazy DNA
- endonukleazy apurynowe/apirymidynowe (5’-
AP i 3’-AP)
- białka pomocnicze
- fotoliazy DNA
- metyotransferaza O
6
-metyloguanina-DNA
(MGMT)
MECHANIZMY NAPRAWY DNA

• Naprawa DNA może być kompletna i niekompletna
(efektem są mutacje punktowe, aberracje
chromosomowe)
• W przypadku braku możliwości naprawy DNA komórka
powinna ulec programowanej śmierci komórki-apoptozie
• Procesy naprawy zachodzą w okresie
przedreplikacyjnym, podczas replikacji lub w okresie
poreplikacyjnym
• Szybkość naprawy chromatyny aktywnej transkrypcyjnie
jest większa niż nieaktywnej chromatyny
MECHANIZMY NAPRAWY DNA

-
usuwanie błędnie sparowanej zasady
- naprawa przez wycinanie zasad
azotowych
- naprawa przez wycinanie
nukleotydów
- naprawa rekombinacyjna
- odpowiedź SOS
MECHANIZMY NAPRAWY DNA

USUWANIE BŁĘDNIE
SPAROWANEJ ZASADY
• Dotyczy usuwania błędów replikacyjnych, nie
naprawionych przez polimerazę DNA, podczas
replikacji oraz błędów w parowaniu zasad
podczas rekombinacji DNA, a także w wyniku
działania niektórych związków chemicznych
• Etapy naprawy u E.coli :
- rozpoznanie miejsca pomyłki przez białko MutS
- połączenie białka MutS z DNA stabilizowane
przez białko MutL, które odpowiada za
połączenie z białkiem MutH
- białko MutH przyłącza się naprzeciw najbliżej
zmetylowanej adeniny w nici „rodzicielskiej” i
nacina nić potomną

- Procesy wycięcia nieprawidłowej nici i
dosyntetyzowanie prawidłowej
przebiega z udziałem : helikazy II,
egzonukleazy, polimerazy DNA III,
ligazy DNA
U Eukaryota mechanizm naprawy nie
został dostatecznie poznany.

NAPRAWA PRZEZ WYCINANIE
ZASAD AZOTOWYCH
• Mechanizm wykorzystywany głownie do usuwania
uracylu lub alkilowanych zasad np. 3-
metyloadeniny
• Etapy :
1. Usunięcie nieprawidłowej zasady przez
specyficzną N-glikozylazę i wytworzenie miejsca
apurynowego /apirymidynowego (miejsce AP).
Alkilowane zasady azotowe mogą również
oddysocjować od reszt dezoksyrybozy
samorzutnie.
2. Nacięcie przez endonukleazę AP wiązania
fosfodiestrowego powyżej miejsca AP i
wytworzenie wolnego końca 3’-OH

3. Wypełnienie miejsca AP przez polimerazę
DNA
Różnice pomiędzy poszczególnymi typami tego
mechanizmu dotyczą pierwszego etapu, w
którym występują różne glikozylazy,
specyficznie wycinające dany typ
nieprawidłowej zasady.
Powstające po wycięciu zasady miejsce AP jest
identyczne do powstającego po spontanicznej
depurynacji lub depirymidynacji.

NAPRAWA PRZEZ WYCINANIE
NUKLEOTYDÓW
• Rozpoznanie uszkodzenia
• Przyłaczenie kompleksu białkowego w miejscu
uszkodzenia
• Podwójne nacięcie uszkodzonej nici w miejscu
oddalonym o kilka nukleotydów od miejsca
uszkodzenia zarówno po stronie 3’, jak i 5’
• Usunięcie oligonukleotydu zawierającego
uszkodzenie pomiędzy nacięciami
• Wypełnienie powstałej luki przez polimerazę
DNA
• Połączenie wolnych końców przy udziale ligazy
DNA

NAPRAWA REKOMBINACYJNA
• Naprawa pęknięć dwuniciowych
• U Eukaryota wyróżnia się trzy szlaki
naprawy (rekombinacja
homologiczna, dopasowywanie
pojedynczych nici DNA, rekombinacja
niehomologiczna)
• U ssaków w zależności od pozycji w
cyklu komórkowym dominują różne
mechanizmy

ODPOWIEDŹ SOS
• W komórkach bakteryjnych uruchamiany w
przypadku powstania licznych mutacji lub została
zatrzymana replikacja
• W odpowiedzi bierze udział ok..20 różnych genów
(liczne z nich kodują syntezę enzymów naprawy
DNA)
• System SOS może po naprawie pozostawiać błędy
(system mutagenny)
• System ten jest uruchamiany, gdy wcześniejsze
procesy nie wystarczyły do usunięcia uszkodzeń
• Uruchomienie systemu zatrzymuje podział
komórki

DZIĘKUJĘ ZA UWAGĘ
Document Outline
- Slide 1
- Slide 2
- Slide 3
- Slide 4
- Slide 5
- Slide 6
- Slide 7
- Slide 8
- Slide 9
- Slide 10
- Slide 11
- Slide 12
- Slide 13
- Slide 14
- Slide 15
- Slide 16
- Slide 17
- Slide 18
- Slide 19
- Slide 20
- Slide 21
- Slide 22
- Slide 23
- Slide 24
- Slide 25
- Slide 26
- Slide 27
- Slide 28
- Slide 29
- Slide 30
- Slide 31
- Slide 32
- Slide 33
- Slide 34
- Slide 35
- Slide 36
- Slide 37
- Slide 38
- Slide 39
- Slide 40
- Slide 41
Wyszukiwarka
Podobne podstrony:
ZMIENNOSC I MUTACJE, fizjoterapia, biologia medyczna
2 Regulacja funkcji genów Zmienność i mutacje
Zmienność i mutacje
ZMIENNOŚĆ MUTACJE
ZMIENNOŚĆ i MUTACJE, BIOLOGIA MEDYCZNA
2 Regulacja funkcji genów Zmienność i mutacje
Zmienność, mutacje
Zmienność i mutacje
ZMIENNOSC I MUTACJE, fizjoterapia, biologia medyczna
Zmienność i mutacje
2 Regulacja funkcji genów Zmienność i mutacje
Zmienność i mutacje
Zmiennosc i mutacje
ZMIENNOŚCI I MUTACJE
Zmienność i mutacje
W5 Zmienność Podział mutacji
¦ćw 6 Zmienno Ť¦ç i mutacje
więcej podobnych podstron