
Endonukleazy restrykcyjne
– molekularne nożyczki

1869 Odkrycie DNA (Miescher).
1944 Funkcja DNA: przechowywanie informacji genetycznej (Avery)
1953 Podwójna helisa - odkrycie struktury DNA (Crick, Watson)
1955 Arthur Kornberg wyizolował polimerazę DNA
1966 Odczytanie kodu genetycznego (Khorana, Nirenberg i in.)
1966 Weiss i Richardson wyizolowali ligazę DNA
1974 Początek inżynierii genetycznej: przeniesienie genu do bakterii
(Boyer i in.)

Odkrycie enzymów restrykcyjnych
1962 r.
Arber i Dussoix
– wyjaśnili zjawisko restrykcji – ograniczenia
rozwoju fagów w
E. coli.
Zidentyfikowali endonukleazę,
która trawiła niechroniony ( o innym wzorcu metylacji) DNA.
Metylacja charakterystyczna dla gospodarza bakteryjnego
chroniła DNA przed restrykcją (trawieniem)
1968 r.
Arber i Linn
oczyścili enzym
EcoB
i wykazali jego działanie in
vitro. Enzym ten trawił DNA
E.coli
K ale nie trawil
E.coli
B
.
1968 r.
Smith, Wilcox, Kelly
(John Hopkins Univeristy) wyizolowali
pierwszy enzym II klasy i określili charakterystykę miejsca
rozpoznania dla
Hind
II
1972 r. Enzym ten został użyty do sporządzenia
pierwszej mapy
restrykcyjnej wirusa SV40 prez D. Nathansa
1978 r.
Nagroda Nobla dla Arbera, Smitha i Wilcoxa
za odkrycie
enzymów restrykcyjnych
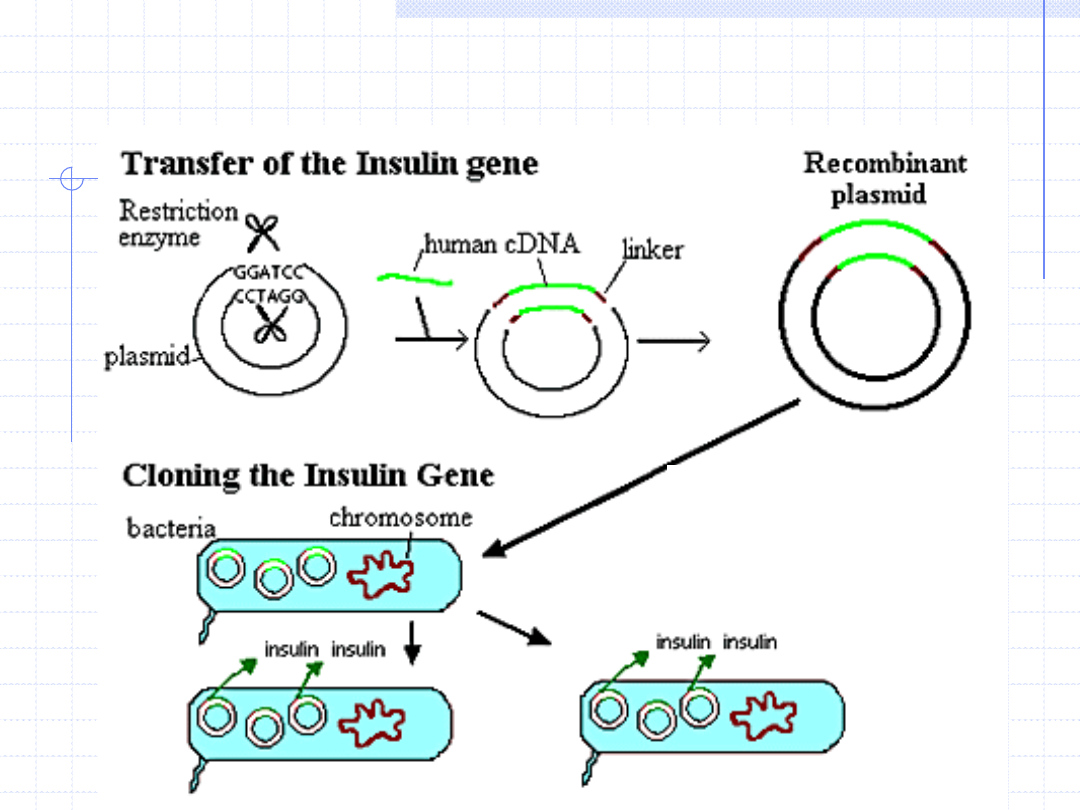
Transformation
Klonowanie genu ludzkiej insuliny i produkcja
zrekombinowanej insuliny w bakteriach
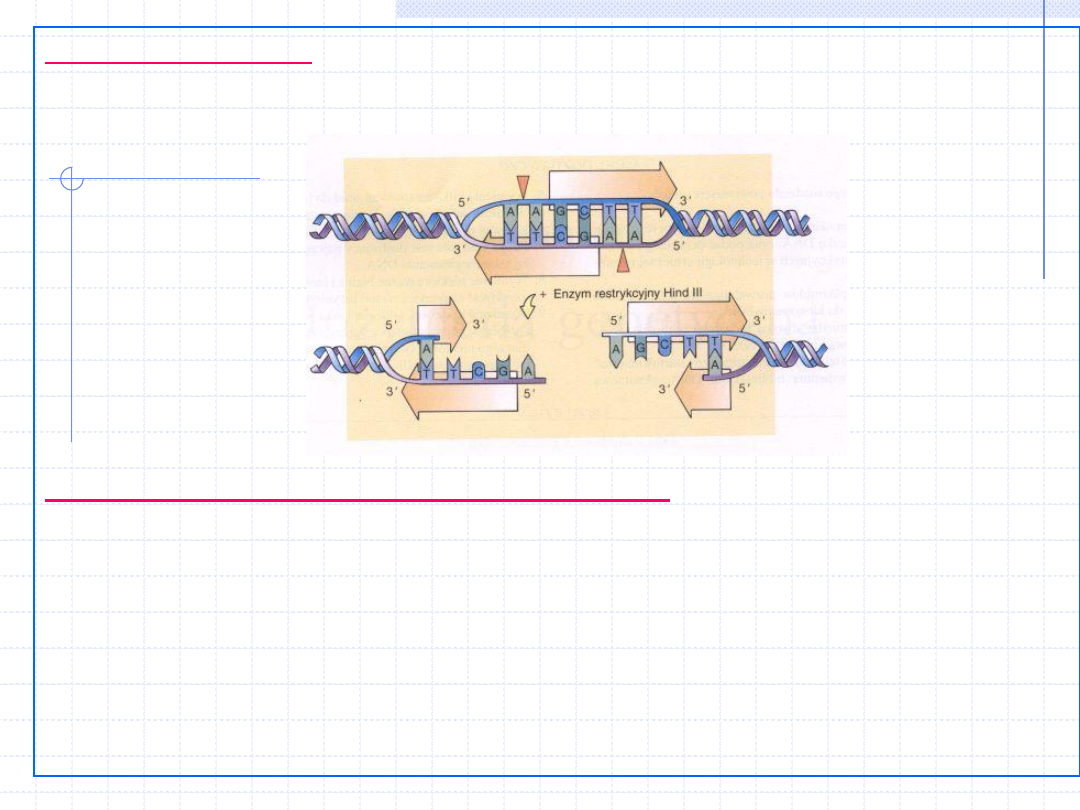
Enzymy restrykcyjne
to endonukleazy
–
przecinają szkielet cukier-fosforan w DNA
Podział enzymów restrykcyjnych na klasy I, II i III
Główna różnica dotyczy enzymów
II klasy
, gdzie system restrykcji jest
oddzielony od systemu metylacji
.
W klasie I i III te dwie aktywności są w obrębie jednego kompleksu
enzymatycznego, złożonego z dwóch lub trzech heterologicznych
podjednostek.
Enzymy poszczególnych klas różnią się także wymaganiami co do kofaktorów

Enzyme Organism from which derived
Target sequence
(cut at *) 5' -->3'
Ava I
Anabaena variabilis
C* C/T C G A/G G
Bam HI
Bacillus amyloliquefaciens
G* G A T C C
Bgl II
Bacillus globigii
A* G A T C T
Eco RI
Escherichia coli RY 13
G* A A T T C
Eco RII
Escherichia coli R245
* C C A/T G G
Hae III
Haemophilus aegyptius
G G * C C
Hha I
Haemophilus haemolyticus
G C G * C
Hind III
Haemophilus inflenzae Rd
A* A G C T T
Hpa I
Haemophilus parainflenzae
G T T * A A C
Kpn I
Klebsiella pneumoniae
G G T A C * C
Mbo I
Moraxella bovis
*G A T C
Mbo I
Moraxella bovis
*G A T C
Pst I
Providencia stuartii
C T G C A * G
Sma I
Serratia marcescens
C C C * G G G
SstI
Streptomyces stanford
G A G C T * C
Sal I
Streptomyces albus G
G * T C G A C
Taq I
Thermophilus aquaticus
T * C G A
Xma I
Xanthamonas malvacearum
C * C C G G G
Nazewnictwo
(Smith i
Nathans)
Nazewnictwo
enzymów
restrykcyjnych
opiera się na
literowych
skrótach, w
których
pierwsza litera
pochodzi od
rodzaju bakterii,
a druga i trzecia
od gatunku.
Następna litera
oznacza szczep
lub typ, a
kolejne enzymy
z danego typu
lub szczepu
otrzymują
liczby rzymskie.
Endonukleazy restrykcyjne II klasy
•Posiadają rozdzieloną aktywność restryktazy i metylazy, ich geny zwykle
sąsiadują w genomie
•Substratem jest dwuniciowe DNA -
dsDNA
(nieliczne enzymy mogą przecinać
określone sekwencje w jednoniciowym ssDNA).
•W większości przypadków enzymy przecinają obydwa pasma DNA w obrębie
krótkiej sekwencji (4 -12 nukleotydów), która nazywa się
miejscem rozpoznania
(recognition site).
•Większość miejsc rozpoznania jest
ścisłym palindromem
4, 5, 6, 7, 8 nt z
symetrią rotacyjną
.
Niekiedy palindrom może być przerwany przez serie 1-9 dowolnych nt
np. Sfi I GGCCNNNNNGGCC
Większość enzymów restrykcyjnych nie przecina zmetylowanego DNA na
jednym lub obu pasmach ich miejsca rozpoznania,
choć nieliczne wymagają
metylacji
•do aktywności in vitro bezwzględnie wymagają jonów magenzu

Długość sekwencji rozpoznawanej warunkuje
częstość trawienia DNA
np.: (zakładając że G+C = 50%)
enzymy z 8 nt sekwencją rozpoznania tną DNA co ok.. 65
kpz (slużą np. do konstrukcji bibliotek genomowych)
z 6 nt sekwencją rozpoznania tną DNA co 4096 bp (4
6
)
(wygodne do klonowania genów)
4 nt sekwencją rozpoznania trawioną DNA z częstością co
256 bp (4
4
) (tworzenie tzw. odcisków palca DNA –
fingerpinting)
Enzymy rzadkotnące
:
•Rozpoznają 7-8 nukleotydowe sekwencje np.:
SapI
5’- GCTCTTC -3’
Sgf1
5’- GCGATCGC -3’
•Enzymy, które rozpoznają sekwencje bogate w pary GC albo AT
SmaI
rozpoznaje 5’ – CCCGGG -3’, trawi co 78 kb;
NotI rozpoznaje
5’ –GCGGCCGC – 3’ , trawi co 10 MB
.
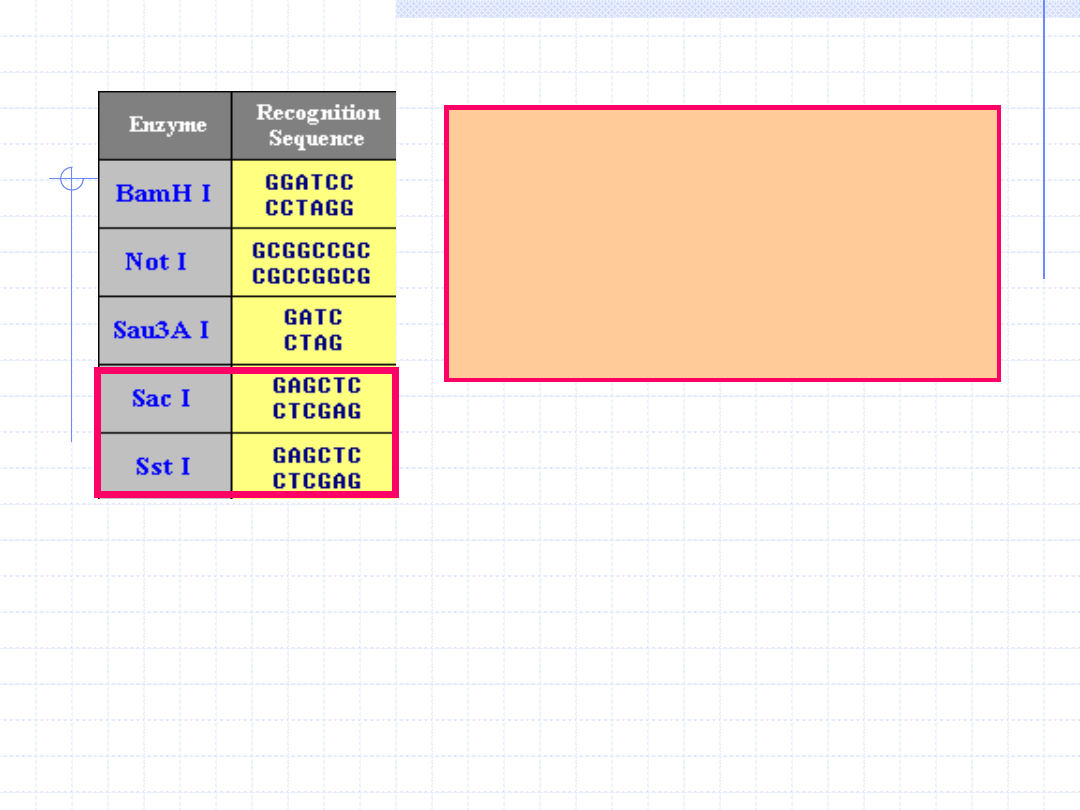
Niektóre enzymy pochodzące
z różnych bakterii rozpoznają
identyczne sekwencje, są to
tzw.
izoschizomery
i mogą
przecinać te sekwencje w
dokładnie taki sam sposób
Neoizoschizomery
– enzymy rozpoznające taką samą sekwencję, ale
przecinające ją w inny sposób.
Przykłady
:
SmaI
5’…CCC▼GGG…3’
XmaI
5’…C▼CCGGG…3’
SacI
5’…GAGCT▼C…3’
SstI
5’…GAGCT▼C…3’
EcoICRI
5’…GAG▼CTC…3’
Ecl136
5’…GAG▼CTC…3’
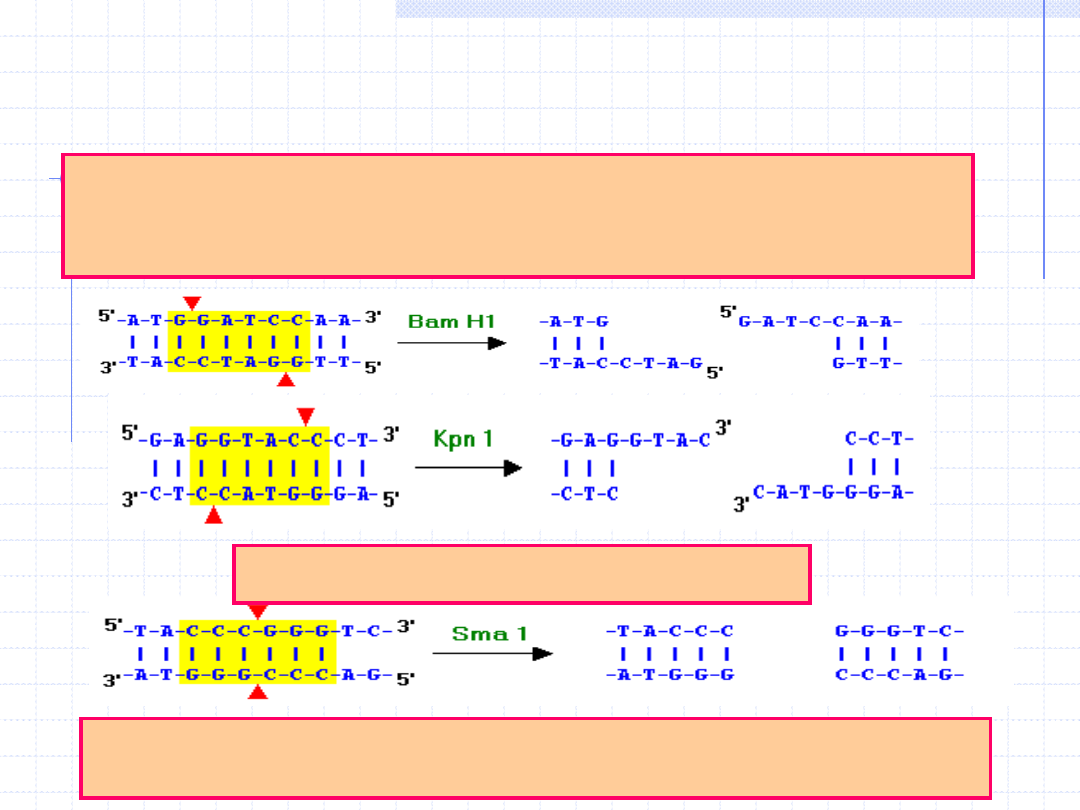
Lepkie końce
mogą być różnej długości (
zwykle są 2 lub 4 nt, ale
mogą być także 1 lub 3 nt
) i mogą mieć nadmierności typu 5’ lub 3’,
w zależności od użytego enzymu restrykcyjnego.
Mogą też tworzyć tępe końce
Trawienie enzymami restrykcyjnymi tworzy
wolne końce DNA
Większość enzymów trawi DNA generując końce z 5’- fosforanem lub
3’ –OH (wyjątek np.
Nci
I tworzy końce: 3’-fosforan i 5’-OH)
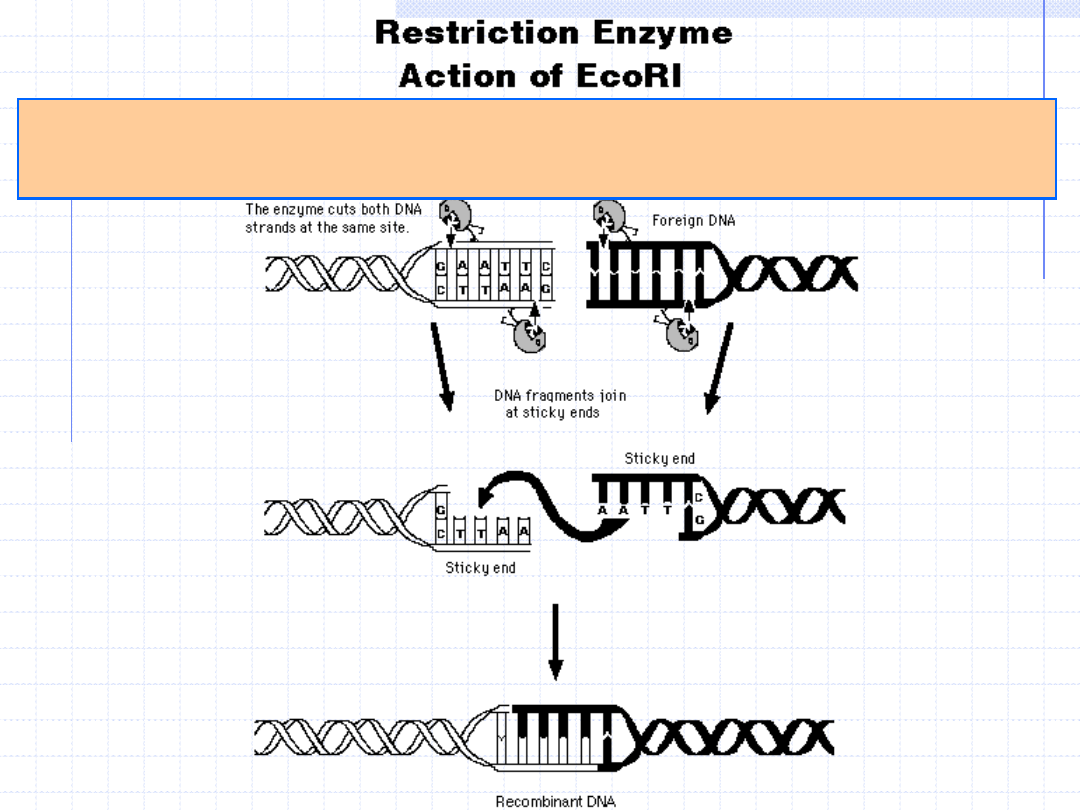
Jeśli lepkie końce są komplementarne,
ligaza
może je
łączyć
niezależnie od pochodzenia DNA,
tworząc formy rekombinacyjne.

Zastosowanie enzymów restrykcyjnych
1. Izolacja i identyfikacja genów
2. Rekombinowanie i klonowanie genów
3. Mapowanie fizyczne genomów
4. Diagnostyka (np. chorób) i identyfikacja (np.
ustalanie pokrewieństwa

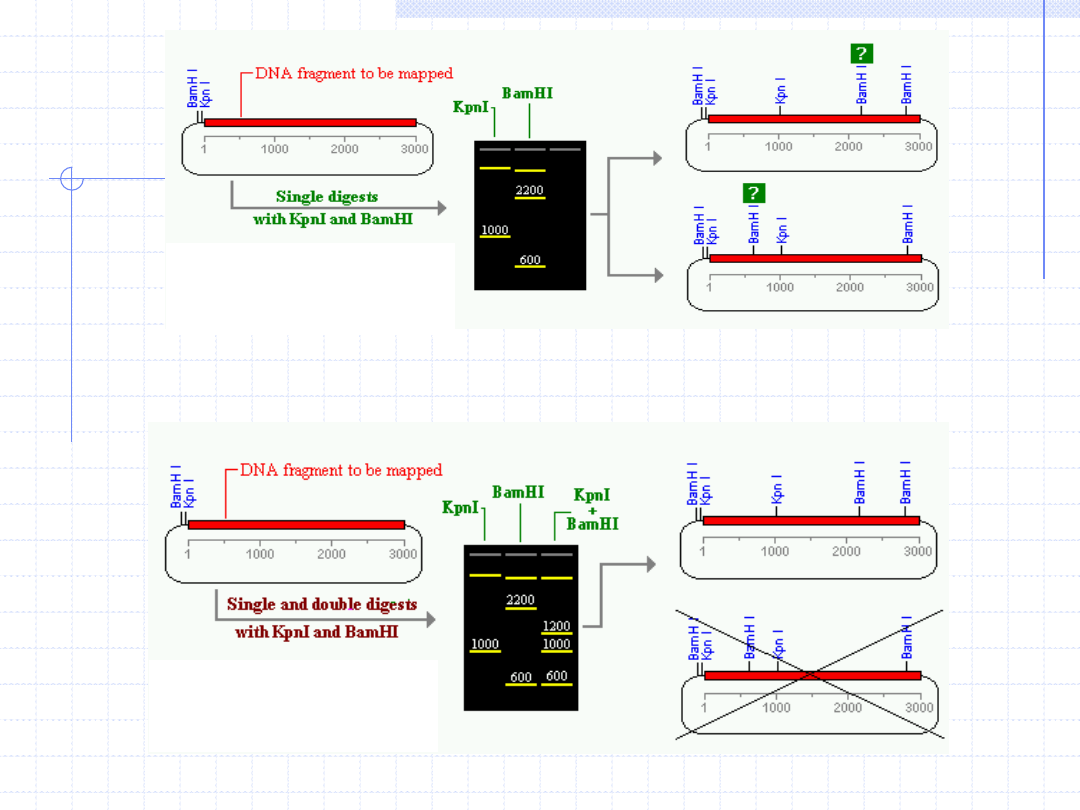
Mapowanie restrykcyjne
Mapa restrykcyjna
zawiera dokładną lokalizację miejsc restrykcyjnych w
obrębie fragmentu (cząsteczki) DNA.
Najprostszą metodą jest trawienie próbek DNA pojedynczymi enzymami, a
następnie parami tych enzymów. Produkty trawienia są rozdzielane w żelu
agarozowym dla określenia wielkości fragmentów, a następnie odtwarza się
kolejność ułożenia enzymów danym fragmencie lub cząsteczce.
Pojedyncze trawienie zwykle mówi ile jest fragmentów restrykcyjnych w
badanym DNA, a podwójne trawienie mówi o kolejności i orientacji
fragmentów.
Warunkiem jest całkowite strawienie fragmentów.
Nakładanie się dwóch identycznych pod względem wielkości fragmentów jest
widoczne jako jaśniejsze świecenie.
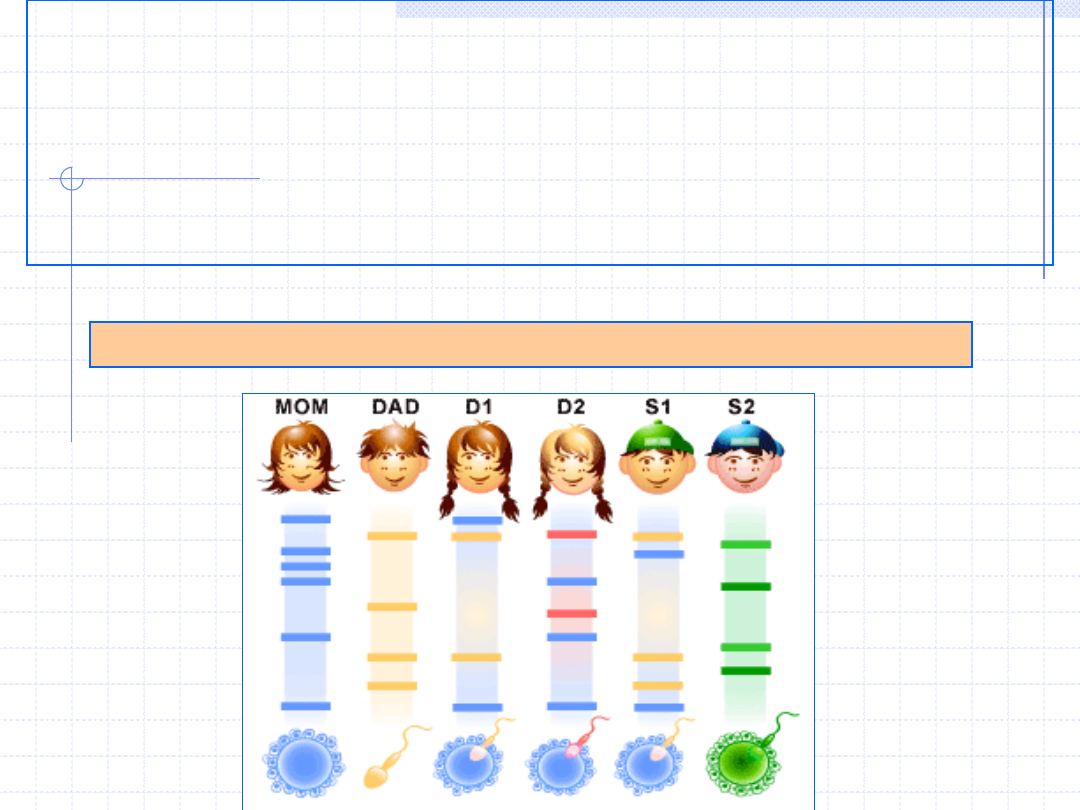
Zastosowanie enzymów w diagnostyce
chorób genetycznych
Analiza polimorfizmu DNA
RFLP
–
restriction fragment length polymorphism.
Różnice w dłg.
fragmentów restrykcyjnych tworzonych po cięciu DNA restryktazami II
typu.
Wykrywa się głównie przez trawienie DNA amplifikowanego w reakcji
PCR i elektroforezę agarozową produktów reakcji (PCR-RLFP).
Polimorfizm restrykcyjny może być stosowany do oznaczania ojcostwa
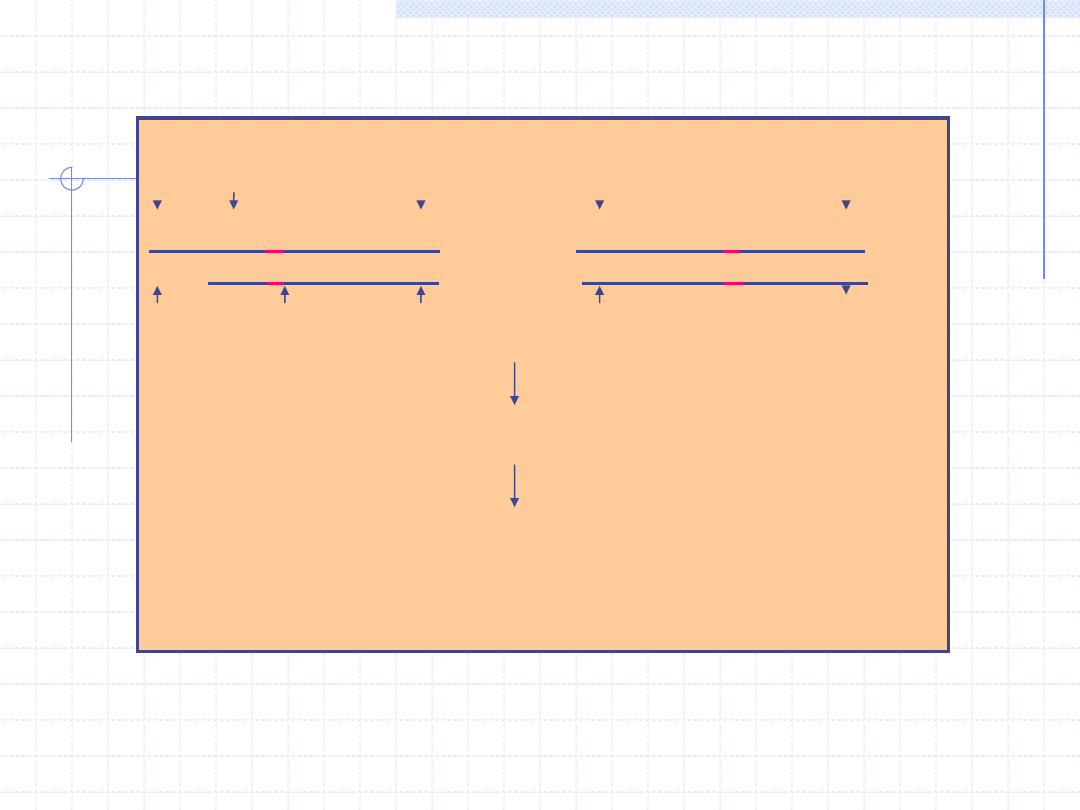
Normalny gen globiny
Zmutowany gen globiny
___________________
___________________
____CTG
A
G_________ ______CTG
T
G_______
____GAC
T
C_________ _____ GAC
A
C_______
Trawienie enzymem Dde I
Elektroforeza agarozowa
Dwa
fragmenty o dłg:
Jeden
fragment o dłg. 376 bp
201 i 175 bp
Wykrywanie mutacji przy pomocy enzymów restrykcyjnych
np.: anemia sierpowata
Wyszukiwarka
Podobne podstrony:
enzymy restrykcyjne
Biochemia TZ wyklad 3 enzymy low
Enzymy restrykcyjne referat
ENZYMY RESTRYKCYJNe
1001562-enzymy restrykcyjne, semestr IV, genetyka, Genetyka
Inżynieria genetyczna enzymy restrykcyjne
enzymy restrykcyjne-stud, Studia, Inżynieria genetyczna
enzymy restrykcyjne-stud, Biologia molekularna
Enzymy restrykcyjne
Enzymy restrykcyjne
Biochemia wykład enzymy, kinetyka
1 Trawienie wektora enzymy restrykcyjne
enzymy restrykcyjne
Biochemia TZ wyklad 3 enzymy low
Enzymy restrykcyjne i reakcja łańcuchowa polimerazy(PCR) zastosowanie (2)
enzymy restrykcyjne
więcej podobnych podstron