
Enzymy restrykcyjne
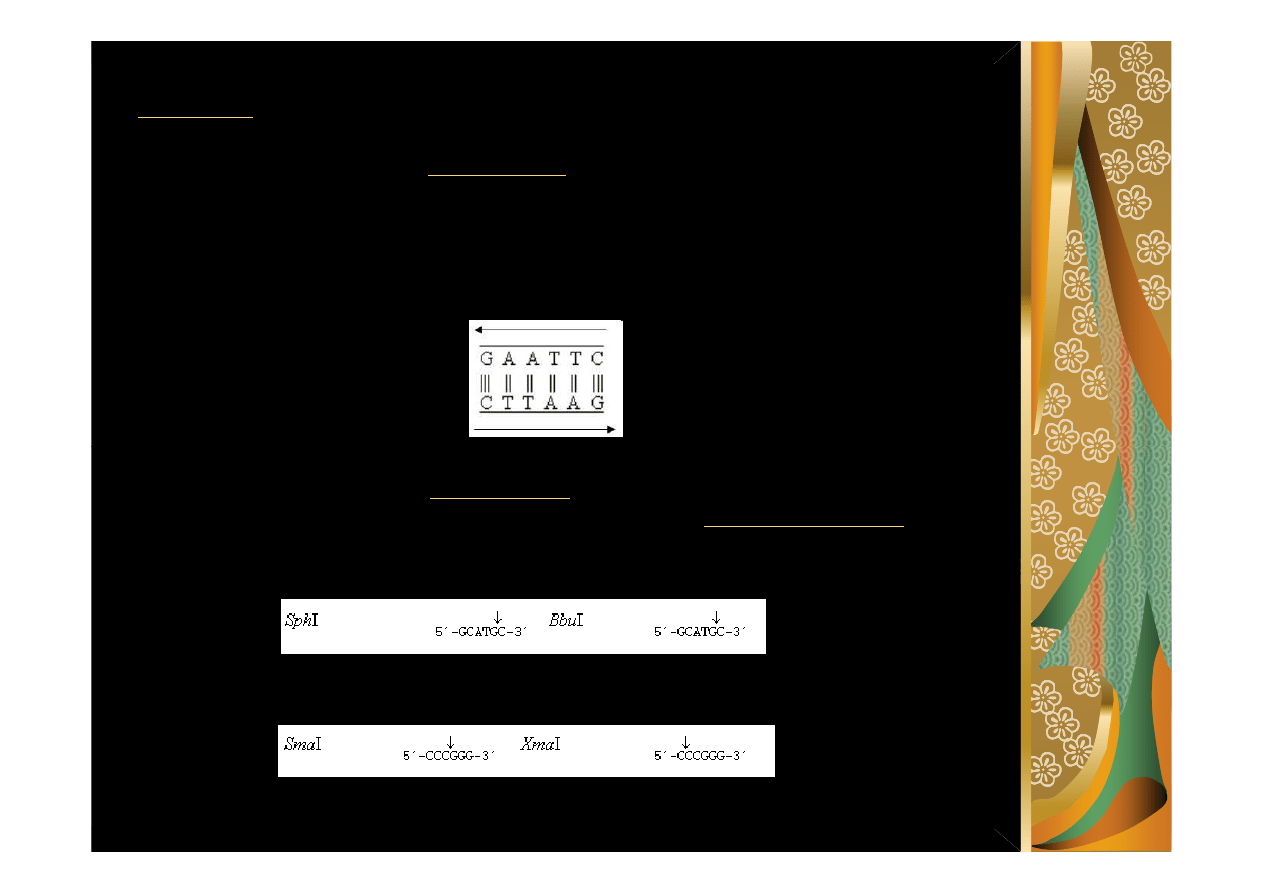
Restryktazy
(inaczej enzymy restrykcyjne , endonukleazy restrykcyjne) - to enzymy
izolowane z bakterii , zdolne do rozpoznawania specyficznych sekwencji w DNA
(z reguły są to sekwencje
palindromowe
) i do przecinania dwuniciowej cząsteczki
DNA w ściśle określonym miejscu , w obrębie lub okolicy sekwencji
rozpoznawanej. Otrzymywane fragmenty DNA nie są losowe a w każdym prążku
na żelu znajdują się cząsteczki DNA o identycznej sekwencji nukleotydowej. Z
reguły różne enzymy rozpoznają odmienne sekwencje DNA.
Istnieją jednak wyjątki - tzw.
izoschizomery
- enzymy izolowane z różnych
organizmów ale rozpoznające te same sekwencje oraz
heteroizoschizomery
-
enzymy rozpoznające te same sekwencje ale tnące w innym miejscu.

Typy enzymów:
Typ I
- wielopodjednostkowe kompleksy enzymatyczne zawieraj
ą
ce aktywno
ś
ci
metylazy i restryktazy. Przecinaj
ą
DNA z dala od rozpoznawanej sekwencji, w bli
ż
ej
nieokre
ś
lonym miejscu. Z tego powodu nie maj
ą
wi
ę
kszego zastosowania
praktycznego.
Typ II
- przecinaj
ą
DNA w zdefiniowanym miejscu, w obszarze rozpoznawanej
sekwencji lub w jej pobli
ż
u. Składaj
ą
si
ę
z pojedynczych polipeptydów. Rozpoznaj
ą
sekwencje symetryczne.
Typ IIs
- zbli
ż
one do typu II, przecinaj
ą
z jednej strony rozpoznawanej sekwencji, która
jest asymetryczna.
Typ III
- du
ż
e kompleksy, które wymagaj
ą
dwóch sekwencji rozpoznawanych w pobli
ż
u
siebie. Bez znaczenia praktycznego.
Typ IV
- zbli
ż
one do typu II. Zawieraj
ą
aktywno
ść
metylazy i restryktazy w tym samym
polipeptydzie. Przecinaj
ą
DNA w zdefiniowanym obszarze poza sekwencj
ą
rozpoznawania. Aktywno
ś
ci metylazy i restryktazy nie mog
ą
działa
ć
równocze
ś
nie.
Enzym "przeł
ą
cza" si
ę
w zale
ż
no
ś
ci od substratu.

Enzymy restrykcyjne klas I-III
Charakterystyka
Typ I
Typ II
Typ III
Aktywność
restrykcyjna i
modyfikacyjna
Pojedynczy enzym
wielofunkcyjny
Oddzielne
restryktazy i
metylazy
Oddzielne e nzymy ze
wspólna podjednostką
Struktura białkowa
endonukleazy
restrykcyjnej
Trzy różne
podjednostki
Prosta
Dwie różne
podjednostki
Wymagania do
restrykcji
ATP, Mg
2+
,
S-adenozylometionina
Mg
2+
ATP, Mg
2+
,
(S-adenozylometionina)
restrykcji
S-adenozylometionina
(S-adenozylometionina)
Charakterystyka
rozpoznawanej
sekwencji
EcoB: TGAN
8
TGCT
EcoK:AACN
6
GTGC
Symetria
rotacyjna
(palindromowa)
(nie dotyczy
IIs)
EcoP1: AGACC
EcoP15: CAGCAG
Miejsce trawienia Przypuszczalnie
przypadkowe, co
najmniej 1000 bp od
miejsca rozpoznawanej
sekwencji
W lub w
pobliżu miejsca
rozpoznawanej
sekwencji
24-26 bp po stronie 3’
od miejsca
rozpoznawanej
sekwencji
Przemieszczenie
DNA
Tak
Nie
Nie
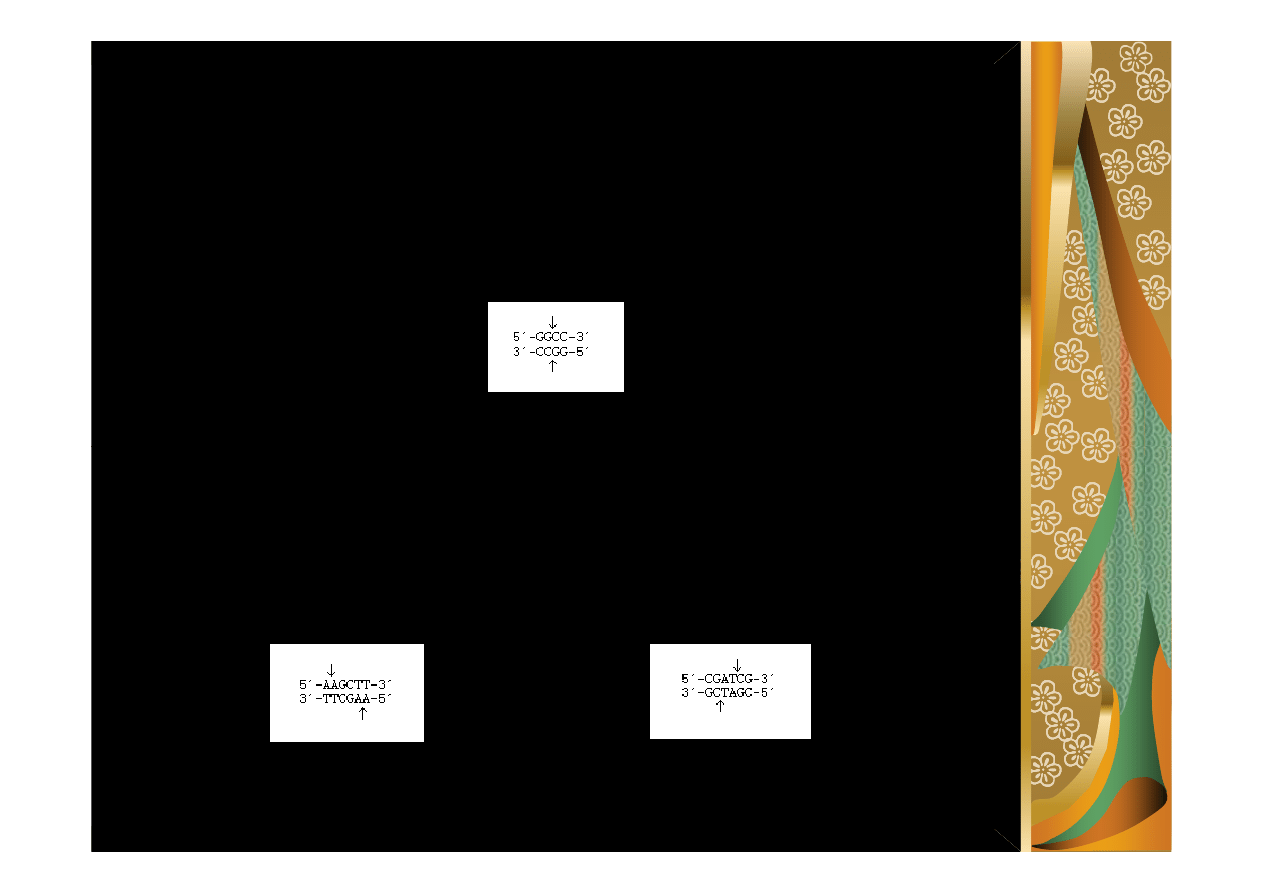
Podział według rodzaju wytwarzanych końców:
tępe końce
- nici rozcięte naprzeciwko siebie - wszystkie nukleotydy są sparowane
z komplementarnymi nukleotydami na przeciwnym łańcuchu
lepkie końce
- 3` lub 5` ssDNA (jednoniciowe) ogony na obu końcach utworzone
lepkie końce
- 3` lub 5` ssDNA (jednoniciowe) ogony na obu końcach utworzone
przez niesymetryczne cięcie , komplementarne do podobnych tworzonych w
innych cząsteczkach DNA przez te same enzymy restrykcyjne niezależnie od
ź
ródła DNA , co pozwala na ligowanie DNA nawet bardzo różniących się
gatunków czyli formowanie chimerycznych molekuł.

Podział według sekwencji rozpoznawanej:
czwórkowe
- rozpoznają sekwencję DNA złożoną z czterech nukleotydów.
Statystycznie w dowolnym DNA takich miejsc jest dużo - co 256 bp.
Restryktazy takie mogą strawić DNA na bardzo małe kawałki.
szóstkowe
- rozpoznają sekwencję DNA złożoną z sześciu nukleotydów.
Dowolne miejsce restrykcyjne złożone z sześciu nukleotydów występuje
statystycznie co około 4096 bp w DNA , w którym ilości poszczególnych
nukleotydów są równe. Proporcje te zmieniają się w zależności od organizmu ,
dlatego dobór enzymu szóstkowego i warunki należy ustalić
dlatego dobór enzymu szóstkowego i warunki należy ustalić
eksperymentalnie.
ósemkowe
- stosowane niezbyt często. Tną DNA bardzo rzadko.

Nomenklatura endonukleaz
Nazewnictwo opiera się na literowych skrótach, w których:
•pierwsza litera pochodzi od rodzaju bakterii ,
•druga i trzecia od gatunku,
•następna litera oznacza szczep lub typ,
•kolejne enzymy z danego szczepu lub typu otrzymują litery rzymskie
Przykłady nomenklatury:
-
NgoM IV - czwarty enzym wyizolowany z Neisseria gonorrhoeae szczepu MS11.
-
Uba 58 I - enzym wyizolowany z niezidentyfikowanej bakterii (Unidentified bacterium)
RFL58
-
BamH I - pierwszy enzym wyizolowany z Bacillus amyloliquefaciens szczepu H.

Restryktazy klasy II
Restryktazy klasy II

Zastosowanie enzymów restrykcyjnych:
Konstrukcja map restrykcyjnych
- Analizuj
ą
c na
ż
elach produkty trawienia danego DNA
ró
ż
nymi enzymami restrykcyjnymi , stosowanymi pojedynczo , w kombinacjach oraz w
ilo
ś
ciach wystarczaj
ą
cych lub niewystarczaj
ą
cych do pełnego strawienia preparatu , mo
ż
na
ustali
ć
wzajemne poło
ż
enie i odległo
ś
ci pomi
ę
dzy sekwencjami rozpoznawanymi przez te
enzymy. Mapa restrykcyjna to obraz cz
ą
steczki DNA , na którym zaznaczone s
ą
miejsca
rozpoznawane przez ró
ż
ne enzymy restrykcyjne z uwzgl
ę
dnieniem odległo
ś
ci mi
ę
dzy nimi
wyra
ż
onej w nukleotydach.
Klonowanie i obróbka DNA
- DNA poci
ę
ty enzymami restrykcyjnymi mo
ż
na poddawa
ć
ligacji z wektorem. W ten sposób tworzone s
ą
zrekombinowane plazmidy czyli plazmidy
zawieraj
ą
ce sklonowany DNA , który mo
ż
na pó
ź
niej poddawa
ć
ró
ż
nym manipulacjom przy
u
ż
yciu odpowiednich enzymów restrykcyjnych.
Badanie polimorfizmu miejsc restrykcyjnych
(RFLP).
RFLP

Ligazy DNA
katalizują formowanie
wiązań fosfodiestrowych pomiędzy
końcem hydroksylowym 3` a końcem
fosforowym 5` DNA. Pozwala to na
reperowanie jednoniciowych przerw w
dupleksie DNA , łączenie fragmentów
restrykcyjnych posiadających
homologiczne lepkie czy też nawet tępe
końce.
Wyszukiwarka
Podobne podstrony:
enzymy restrykcyjne
Enzymy restrykcyjne referat
ENZYMY RESTRYKCYJNe
1001562-enzymy restrykcyjne, semestr IV, genetyka, Genetyka
Inżynieria genetyczna enzymy restrykcyjne
enzymy restrykcyjne-stud, Studia, Inżynieria genetyczna
enzymy restrykcyjne-stud, Biologia molekularna
Enzymy restrykcyjne
1 Trawienie wektora enzymy restrykcyjne
enzymy restrykcyjne
Enzymy restrykcyjne i reakcja łańcuchowa polimerazy(PCR) zastosowanie (2)
enzymy restrykcyjne
Wykład 1 Enzymy restrykcyjne
enzymy
pros 4 Enzymy 1
inhibicja enzymy wykresy
ENZYMY prezentacja biochemia
Enzymy
więcej podobnych podstron