Konspekt wykładu 11
Wykład 11
Genetyka bakterii
Genom to całkowity zestaw genów danego organizmu lub wirusa. Genom prokariotyczny składa się najczęściej z jednego chromosomu i plazmidów. Chromosom koduje geny metabolizmu podstawowego (ang. housekeeping genes), tzn. takie, które są niezbędne w metabolizmie komórki we wszystkich warunkach wzrostu, zaś plazmidy mogą nieść geny kodujące pewne cechy specjalne, które są przydatne bakterii tylko w pewnych określonych warunkach środowiska.
Replikon - cząsteczka DNA, która ma system replikacyjny umożliwiający jej autonomiczną replikację w odpowiedniej komórce.
Chromosom prokariotyczny najczęściej - kolista cząsteczka dsDNA i nie oddzielona od reszty składników cytoplazmy żadną błoną. Tworzy on zwartą masę, którą można uwidocznić i która nosi nazwę nukleoidu. Wyjątki: dwie bakterie (należące do taksonu Planctomyces/Pirellula), Pirellula sp., która ma pojedynczą błonę otaczającą materiał genetyczny, a drugą Gemmata obscuriglobus, która ma nukleoid otoczony podwójną błoną, podobnie jak to ma miejsce u eukariotów.
Chromosomy liniowe -. Borrelia burgdorferi i promieniowce z rodzaju Streptomyces.
Istnieją jednak bakterie, które mają kilka chromosomów, np. Rhodobacter sphaeroides, ma dwa chromosomy (3,1 Mb i 0,90 Mb) i 5 różnych plazmidów.
Podstawowe pojęcia genetyczne
Genotyp to suma informacji genetycznej zawarta w genomie. Genotyp nie determinuje bezpośrednio określonego fenotypu, ale pewien zakres fenotypowych możliwości.
Fenotyp to dające się stwierdzić strukturalne lub funkcjonalne właściwości ukształtowane w wyniku współdziałania genotypu danego organizmu i czynników środowiska.
Mutacja to dziedziczna zmiana materiału genetycznego, która nie jest wynikiem segregacji ani rekombinacji genetycznej. Polega ona na zmianie chemicznej lub fizycznej budowy DNA lub RNA. Istnieją mutacje spontaniczne, które pojawiają się samoistnie i mutacje indukowane, które są celowo indukowane za pomocą mutagenów. Czynnik indukujący powstawanie mutacji to mutagen, a proces powstawania mutacji to mutageneza.
Nie wszystkie zmiany genotypu znajdują odbicie w fenotypie, istnieją mutacje kryptyczne, które nie powodują żadnych obserwowalnych zmian, ani morfologicznych, ani fizjologicznych.
Główną przyczyną mutacji spontanicznych są pomyłki polimeraz DNA. Polimerazy DNA to enzymy, które syntetyzują DNA na matrycy DNA. W komórce E. coli występują główną rolę w replokacji DNA odgrywa polimeraz I i III.
REPLIKACJA DNA
W chromosomie, np. E. coli, znajduje się miejsce zwane origin replikacji (ori), w którym rozpoczyna się jego replikacja. Miejsce to składa się z około 300 zasad, które są rozpoznawane przez specyficzne białka inicjujące replikację.
1) Cząsteczki białka DnaA wiążą się ze specyficznymi miejscami w obrębie ori, indukując miejscowe rozdzielenie nici DNA.
2) Z origin replikacji wiąże się też helikaza, która wykorzystując energię z hydrolizy ATP, rozwija podwójną helisę DNA i posuwa się wzdłuż niej tworząc widełki replikacyjne.
3) Replikacja chromosomu E. coli jest dwukierunkowa, więc powstają dwie pary widełek replikacyjnych.
4) Rozdzielone nici przyłączają białka SSB (ang. single-strand binding), które stabilizują stan częściowej denaturacji, zapobiegając tworzeniu się wiązań wodorowych. W wyniku tego powstaje forma DNA zwana strukturą theta
5) Na obu niciach powstaje starter - odcinek RNA zawierający wolną grupę 3'-OH, syntetyzowany przez prymazę RNA.
6) Kompleks helikazy z prymazą zwany jest prymosomem.
7) Przy replikacji nici ciągłej starter powstaje tylko w miejscu origin. Natomiast w przypadku nici opóźnionej synteza startera poprzedza syntezę każdego kolejnego fragmentu Okazaki.
8) Po powstaniu startera polimeraza DNA III dołącza nukleotydy do jego końca 3'-OH.
9) Po spełnieniu swojej funkcji starter jest usuwany, a na jego miejsce zostaje zsyntetyzowany odpowiedni kawałek DNA.
10) Reakcje te przeprowadza polimeraza DNA I, która ma aktywność polimerazy i egzonukleazy 5'→3'. Następnie odłącza się ona od DNA;
11) wiązanie fosfodiestrowe wprowadza ligaza DNA.
12) Poruszające się w przeciwne strony widełki replikacyjne spotkają się w miejscu ter położonym o 180o od miejsca oriC i z udziałem białka Tus, które jest antyhelikazą.
Replisom to kompleks zawierający:
dimer polimerazy DNA III
prymosom składający się z :
helikazy (6 ułożonych w pierścień białek DnaB);
prymazy syntetyzującej starter RNA.
Polimeraza DNA III, skomplikowany enzym złożony z wielu podjednostek, zdolna jest do naprawy własnych błędów. W tym celu wykorzystuje aktywność egzonukleazy 3'→5','która wycina wadliwie wstawiony nukleotyd, zastępując go właściwym, co zapewnia większą wierność replikacji. Aktywność korektorską ma też polimeraza I, ale enzym ten popełnia więcej błędów.
Częstość pomyłek wynosi:
- dla polimerazy I - 1 błąd na 5 x 107 zasad;
- dla polimerazy III - 1 błąd na 5 x 109 zasad.
Błędy powstające w czasie replikacji DNA i prowadzące do mutacji są dość rzadkie.
MUTACJE
Istnieją różnego rodzaju czynniki chemiczne, fizyczne i biologiczne, które indukują mutacje. Do mutagenów należą:
analogi zasad, które zostają włączone do DNA zamiast prawidłowych zasad,
2) związki chemiczne oddziałujące z DNA, takie jak HNO2, hydroksyloamina, akrydyny, bromek etydyny i inne;
3) promieniowanie UV i jonizujące.
4) elementy transpozycyjne.
Większość bakterii ma różnego typu mechanizmy naprawy uszkodzeń. Systemy te mogą być konstytutywne, a więc zawsze aktywne, lub mogą być indukowane przez uszkodzenia DNA.
Systemy naprawy DNA
1) naprawa przez bezpośrednie usunięcie błędu;
np., fotoreaktywacja usuwająca dimery tyminy powstałe w wyniku działania UV;
2) naprawa przez wycinanie; wycięcie błędu niekiedy wraz z krótkim odcinkiem przylegającego DNA, np. dimerów tyminy
3) naprawa rekombinacyjna, zależna od białka RecA, działa przy silnym uszkodzeniu DNA; (np. Deinococcus radiodurans ma bardzo skuteczny mechanizm naprawy rekombinacyjnej, dzięki której może przeżywać dawki promieniowania ultrafioletowego i jonizacyjnego, zabijające inne organizmy).
System SOS
- jest indukowany silnymi uszkodzeniami DNA
- składa się z:
(a) kilkunastu operonów kodujących w większości białka związane z ww. systemami naprawy;
(b) systemu naprawy błędnej, który usuwa luki i pęknięcia w DNA, aby nie doszło do zatrzymania replikacji (zmiany takie są letalne).
Niektóre z tych mechanizmów naprawy popełniają błędy i to sam proces naprawy wprowadza mutacje. Ale lepiej by komórka błędnie naprawiła swój DNA, niżby miała zginąć.
Zmienność bakterii wynika z:
1) mutacji, które powstają w wyniku:
- błędów polimeraz;
- działania mutagenów;
- działania niektórych mechanizmów naprawy;
2) pobierania przez bakterie obcego DNA, który do komórki może się dostać w wyniku 3 różnych procesów:
a) transformacji;
b) transdukcji;
c) koniugacji;
3) transpozycji.
Test Amesa
Wiele związków rakotwórczych dla człowieka ma działa mutagennie na bakterie.
W teście wykorzystuje się szczep auksotroficzny Salmonella enterica, wymagający do wzrostu histydyny. Gęstą zawiesinę tego szczepu wysiewa się na płytkę z podłożem nie zawierającym histydyny
Na związek badany działa się preparatem enzymatycznym z wątroby szczurzej (niektóre związki stają się rakotwórcze dopiero w wyniku ich przekształcania przez niektóre enzymy wątroby, np. oksygenazy).
Tak potraktowanym zawiązkiem badanym nasącza się krążek bibuły i umieszcza na płytce z bakteriami.
Jeżeli związek ma działanie mutagenne, to liczba kolonii mutantów powrotnych (rewertantów His+) zdolnych do syntezy histydyny wzrośnie.
Zmienność bakterii wynika z:
1) mutacji, które powstają w wyniku:
- błędów polimeraz;
- działania mutagenów;
- działania niektórych mechanizmów naprawy;
pobierania przez bakterie obcego DNA, który do komórki może się dostać w wyniku 3 różnych procesów:
a) transformacji;
b) transdukcji;
c) koniugacji;
3) transpozycji.
Jeśli obcy DNA, pobrany przez komórkę w wyniku transformacji, transdukcji lub koniugacji nie jest replikonem, to musi zajść następnie proces rekombinacji, aby komórka mogła go stabilnie odziedziczyć. Jeżeli nie dojdzie do rekombinacji, fragment zostanie utracony, gdyż nie jest zdolny do replikowania się.
REKOMBINACJA
Proces, w którym następuje przerwanie ciągłości jednej lub obu nici cząsteczki DNA i połączenie z jedno- lub dwuniciowym fragmentem innekj cząsteczki DNA. Główny jej mechanizm to pęknięcie i ponowne połączenie (ang. breakage and rejoining).
Za względu na charakter cząsteczek, które uczestniczą w rekombinacji wyróżnia się u bakterii:
rekombinację homologiczną (zwaną także ogólną) tj. zachodzącą między cząsteczkami o komplementarnych sekwencjach nukleotydowych;
rekombinację nieuprawnioną, gdy rekombinujące cząsteczki wykazują:
homologię ograniczoną do bardzo krótkich odcinków - rekombinacja specyficzna wobec miejsca (rekombinacja zlokalizowana) (ang. site specific recombination); do jej zajścia konieczne są specyficzne enzymy rozpoznające te sekwencje homologiczne w obu oddziałujących cząsteczkach DNA.
kompletny brak homologii - transpozycja zachodząca z udziałem transpozaz.
Rekombinacja ogólna (homologiczna) zachodzi między homologicznymi odcinkami DNA, a więc takimi, w których sekwencje nukleotydów są bardzo podobne. Jest ona realizowana poprzez pęknięcie nici DNA i połączenie ich w nowych kombinacjach. Minimalny region homologii odcinków DNA wymagany do efektywnego zajścia krzyżowej wymiany fragmentów nici (ang. crossover) wynosi 50 do 100 bp. W wyniku rekombinacji informacja genetyczna pochodząca z innej, lecz pokrewnej komórki, zostaje na trwale włączona do chromosomu biorcy, w wyniku czego powstaje rekombinant genetyczny. W rekombinacji homologicznej biorą udział bardzo liczne układy enzymatyczne przeprowadzające wymianę odcinków między cząsteczkami DNA. U bakterii proces ten zależy od genu recA. Białko RecA wiąże się z jednoniciowym DNA nadając mu strukturę helikalną, co ułatwia rekombinację.
Warunkiem bezwzględnie koniecznym do zajścia rekombinacji homologicznej jest wytworzenie u haploidalnych bakterii stanu, przynajmniej częściowej, diploidalności.
Rekombinacja zlokalizowana
wymaga obecności krótkich specyficznych sekwencji homologicznych występujących w 2 różnych cząsteczkach DNA (np. w chromosomie E. coli występuje miejsce attB, zaś w genomie faga λ homologiczne miejsce attB - ścisła homologia obejmuje 17 nukleotydów);
Oprócz tych specyficznych miejsc homologicznych wymagana jest obecność określonego enzymu typu topoizomerazy (a więc jednocześnie aktywność nukleazy i ligazy). W przypadku faga λ jest to integraza fagowa, która katalizuje włączanie genomu faga w chromosom E. coli.
TRANSFORMACJA
przenoszenie informacji genetycznej w postaci wolnego DNA z jednej bakterii do drugiej.
proces wrażliwy na DNazy (w przeciwieństwie do koniugacji i transdukcji);
materiał przenoszony: dwuniciowy DNA chromosomowy, plazmidowy i fagowy (transfekcja);
4) do transformacji naturalnej zdolne są niektóre archeony oraz niektóre bakterie, zarówno gramdodatnie (np. Bacillus, Streptococcus), jak i gramujemne (np. Azotobacter, Haemophilus, Helicobacter, Campylobacter, Neisseria).
- osiągają one tzw. stan kompetencji, który niezbędny jest do pobrania DNA.
- uzyskiwanie stanu kompetencji jest zdeterminowana genetycznie.
Kompetencja naturalna to odpowiedni stan fizjologiczny związany z ekspresją genów kodujących specjalne białka biorące udział w pobieraniu i procesowaniu DNA. W ich skład mogą wchodzić tzw. czynniki kompetencji:
białka biorące udział w modyfikacji osłon (np. autolizyny ściany komórkowej);
różne nukleazy (endonukleazy i egzonukleazy);
białka wiążące DNA.
Zwykle stan kompetencji wiąże się z określoną fazą wzrostu (np. początek fazy stacjonarnej u Streptococcus) lub gwałtowną zmianą warunków (np. u Haemophilus przeniesienie z podłoża bogatego do podłoża minimalnego) i jest zależny od składu podłoża. Zwykle stanowi temu towarzyszy spadek lub zahamowanie syntezy DNA. Mechanizm kompetencji nie jest całkowicie zrozumiały.
Etapy transformacji (ogólny schemat):
Związanie dwuniciowego DNA z komórkami kompetentnymi; (komórki kompetentne wiążą 1000 razy więcej DNA niż komórki niekompetentne).
(etap ten jest najpierw odwracalny - można usunąć DNA przez wymycie - i podatny na trawienie DNazami);
Uzyskanie oporności na DNazy;
Pobranie DNA i jego procesowanie;
Przeniesienie pobranego DNA w kierunku chromosomu;
Włączenie w wyniku rekombinacji do DNA biorcy.
Transformacja została po raz pierwszy odkryta w gramdodatnim S. pneumoniae (Griffith, 1928), potem w gramujemnym H. influenzae i wreszcie w B. subtilis i w tych trzech bakteriach proces ten został najlepiej poznany. Początkowe etapy transformacji są całkowicie różne u bakterii gramdodatnich i gramujemnych.
Bakterie gramdodatnie: Bacillus subtilis
- optymalna kompetencja - faza stacjonarna, podłoże minimalne (około 20% komórek jest w stanie kompetencji i zachowuje go przez kilka godzin);
- na powierzchni komórek pojawia się białkowy kompleks kompetencyjny, z którym dwuniciowy DNA wiąże się najpierw odwracalnie, potem nieodwracalnie;
- endonukleaza wchodząca w skład kompleksu procesuje DNA na kawałki około 15 kb, które wiążą się z błoną cytoplazmatyczną;
- w dalszym etapie procesowania DNA, w wyniku działania swoistej egzonukleazy występującej w błonie cytoplazmatycznej, następuje degradacja jednej z nici, a druga jest pobierana do cytoplazmy;
- pobierany ssDNA jest pokrywany w cytoplazmie białkami, które chronią go przed nukleazami.
- potem następuje rekombinacja z chromosomem z udziałem białka RecA.
Transformacja zachodzi w środowisku naturalnym, o czym świadczy:
występowanie stanu naturalnej kompetencji, który pozwala pobrać DNA ze środowiska zewnętrznego (i ewentualnie uzyskać pewne cechy korzystne od genetycznie odmiennych organizmów),
U bakterii gramujemnych, np. Haemophilus influenzae, pobierany jest wyłącznie DNA zawierający 11-nukleotydowe sekwencje (AAGTGCGGTCA), które występują z nieoczekiwanie dużą częstością w DNA chromosomalnym Haemophilus (~600 takich sekwencji).
Transformacja sztuczna
Metoda chemiczna
1) inkubacja komórek w lodowatym roztworze Ca2+;
2) dodanie do nich DNA i przetrzymywaniu całości w niskiej temperaturze;
3) krótki szok cieplny (37 do 42oC przez 30 - 120 sek.);
4) inkubacja w podłożu w temperaturze optymalnej;
5) wysiew na płytki selekcyjne.
Po optymalizacji warunków dla konkretnej bakterii można uzyskać od 106 do 109 transformantów na 1 g ccc plazmidowego DNA.
W jaki sposób powstaje stan „kompetencji” sztucznej?
Jedna z hipotez mówi, że pod wpływem Ca2+ następują zmiany w ułożeniu LPS i białek błony zewnętrznej, a w niskiej temperaturze następuje usztywnienie tej zmienionej struktury. Umożliwia to oddziaływanie komórki z DNA. Wydaje się, że kationy dwuwartościowe umożliwiają tworzenie względnie stabilnych kompleksów asocjacyjnych między błona zewnętrzna i DNA.
Szok cieplny powoduje upłynnienie tej quasi krystalicznej struktury osłon i wprowadzenie DNA do komórek. Niektórzy uważają, że naruszenie integralności i organizacji LPS prowadzi do odkrycia lub aktywacji kanałów, przez które może zajść pobranie DNA.
Elektrotransformacja
1) odpłukanie komórek w niskiej temperaturze i zawieszenie w H2O dejonizowanej;
2) dodanie DNA, inkubacja w 4oC;
3) zastosowanie krótkiego (2-20 msek) impulsu prądu elektrycznego o wysokim natężeniu;
- destabilizacja błon i zwiększenie ich przepuszczalności,
- pobranie DNA
- przy dobrze dobranych parametrach można uzyskać nawet 6 x 1010 transformantów na 1 μg DNA (przy małych plazmidach).
4) inkubacja w podłożu w temperaturze optymalnej.
wysiew na płytki selekcyjne.
TRANSDUKCJA (Zinder i Lederberg, 1952)
przenoszenie informacji genetycznej z jednej bakterii do drugiej za pośrednictwem wirulentnych lub umiarkowanych fagów;
ilość przenoszonego materiału genetycznego jest ograniczona pojemnością główek fagowych;
tzw. cząstki transdukujące, a więc kapsydy fagowe zawierające DNA bakterii, powstają w komórkach dawcy w czasie rozwoju faga;
w transdukcji ograniczonej cząstki transdukujące zawierają zarówno DNA wirusa jak i gospodarza w postaci pojedynczej cząsteczki DNA;
w transdukcji ogólnej cząstka transdukująca zawiera DNA bakteryjny,
Z transdukcją ograniczoną mamy do czynienia w przypadku fagów lizogennych, włączających się do chromosomu bakteryjnego. Proces ten zachodzi, gdy odłączający się od chromosomu bakteryjnego DNA fagowy, w wyniku błędnego wycięcia, zabierze ze sobą mały, sąsiadujący z nim fragment DNA bakteryjnego, zostawiając kawałek swojego genomu w chromosomie gospodarza. Takie hybrydowe cząsteczki ulegają replikacji i są pakowane w główki fagowe. Wszystkie fagi uwalniające się z takiej komórki mają w swoim genomie taki sam kawałek chromosomu gospodarza i pozbawione są takiego samego kawałka własnego genomu. Zakres cech gospodarza, które mogą być w ten sposób transdukowane jest ograniczony do tych, które są kodowane przez geny przylegające do profaga.
Na przykład np. fag lambda włącza się do chromosomu E. coli między geny gal i bio i właśnie te geny mają szansę być przeniesione przez faga w drodze transdukcji. Takie zdarzenie zachodzi bardzo rzadko.
W transdukcji ogólnej (niespecyficznej) cząstka transdukująca w ogóle nie zawierają DNA wirusowego, lecz jedynie DNA gospodarza. Cząstki transdukujące powstają, gdy fragmenty DNA gospodarza (o wielkości zbliżonej do wielkości DNA faga) są omyłkowo pakowane do główek fagowych zamiast DNA fagowego. Dzieje się to z częstością 10-4-10-7 na cząstkę faga w zależności od faga. Każdy gen bakteryjny może być przeniesiony do komórki biorcy. Do przeprowadzania transdukcji ogólnej zdolny jest bakteriofag P1.
Bakteriofag P1 - fag umiarkowany :
ikosaedralna (dwudziestościan) główka i znacznie od niej dłuższy, sztywny ogonek, płytka podstawowa i sześć skręconych włókienek.
Dwuniciowy, liniowy DNA charakteryzujący się nadmiarem terminalnym;
Główka może pomieścić 100 kb DNA.
Cl lityczny i cykl lizogenny, w którym profag istnieje w cytoplazmie gospodarza w postaci kolistego plazmidu (1 lub 2 kopii). Cząstki transdukujące powstają w czasie cyklu litycznego faga P1.
Szlak lityczny faga P1
1) Receptor - lipopolisacharyd;
2) wstrzyknięty dwuniciowy DNA o wielkości 100 kb.
3) liniowa forma z nadmiarem terminalnym przekształca się w kolistą formę replikacyjną;
4) kilka rund replikacji dwukierunkowej wg modelu theta;
5) potem replikacja wg modelu toczącego się koła i powstają długie konkatemery dwuniciowego DNA;
7) transkrypcja późnych genów - powstają białka budujące kapsydy fagowe, białka uczestniczące w dojrzewaniu cząstek fagowych i lizie komórki gospodarza.
8) z długich konkatemerów DNA odmierzane są kawałki mieszczące się w główkach fagowych i pakowane do nich;
9) nadmiar DNA jest odcinany i pakowany do następnej główki, itd.
10) cykl lityczny - 60 min i daje 100-200 cząstek fagowych na bakterię,
11) liza komórki gospodarza z udziałem fagowego lizozymu
Cząstki transdukujące mogą powstawać, jeśli:
1) nukleazy faga nie degradują zbytnio chromosomu bakteryjnego (tak jest w przypadku faga P1, natomiast np. fag T4 degraduje DNA gospodarza;
2) Mechanizm pakowania DNA w kapsydy pozwala na omyłkowe zapakowanie DNA bakteryjnego, zamiast fagowego.
Cząstki transdukujące zawierają taki kawałek DNA bakteryjnego, jaki może pomieścić główka. W przypadku faga P1 cząstki transdukujące powstają, gdy mechanizm pakujący faga odcina pomyłkowo DNA gospodarza zamiast konkatemerów DNA fagowego. W lizacie fagowym cząstki transdukujące występują z częstością 0,3%.
Powstanie stabilnych transduktantów zależy od zastąpienia kawałka DNA biorcy przez transdukowany DNA, co jest zależne od systemu rekombinacji homologicznej biorcy.
Rola transdukcji w naturze
Fagi przeprowadzające transdukcję ogólną znane są dla przeszło 20 różnych rodzajów bakterii, ale często mają one wąski zakres gospodarza, więc może wydawać się, że transdukcja ma mniejsze znaczenie w transferze genów niż koniugacja. W środowisku naturalnym fagi mogą występować w znaczących ilościach:
a) w wodzach słodkich - 103 - 104 pfu/ml;
b) w wodzie morskiej - 105 - 1011 pfu/ml;
c) w ściekach - 108 - 1010 pfu/ml.
Wykazano, że transdukcja zachodzi w glebie, wodzie słodkiej, ściekach. I choć często do infekcji fagiem dochodzi w wyniku swoistego oddziaływania białek faga ze swoistymi receptorami na powierzchni bakterii, to istnieją liczne przykłady fagów, których zakres gospodarza przekracza bariery gatunkowe, a nawet rodzajowe. Na przykład fagi PRR1 i PRD1 infekują wszystkie bakterie gramujemne zawierające plazmid z grupy IncP-1 (który koduje receptor swoisty dla faga), a inne pośredniczą w transferze genów między gronkowcami i Enterobacteriaceae.
DNA zawarty w główkach fagowych może stabilnie przetrwać w środowisku naturalnym długie okresy czasu.
PLAZMIDY
Plazmidy - pozachromosomowe elementy genetyczne, będące autonomicznymi replikonami; występują u większości prokariotów i tylko u nielicznych eukariotów. Nie znaleziono ich u niektórych bezwzględnych patogenów, np. Rickettsia. Zwykle są to replikony znacznie mniejsze od chromosomów i na ogół koliste, choć istnieją też plazmidy liniowe, np. u Streptomyces i Borrelia.
Zawierają one:
1) system replikacji (gen rep, inicjujący replikację plazmidu i ori);
2) mechanizm/y stabilizujące:
aktywny rozdział (partycja) - zapewnia prawidłowy rozdział plazmidów do komórek potomnych.
rozdział dimerów (ang. dimer resolution); w wyniku rekombinacji homologicznej między siostrzanymi plazmidami mogą powstawać dimery plazmidów, które mogłyby zakłócić aktywny rozdział;
system trucizna-antidotum (ang. addiction system), który zabija komórki bezplazmidowe.
3) system transferu koniugacyjnego (geny tra i oriT);
4) inne geny, przydatne komórce w pewnych określonych warunkach środowiska.
Plazmidy różnią się od wirusów dwiema cechami:
1.) nie szkodzą na ogół swojemu gospodarzowi, wręcz przeciwnie mogą mu nadawać jakieś cechy dla niego korzystne;
2.) nie mają formy zewnątrzkomórkowej.
Plazmidy mogą mieć wielkość od około 1 kb do ponad 1000 kb. Największe z nich, zwane megaplazmidami, niekiedy trudno odróżnić od chromosomów. O plazmidach, które nie nadają swojemu gospodarzowi żadnych cech fenotypowych mówimy, że są kryptyczne.
Replikacja plazmidów
- u bakterii gramujemnych - wg modelu theta (dwukierunkowa lub jednokierunkowa);
- większość u bakterii gramdodatnich - wg modelu toczącego się koła;
inicjacja replikacji zachodzi z częstością, która dla danego plazmidu daje określoną, charakterystyczną liczbę kopii plazmidu, np.
- plazmid F - 1-2 kopii na komórkę,
- plazmid ColE1 - 10-30 kopii.
Plazmid F
plazmid koniugacyjny
kolista cząsteczka dwuniciowego DNA (100 kb);
1-2 kopie na komórkę.
koduje m.in. następujące cechy:
a) inicjację własnej replikacji (rep) i jej regulację;
b) zdolność do koniugacji i syntezę pili (geny tra - 30 kb);
- c) oriT - miejsce początkowe przy przenoszeniu plazmidu w czasie koniugacji.
d) IS2, IS3, transpozon Tn1000.
Plazmid F jest episomem, tzn. plazmidem, który może ulec włączeniu do chromosomu bakteryjnego. Jest to możliwe dzięki występowaniu na plazmidzie i w chromosomie sekwencji insercyjnych. W plazmidzie występuje jedna sekwencja IS2 i dwie - IS3, zaś w chromosomie jest tych sekwencji około 20. Integracja F zachodzi w wyniku rekombinacji homologicznej między dwiema takimi samymi IS, z których jedna jest w plazmidzie, a druga w chromosomie (nie jest to transpozycja!). W zależności od miejsca integracji i jej orientacji powstają różne szczepy Hfr, mające plazmid F włączony w różne miejsca chromosomu. W stanie zintegrowanym plazmid jest powielany jako część chromosomu. Jego geny tra ulegają ekspresji, więc komórka jest nadal zdolna do koniugacji.
W populacji F+ spontaniczna integracja plazmidu F do chromosomu zachodzi z częstością z częstością 10-7 (powstają komórki Hfr).
KONIUGACJA
Koniugacja to przenoszenie informacji genetycznej z jednej komórki do drugiej komórki, zachodzące podczas bezpośredniego kontaktu obu komórek. Proces ten jest uwarunkowany obecnością w komórce dawcy plazmidu koniugacyjnego, kodującego geny tra (od transfer), determinujące poszczególne etapy tego procesu Niekiedy w czasie koniugacji inne elementy genetyczne mogą ulec mobilizacji i zostają przeniesione do komórki biorcy. Mogą to być inne plazmidy lub chromosom gospodarza plazmidu.
Koniugacja u E. coli Komórki E. coli zawierające plazmid F w formie autonomicznej, określane są jako komórki F+, zaś komórki bez tego plazmidu - jako F-. Komórki, w których plazmid F jest w formie zintegrowanej z chromosomem to komórki Hfr (ang. high frequency of recombination).
Etapy koniugacji F- z F+
1) tworzenie swoistych par uwarunkowane obecności pilusów płciowych na komórkach męskich i receptorów na komórkach żeńskich;
2) tworzenie par efektywnych; pilus płciowy ulega retrakcji i obie komórki zostają przyciągnięte do siebie, a ich błony cytoplazmatyczne ulegają miejscowej fuzji;
3) w obszarze oriT (origin transferu) plazmidu F dochodzi do jednoniciowego nacięcia DNA i nić z wolnym końcem 5' jest przenoszona do komórki biorcy;
4) równocześnie z przenoszeniem pojedynczej nici DNA dawcy, następuje w biorcy synteza nici komplementarnej, a także synteza uzupełniająca w komórce dawcy, co powoduje, że komórka biorcy uzyskuje kopię plazmidu F bez utraty plazmidu przez komórkę dawcy.
5) komórka biorcy uzyskuje kopie plazmidu F, a więc staje się komórką F
Koniugacja F- z Hfr
1) Pierwsze etapy tego procesu są takie same jak te opisane powyżej, a więc powstają pary koniugacyjne;
2) Chromosom Hfr ulega jednonociowemu nacięciu w miejscu, gdzie znajduje się oriT plazmidu F, i zaczyna wnikać do komórki biorcy (końcem 5').
3) Załóżmy, że plazmid F włączył się do chromosomu między genami pro i lac. Tak więc jako pierwsze wnikają niektóre geny plazmidu F a zaraz za nimi geny chromosomowe, w tym gen pro jako pierwszy. Gdyby cały chromosom wszedł do biorcy (co jest raczej niemożliwe), to gen lac wszedłby jako ostatni gen chromnosomowy.
4) Przekazywanie chromosomu zachodzi z jednoczesną replikacją DNA.
5) Jest to proces polarny, a kolejne geny są przekazywane z mniej więcej stałą prędkością.
6) Gdy pary koniugujące rozrywają się, transfer przestaje zachodzić i biorca otrzymuje jedynie fragment DNA dawcy.
7) Największe jest prawdopodobieństwo przekazania genów znajdujących się blisko oriT;
8) Powstaje merozygota - komórka F- zawierająca kawałek chromosomu dawcy;
9) Jeżeli nastąpi proces rekombinacji homologicznej powstaną rekombinanty, które uzyskały pewne cechy chromosomalne od komórek męskich
10) Rekombinanty są nadal komórkami żeńskimi, gdyż prawie nigdy nie dochodzi do przekazania całego chromosomu z kompletnym plazmidem F.
11) Różne szczepy Hfr będą przekazywały różne geny jako pierwsze. Zależy to od miejsca włączenia plazmidu F i jego orientacji.
Plazmidy koniugacyjne mogą też mobilizować do koniugacji inne plazmidy niekoniugacyjne, pod warunkiem, że mają one oriT podobne do oriT plazmidu koniugacyjnego
Mapowanie. Za pomocą koniugacji, stosując różne szczepy Hfr, można było zbadać rozłożenie genów na chromosomie bakterii, tzn. opracowywać mapy chromosomowe.
Plazmidy koniugacyjne występują zarówno u bakterii gramujemnych jak i gramdodatnich, a także u archeonów. Niektóre z plazmidów należących do grupy niezgodności IncP mogą funkcjonować właściwie we wszystkich bakteriach gramujemnych. Są to plazmidy o szerokim zakresie gospodarza (ang. BHR, broad host range).
Biologiczna rola koniugacji to przede wszystkim przenoszenie plazmidów pomiędzy komórkami danej populacji i to nie tylko jednego gatunku, lecz również między różnymi gatunkami (transfer horyzontalny), a więc rola w ewolucji.. Plazmidy często kodują cechy selekcyjnie korzystne, np. oporność na antybiotyki, sulfonamidy i metale ciężkie, zdolność do wykorzystywania różnych źródeł węgla (w tym ksylen, toluen, kamfora, oktan), wytwarzanie kolicyn, hemolizyn, enterotoksyn itd.
Transfer T-DNA do roślin jest swego rodzaju modyfikacją bakteryjnej koniugacji.
ELEMENTY TRANSPOZYCYJNE
Elementy transpozycyjne (ang. transposable elements, TE) wykryła po raz pierwszy Barbara McClintock w latach pięćdziesiątych w kukurydzy. Występują one zarówno u eukariotów jak i prokariotów i odgrywają ważną rolę w zmienności genetycznej. Są to określone sekwencje DNA, które mogą przenosić się z jednego położenia w danej cząsteczce DNA w inne położenie w tej samej cząsteczce, bądź z jednej cząsteczki DNA do innej, przy czym miejsca te są całkowicie niespokrewnione. Ten proces przeniesienia nazywa się transpozycją, a enzym, który umożliwia ten proces (zwykle kodowany przez element transpozycyjny) - transpozazą.
Transpozycja jest rodzajem rekombinacji nieuprawnionej.
Elementy transpozycyjne występują u wszystkich bakterii i archeonów i są najprawdopodobniej odpowiedzialne za większość rearanżacji genomów prokariotycznych.
Istnieją dwa rodzaje elementów transpozycyjnych:
1) Sekwencje insercyjne, które określa się IS i kolejnym numerem, pisanym kursywą.
2) Transpozony określa się Tn i kolejnym numerem również pisanym kursywą. W pewnych przypadkach w nazwie umieszczane są dodatkowe litery, by wskazać, z jakiej bakterii pochodzi dany element (np. ISRm2 to IS z Rhizobium meliloti).
Elementy transpozycyjne mają dwie wspólne cechy:
1) kończą się odwróconymi sekwencjami powtórzonymi (ang. inverted repeat - IR).
Dwa regiony DNA można nazwać IR, jeśli sekwencja nukleotydowa jednej nici w pewnym regionie, po odczytaniu od końca 5' do 3', jest taka sama jak sekwencja 5' do 3' nici przeciwległej w innym regionie. Końcowe sekwencje IR zwykle mają 15-40 bp długości i są różne w różnych elementach transpozycyjnych. Spokrewnione elementy transpozycyjne mają podobne IR. IR funkcjonują jako miejsca rozpoznawania przez enzymy uczestniczące w transpozycji.
2) Każdy funkcjonalny element transpozycyjny niesie gen/y kodujące białko, zwane transpozazą, które jest niezbędne do transpozycji.
Sekwencje insercyjne (ang. insertion sequence - IS)
najmniejsze i najprostsze elementy transpozycyjne
wielkość od 0,7 do około 3 kb, więc nie mogą kodować więcej niż 2 białka, związane z transpozycją.
nie niosą genów kodujących cechy, które można by selekcjonować.
Wiele IS stanowi końcowe powtórzenia transpozonów.
Transpozony (ang. transposon, Tn)
elementy transpozycyjne większe od IS;
kodują też cechy nie związane z transpozycją, dzięki którym komórka zyskuje określone cechy fenotypowe, np. oporność na antybiotyk. Dzieli się na 3 klasy:
a) transpozony złożone (ang.composite transposons) - budowa modularna, tzn, po obu stronach unikatowej sekwencji centralnej (kodującej geny nie biorące udziału w transpozycji) znajdują się dwie kopie IS;
b) transpozony niezłożone (ang. non-composite transposons) - geny kodujące funkcje nie związane z transpozycją leżą obok genów związanych z transpozycją, transpozon jest oflankowany krótkimi IR, tak jak to jest u większości IS.
c) transpozony koniugacyjne (ang. conjugative transposons - CTn) mają nie tylko zdolność do transpozycji, ale także do przenoszenia się podobnie jak plazmidy koniugacyjne, z jednej komórki do drugiej (więc są b. duże). Pierwszy CTn to Tn916 wyizolowany z Enterococcus faecalis w 1995. Niesie on oporność na tetracyklinę (tetM).
Mechanizm transpozycji
Sekwencje IS są oflankowane krótkimi DR (direct repeats) - 2 - 12 bp, bowiem obie nici w sekwencji docelowej są nacinane z pewnym przesunięciem. Następnie gdy TE zostaje włączony, jednoniciowe luki po obu stronach elementu zostają wypełnione. W ten sposób powstają DR w miejscu włączenia IS do cząsteczki biorcy. Dla danego elementu wielkość DR jest zwykle stała. Jeśli więc sekwencjonując DNA, odkryjemy strukturę oflankowaną DR i IR, to prawdopodobnie jest to element transpozycyjny.
Mechanizm konserwatywny (niereplikacyjny) (ang. cut and paste) Element jest wycinany przez transpozazę z cząsteczki dawcy (jako dwuniciowy DNA) i przenoszony w nowe miejsce do cząsteczki biorcy. W rezultacie cząsteczka dawcy traci element transpozycyjny, który przechodzi do cząsteczki DNA będącej biorcą.
Mechanizm replikacyjny
W czasie transpozycji replikacyjnej, która zachodzi z udziałem 2 enzymów kodowanych przez element transpozycyjny (transpozazy i resolwazy), kopia elementu transpozycyjnego pozostaje w tym samym miejscu cząsteczki dawcy DNA, a druga kopia pojawia się w drugiej cząsteczce DNA, będącej celem transpozycji
Transpozycja jest bardzo precyzyjnie regulowana i zachodzi rzadko, w przeciwnym wypadku genom byłby usiany transpozonami, co miałoby niszczący wpływ na komórkę.
VI Znaczenie elementów transpozycyjnych
TE odgrywają dużą rolę w ewolucji genomów bakteryjnych. Na przykład:
Jeżeli miejscem insercji jest gen strukturalny, powstanie mutacja insercyjna.
Wraz z transpozonem cząsteczka docelowego DNA uzyskuje pewne cechy fenotypowe kodowane przez transpozon, np. geny oporności na antybiotyki czy geny wirulencji.
3) Między dwoma identycznymi TE może zachodzić rekombinacja homologiczna i mogą one pośredniczyć w rearanżacjach DNA gospodarza, włączając w to delecje, inwersje i duplikacje dużych fragmentów DNA.
Wyspy patogenności (ang. pathogenicity islands) to duże skupiska genów związane z wirulencją bakterii, uzyskiwane w drodze pojedynczego wydarzenia transferu horyzontalnego. (np. u enterokrwotocznej E. coli O157:H7). Niekiedy cała taka wyspa patogenności może zostać wycięta i włączona do plazmidu. Wraz z plazmidem może zostać przeniesiona do innej bakterii, gdzie może ulec integracji z jej genomem.
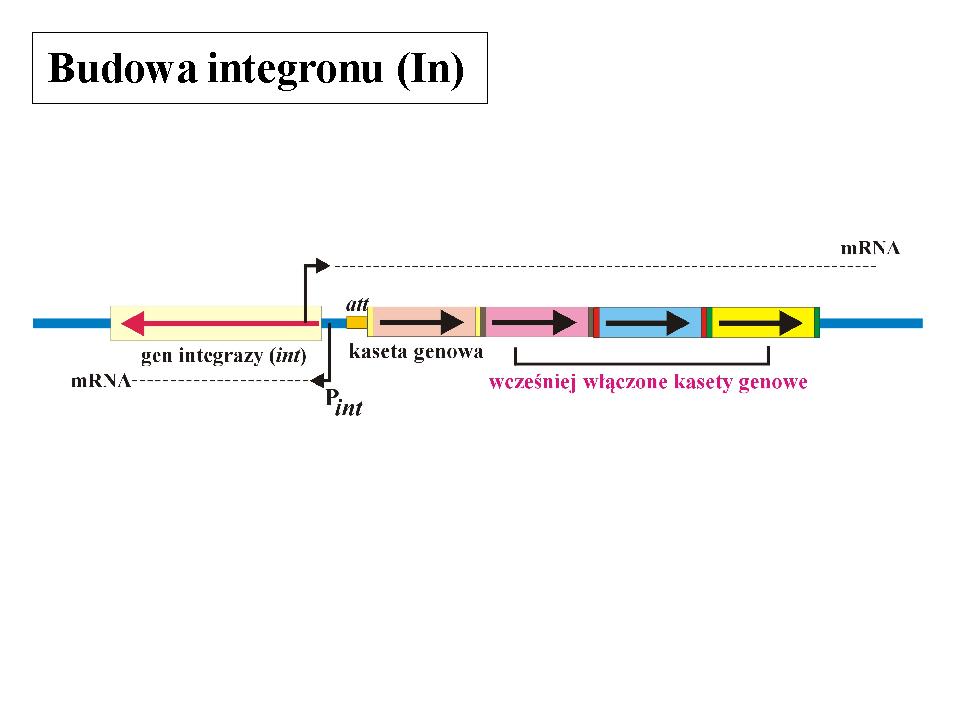
Integrony
Integronami (In) to elementy genetyczne, które mogą „wyłapywać” geny z innych cząsteczek DNA. Te wyłapane geny mogą po włączeniu do integronu ulegać ekspresji.
Integrony zawierają:
1) gen integrazy, która jest enzymem przeprowadzającym rekombinację zlokalizowaną.
2) specyficzną sekwencję DNA, w którą integraza może wstawiać kasety genowe „wyłapane” z innych miejsc;
3) promotor, dzięki któremu może zajść ekspresja genów zawartych w wyłapanych kasetach genowych.
Kasety genowe występują w różnych replikonach i są otoczone z obu stron specyficznymi sekwencjami DNA, rozpoznawanymi przez integrazę. Wydaje się, że te geny nie ulegają ekspresji, dopóki nie staną się częścią integronu, gdzie będą ulegały ekspresji z promotora zawartego w integronie.
Integrony występują w chromosomach i w plazmidach wielu różnych bakterii (np. Escherichia, Pseudomonas, Vibrio), w tym w wielu izolatach klinicznych. Niektóre z izolatów klinicznych zawierają aż 5 różnych kaset genowych. W sumie zidentyfikowano już ponad 40 różnych genów oporności na takich kasetach, a także kilka genów związanych z wirulencją.
Rekombinacja
Typ rekombinacji |
Wymaganie homologii sekwencji |
Udział białka RecA |
Udział enzymów typu topoizomeraz |
Przykład |
homologiczna (ogólna; ang. general recombination) |
+ minimalny region homologii około 50 - 100 zasad |
+ |
- |
szlak RecBCD (rekombinacja pokoniugacyjna) |
zlokalizowana (swoista dla miejsca; ang. site-specific) |
+ (homologia ograniczona) |
- |
+ |
integracja faga λ (integraza; sekwencje attP i attB) |
transpozycja |
- |
- |
+ |
przenoszenie IS i Tn z udziałem transpozaz |
Cechy kodowane przez plazmidy
Cecha kodowana przez plazmid |
Przykłady bakterii/archeonów |
Wytwarzanie antybiotyku |
Streptomyces |
Koniugacja |
Escherichia, Pseudomonas, Rhizobium, Staphylococcus, Streptococcus, Vibrio |
Funkcje fizjologiczne |
|
rozkład oktanu, naftalenu, kamfory |
Pseudomonas |
wykorzystywanie laktozy, sacharozy |
Enterobacteriaceae |
wiązanie azotu |
Rhizbium |
wytwarzanie barwnika |
Staphylococcus |
Oporność |
|
na antybiotyki |
Enterobacteriaceae, Staphylococcus, Neisseria |
metale ciężkie |
Pseudomonas, Staphylococcus |
Patogenność |
|
inwazyjność |
Salmonella, Shigella, Yersinia |
wytwarzanie toksyn |
Escherichia, Staphylococcus |
Nowotwory u roślin |
Agrobacterium |
Wytwarzanie pęcherzyków gazowych |
Halobacterium |
1
14
Wyszukiwarka
Podobne podstrony:
0 konspekt wykladu PETid 1826 Nieznany
konspekt wyklad 1, FIZJOTERAPIA (metody)
DEMOGRAFIA Konspekt wykładu 3
Konspekt wykładów z Podstaw automatyki wykład 5
IX 1 dr M K Grzegorzewska konspekt wykładu 2011
DEMOGRAFIA Konspekt wykładu 6 8
Konspekt z wykładu Krótkie wsporniki
Konspekt wykładu Geografia fizyczna Europy 08
Osoby fizyczne zdolność do czynności prawnych konspekt wykładu z 26 10 2015
konspekt wyklad 2
konspekt-z-wykladu-pediatrii-z-dn-15102007-dla-studentow
Konspekt wykładu 3, mikrobiologia
Dzieje krajoznawstwa konspekt wykladu
Konspekt z wykładu 5
Konspekt z wykładu 2
Gruntoznawstwo (W) Konspekt wykładów
Konspekt wykłady makro I czesc
Konspekt wykładów
0 Konspekt wykładuid 1720
więcej podobnych podstron