
Wewnątrz jądra komórkowego:
- enzymy replikujące muszą odnajdywać miejsca
inicjacji syntezy DNA
- czynniki transkrypcyjne i polimerazy RNA –
promotory i enhancery
- czynniki splicingowe - miejsca wycinania intronów
- mRNA – pory przez które opuści jądro.
Można założyć, że czynniki te swobodnie poruszają się
w soku jądrowym, a przypadkowe spotkania z substratami
doprowadzają do zajścia pożądanych reakcji.

Alternatywny model zakłada, że RNA jest transkrybowane na
stałej „platformie” gromadzącej wszystkie enzymy
potrzebne do transkrypcji, dojrzewania RNA i jego
transportu (Wei i in. 1998). Istnieją powody do takiego
myślenia: mitochondrialny łańcuch transportu elektronów
zachodzi w takim uporządkowanym agregacie, a u bakterii
enzymy syntetyzujące DNA skupione są na wewnętrznej
powierzchni błony komórkowej.
Pytania:
Czy w jądrze istnieje rodzaj retikulum, na którym znajdują
się te enzymy?
Jeśli istnieje, czy geny aktywne transkrypcyjnie lokują się na
nim i czy są tam enzymy syntezy RNA?
jądro komórkowe jest wysoce uporządkowaną
dynamiczną strukturą
chromosomy zajmują określone terytoria
chromosomowe
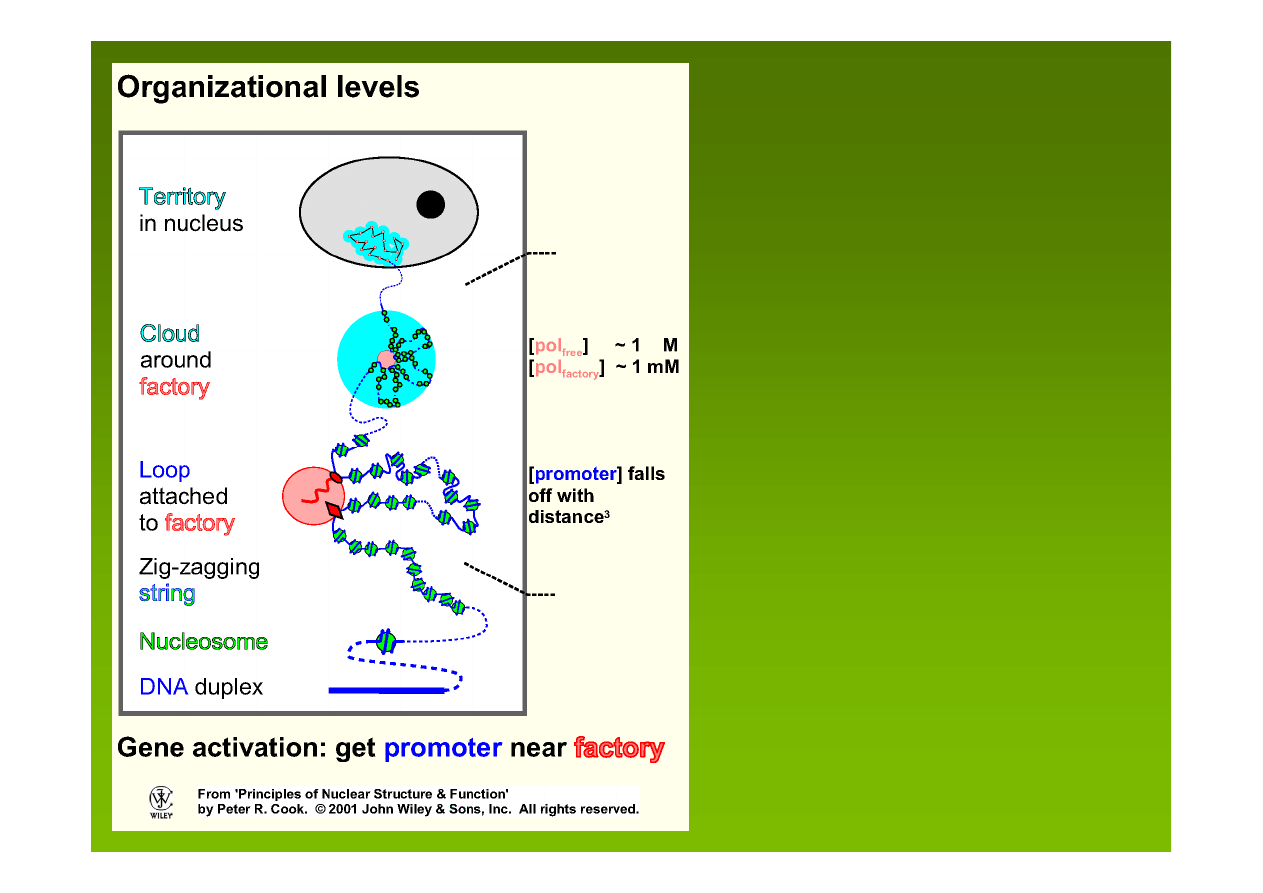
białka biorące udział w replikacji
bądź transkrypcji grupują się
razem w
jądrowe fabryki
kopiujące DNA bądź
transkrybujące geny
w przestrzeniach pomiędzy
chromosomami znajdują się
miejsca przechowywania białek
biorących udział w dojrzewaniu
RNA
dynamiczna struktura, zwana
macierzą jądrową (NM ang.
nuclear matrix), aranżuje
organizację jądrowego DNA
i rozmieszczenie fabryk
białkowych w przestrzeni jądra

NM jest strukturą widoczną albo po ekstrakcji solnej jąder
trawionych nukleazą albo elektroforetycznym usunięciu
z jąder fragmentów chromatyny w fizjologicznych
warunkach jonowych
NM jest dynamicznym strukturalnym zrębem jądra złożonym
z sieci włókien RNAP połączonych z białkami laminy
(Capco et al., 1982) pow. 47 000

włókna chromatynowe są zorganizowane w pętlowe
domeny
geny aktywne transkrypcyjnie znajdują się w pętlach
chromatyny wrażliwych na DNazę I, dostępnych dla
czynników transkrypcyjnych i maszynerii
transkrypcyjnej
geny nieaktywne są w bardziej zaplecionych obszarach
chromatynowych
u podstaw pętli znajdują się sekwencje DNA zwane
S/MARami (ang. scaffold/matrix attachment
regions), które wiążą się z białkami NM
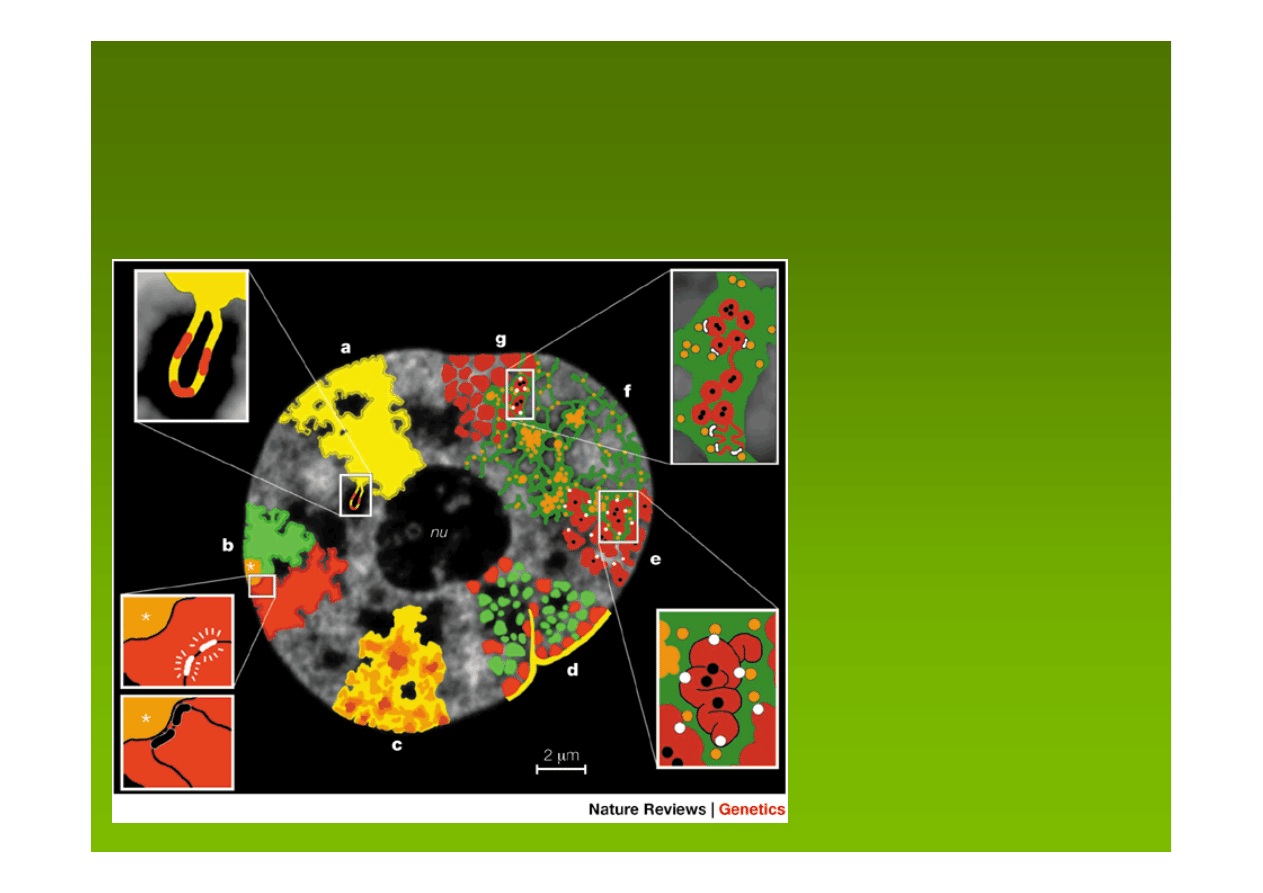
a:
CT mają kompleksowo
ufałdowane powierzchnie –
olbrzymia pętla z kilkoma
aktywnymi genami
wystaje
z powierzchni CT
do
przestrzeni IC
model funkcjonalnej architektury jądrowej zawierającej
terytoria
chromosomowe CT (ang. chromosome-territory)
i
przestrzenie
międzychromosomowe IC (ang. interchromatin compartment)
b: CT zawierają oddzielne
domeny dla ramion
p
i
q
chromosomów i
centromerów
;
geny aktywnie transkrybowane
są w pętli odległej od hetero-
chromatyny centromerowej,
a umieszczenie tych genów
w heterochromatynie powoduje
ich wyciszenie
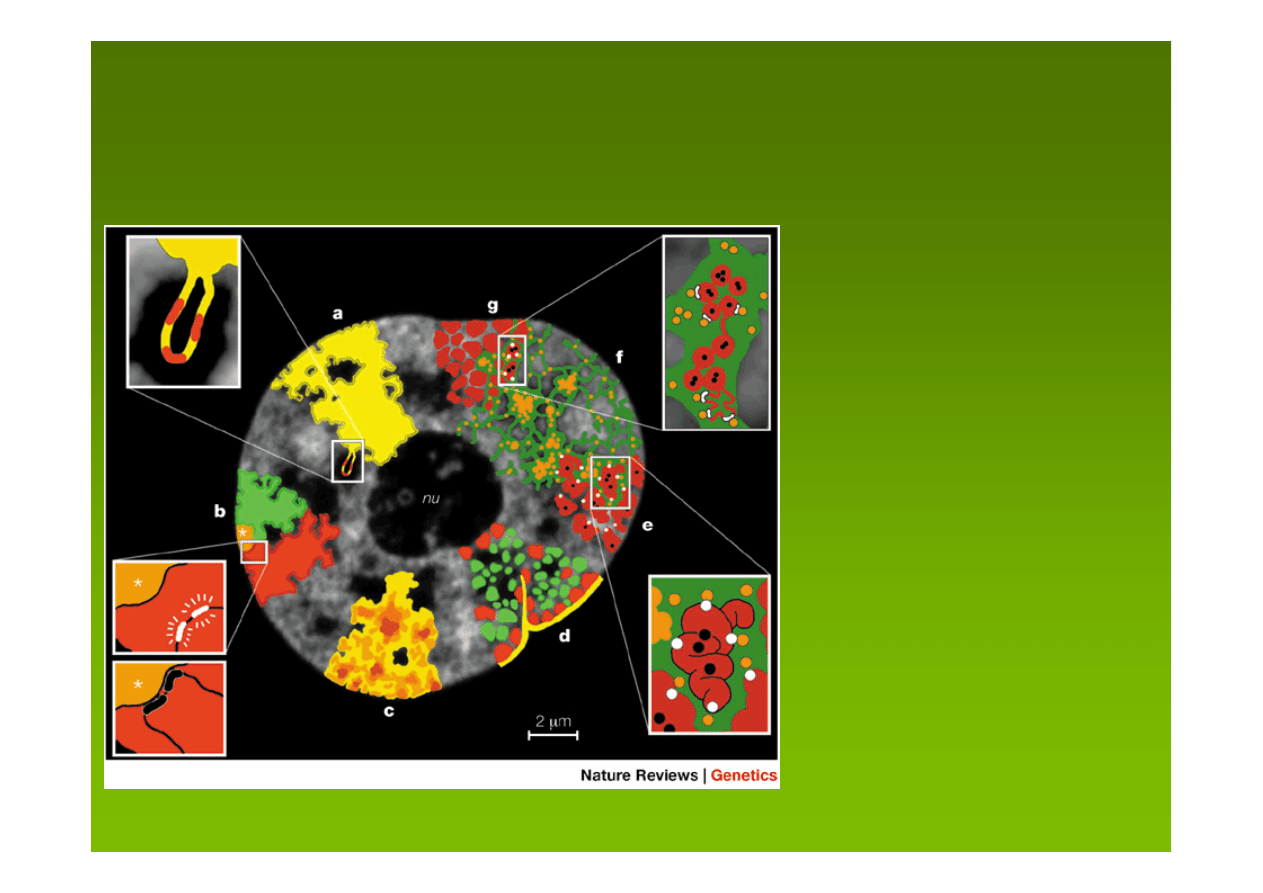
c: CT mają różną gęstość
chromatyny:
wysoką
lub
niską;
luźna
chromatyna wychodzi
do IC,
gęsta
jest z dala od IC
model funkcjonalnej architektury jądrowej zawierającej
CT
(ang. chromosome-territory) i
IC
(ang. interchromatin compartment)
e: wyższorzędową strukturę
chromatyny stanowi hierarchia
włókien chromatynowych;
geny aktywne
znajdują się na
powierzchni ufałdowanych
włókien,
geny wyciszone
mogą być
wewnątrz struktur
chromatynowych
d: CT z 1 Mb domenami
wcześnie
i
późno
replikującymi;
chromatyna
uboga w geny
lokuje się na obrzeżu jądra
blisko
laminy
, jej wpukleń
i wokół jąderka,
bogata w
geny
- lokuje się pomiędzy
przedziałami ubogimi w geny
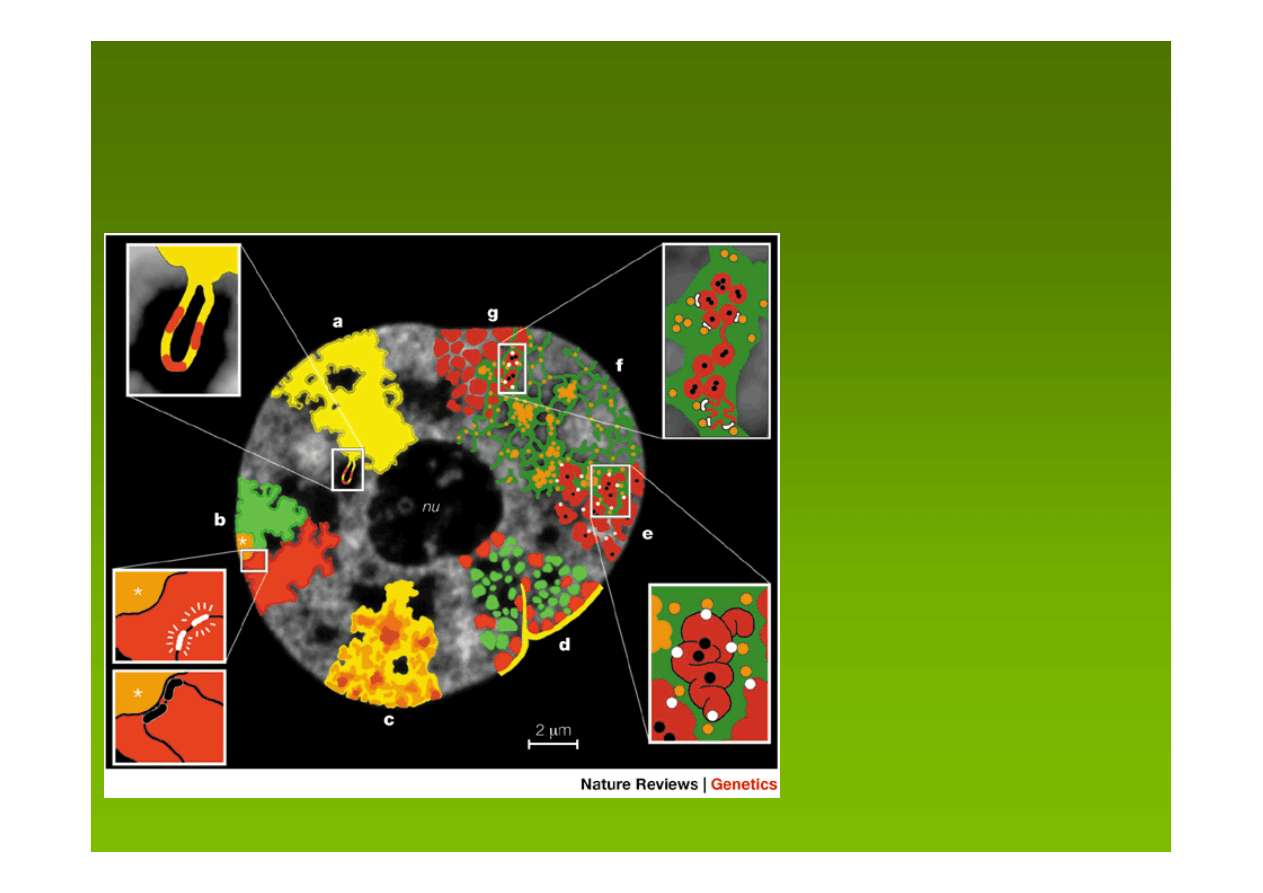
f: model zakłada, że IC
zawiera
kompleksy
(pomarań-
czowe kropki)
i większe nie-
chromatynowe domeny
(skupienia pomarańczowych
kropek)
transkrypcyjne,
splicingowe, replikacyjne i
naprawy DNA
g: CT z 1-Mb
domenami chro-
matynowymi
i IC pomiędzy nimi,
najmniejsze rozgałęzienia IC
kończą się między 100-kb
domenami chromatyny,
geny
aktywne
są na powierzchni tych
domen,
geny wyciszone
wewnątrz nich, alternatywnie
zamknięte 100-kb domeny
z wyciszonymi genami zmieniają
konfigurację na otwartą przed
aktywacją transkrypcyjną
model funkcjonalnej architektury jądrowej zawierającej
CT
(ang. chromosome-territory) i
IC
(ang. interchromatin compartment)
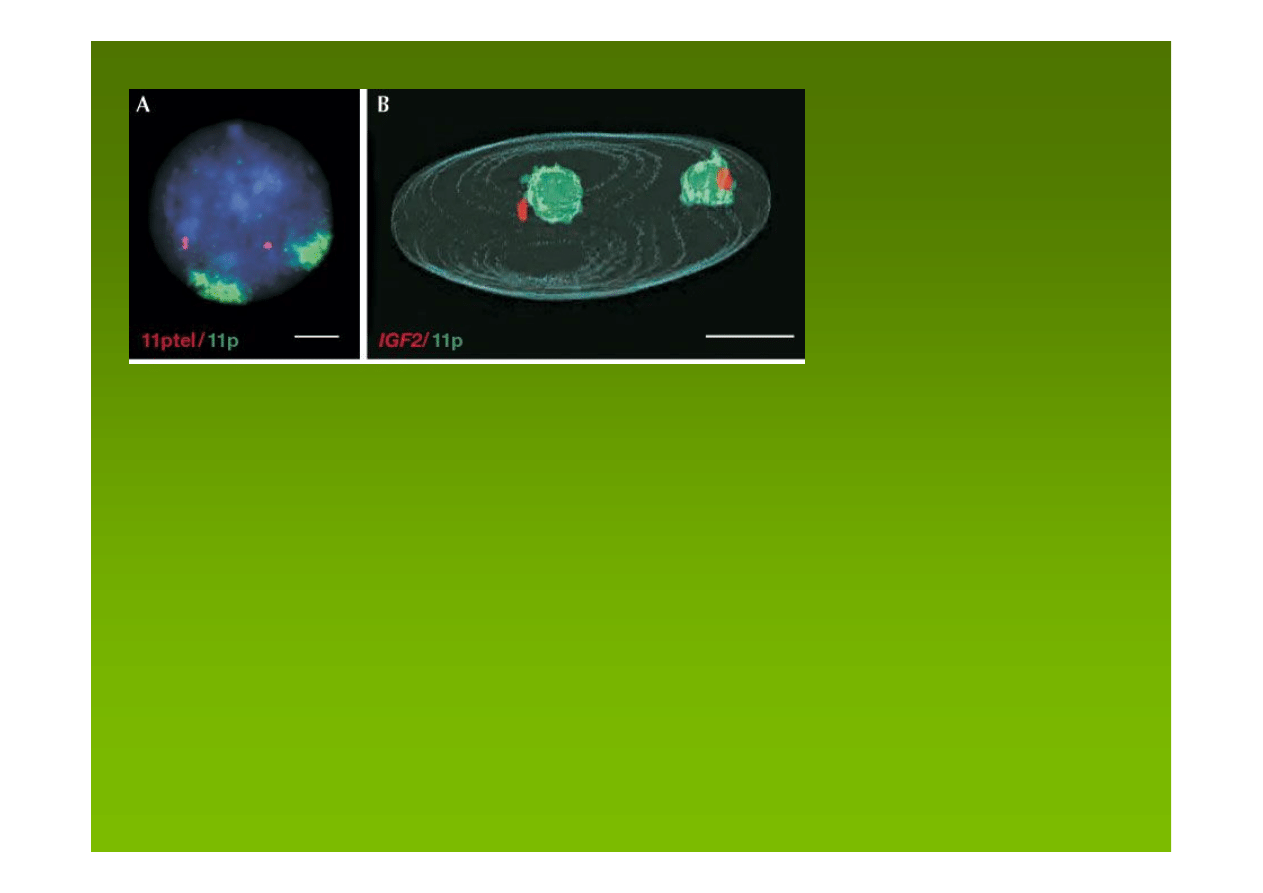
Terytoria chromosomowe
dwa
jasne terytoria 11p
leżą pod powierzchnią jądra,
użycie krótszej sondy odpowiadającej subtelomerowemu
regionowi
11ptel
zawierającemu wyłącznie aktywne geny np.
IGF2
daje sygnał poza terytorium 11p
(A) ludzkie limfocyty (B) ludzkie fibroblasty
Cremer & Cremer, 2001
Mahy i in. 2002 J Cell Biol 159: 753-763
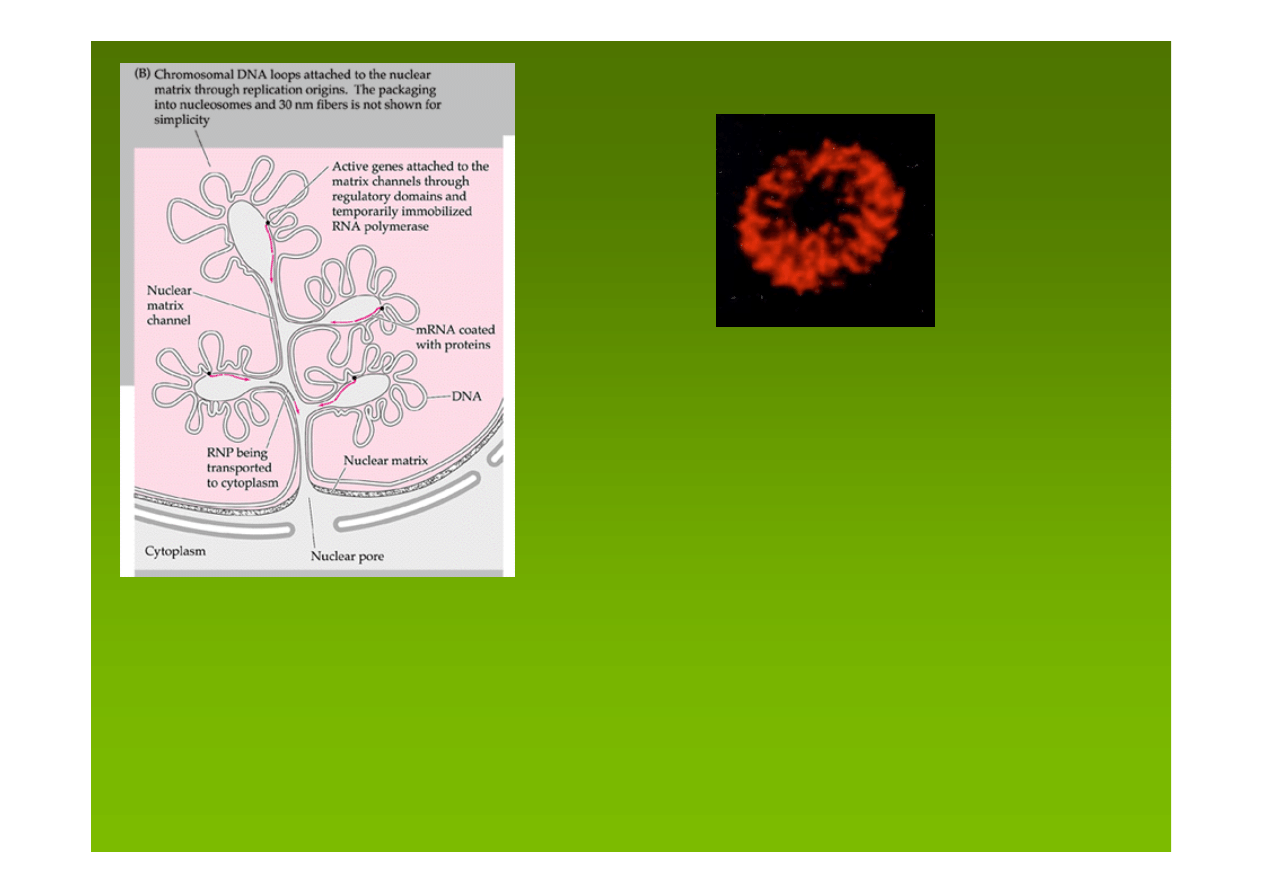
Hipotetyczny model
organizacji interfazowej
chromatyny, pokazujący NM
jako sieć wewnętrznych
kanałów
(Razin i Gromova, 1995.)
Jądra komórkowe erytrocytów potraktowano
DNazą I - wprowadza to 1-niciowe nacięcia w
regiony chromatyny zawierające aktywne geny.
Nacięcia „naprawiono” wewnątrz jądra przy
użyciu metody „nick translation” w obecności
fluorescencyjnie znakowanych nukleotydów.
Fluorescencja pojawiła się pod powierzchnią
jądra i wzdłuż struktur prowadzących od
otoczki jądrowej w głąb jądra.
(Hutchinson i Weintraub, 1985 )
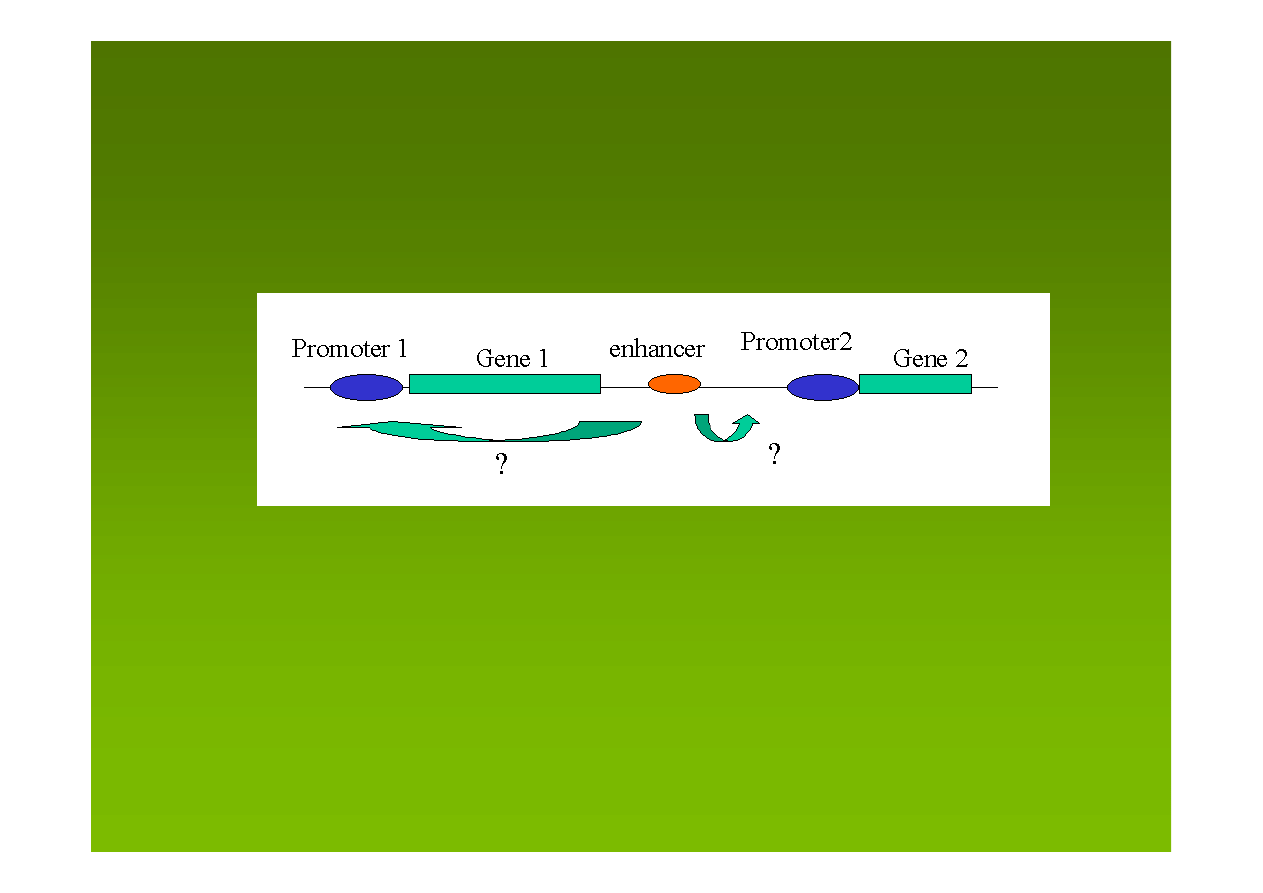
Wyższorzędowa struktura genomu
sama natura oddziaływań promotor–enhancer nie zapewnia
przestrzennej i czasowej specyficzności ekspresji genów
specyficzność ustanawia chromatyna – podział na domeny
transkrypcja ze specyficznego promotora jest wynikiem
aktywacji tylko poprzez enhancery położone w tej samej
domenie chromatynowej
http://140.116.60.1/mdlai/Handout/chromatin-domain-99/sld001.htm
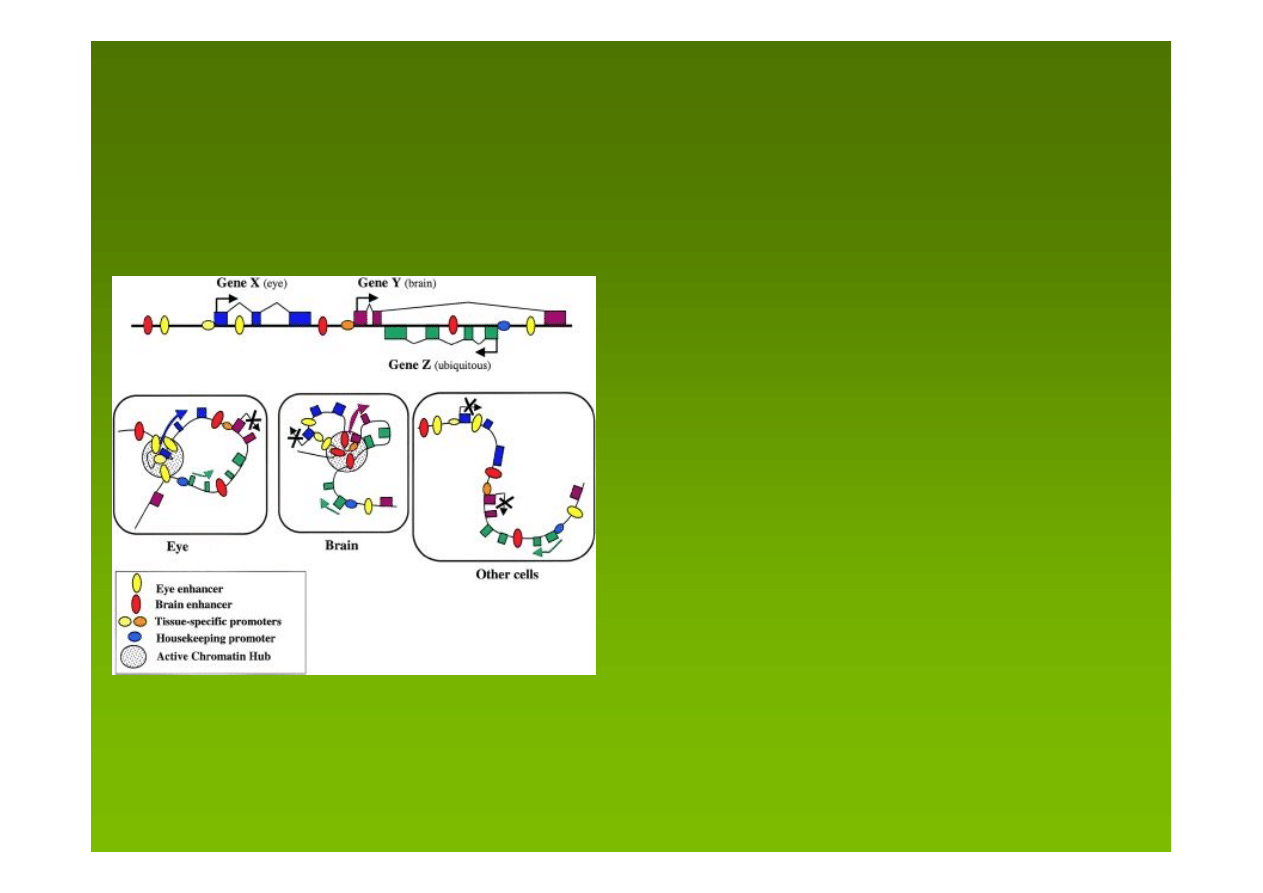
Hipotetyczny region zawierający dwa geny
tkankowo specyficzne i jeden gen
metabolizmu podstawowego (ang.
housekeeping gene). Gen X (eksony
niebieskie) ulega ekspresji w tkankach
oczu, gen Y (e. fioletowe) w mózgu, gen Z
(e. zielone) w każdej tkance. Tkankowo
specyficzna aktywność transkrypcyjna
zależy od utworzenia takiego centrum
aktywnej chromatyny ACH, która obejmuje
tkankowo specyficzne elementy
cis ze
związanymi kompleksami czynników i
umożliwia wybiórczą interakcję z
promotorem danego genu. Utworzenie ACH
zapewnia lokalne wysokie nagromadzenie
czynników transkrypcyjnych i czynników
pozytywnie modelujących chromatynę.
Aktywność genu metabolizmu
podstawowego nie wymaga tworzenia ACH.
Kleinjan DA, van Heyningen V 2005 Am J Hum Genet 76: 8–32.
Model współistnienia w tym samym rejonie genomu
fizycznie zachodzących, ale niezależnie regulowanych genów

Domeny chromatynowe i elementy graniczne
transformacja egzogennego DNA do komórek linii hodowlanych
lub transgeniczne zwierzęta służą do identyfikacji elementów
cis
, które zapewniają aktywację transkrypcyjną niezależnie od
miejsca integracji w genomie
zidentyfikowano 2 typy takich sekwencji:
LCR
y (ang. Locus Control Region) definiują aktywne domeny
ekspresji genowej w sposób dominujący i są wymagane do
tkankowo specyficznie regulowanej transkrypcji wielu
genów u kręgowców
elementy graniczne/izolatory
(ang. insulator) to sekwencje,
które zapewniają transkrypcję niezależną od pozycji
poprzez izolowanie ekspresji genu od sekwencji sąsiednich
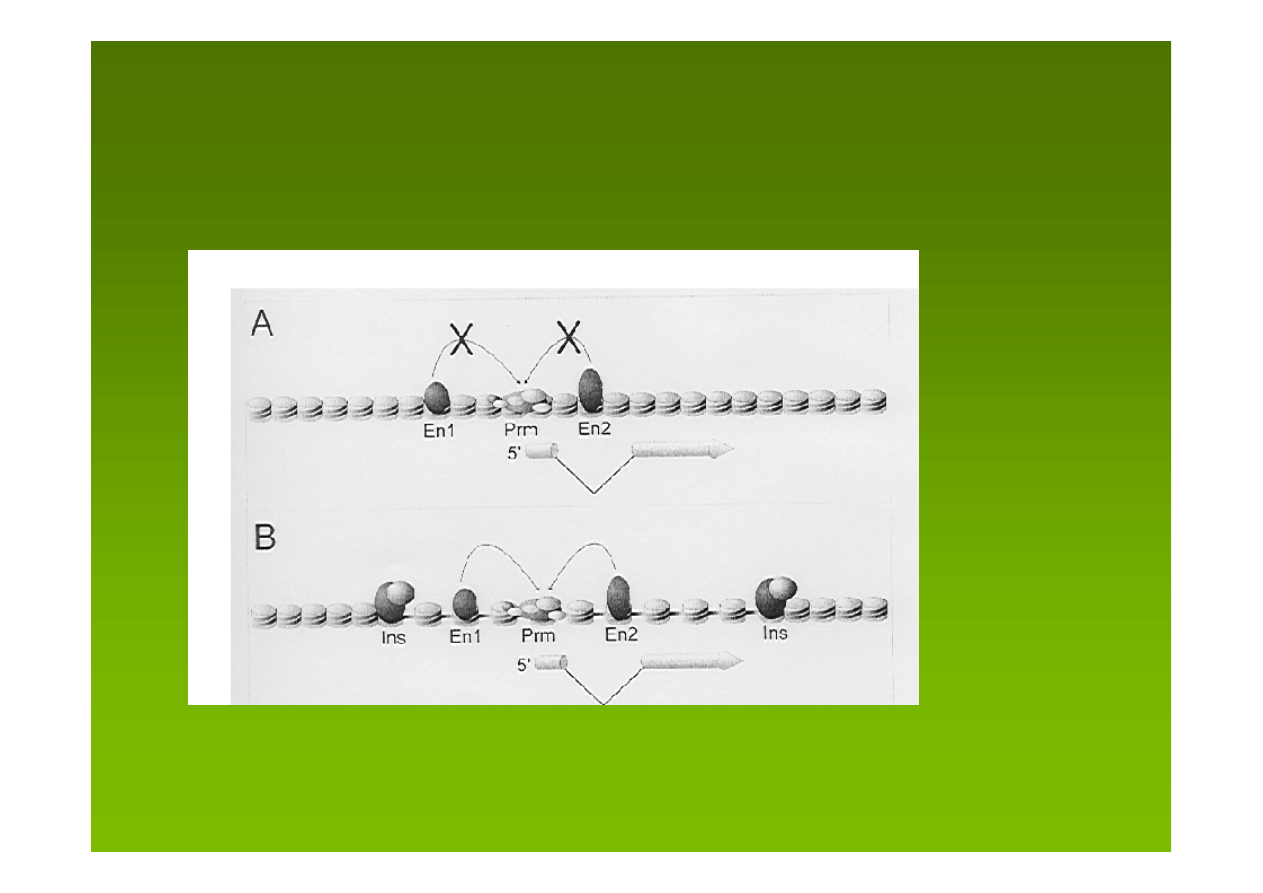
izolatory buforują ekspresję genu od
represji/wyciszającego wpływu przyległej
heterochromatyny
En – enhancer
Prm – promotor
Ins - izolator
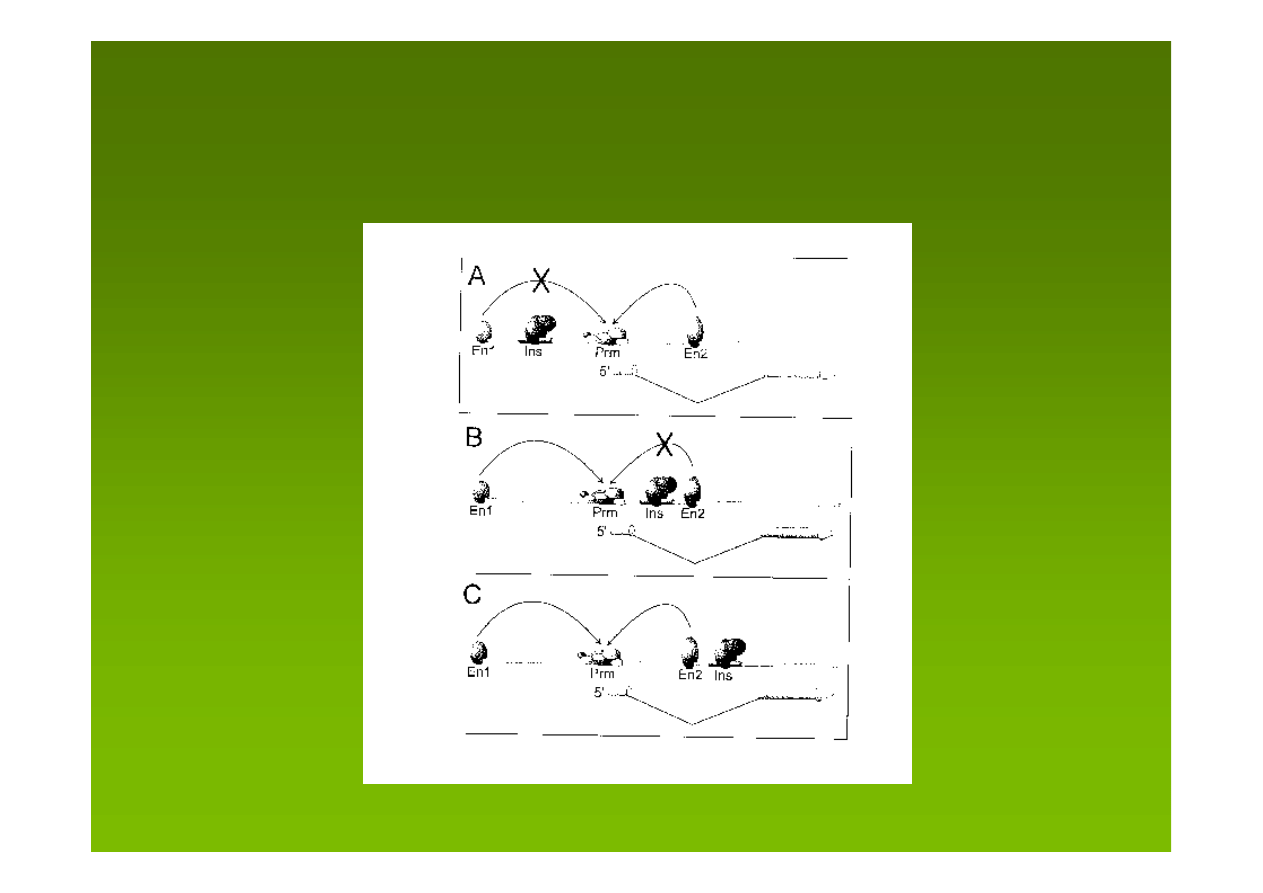
Efekt polarny izolatorów
na oddziaływania promotor-enhancer

Inne (niż dostępność czynników transkrypcyjnych)
mechanizmy regulacji transkrypcji mogące powodować
otwieranie lub represję całych regionów DNA:
* regulacja transkrypcji poprzez przyczepienie
DNA do macierzy jądrowej
* buforowanie przez izolatory
* heterochromatynizacja/remodelowanie chromatyny
* współzawodnictwo enhancerów w LCRach
Jeden ze sposobów wykrywania udostępniania DNA dla czyn-
ników transkrypcyjnych (co oznacza brak blokowania przez
upakowanie i/lub nukleosomy) jest badanie wrażliwości na
DNazę I.
Brown TA Genomy 2009 PWN

otrzymywanie macierzy jądrowej (NM) - ekstrakcja jąder
komórkowych za pomocą:
* LIS (
ang. lithium-3’,5’ diiodosalicylate – łagodny detergent
) lub
* TRITON X-100 + 1 M NaCl (
detergent+wysokie stężenie soli
)
S/MARy (ang. Scaffold/Matrix Attachment Region)
sekwencje DNA o dużym powinowactwie wiązania NM; zwykle zawierają
70% par A+T; co najmniej 200pz; zakotwiczają pętle chromatyny w NM
białka wiążące S/MARy:
gr.I wiążą kooperatywnie (topoizomeraza II, laminy, hnRNP, histon H1)
gr.II rozpoznają specyficzne sekwencje (np. ARBP ang. attachment
region binding protein)
gr.III rozpoznają regiony niesparowanych zasad w S/MARach
(np. STAB1 i nukleolina)
gr.IV wiążące DNA w sposób podobny do distamycyny – wiążą rowek
mniejszy sekwencji oligoA-oligoT (HMGI/Y ang. high mobility group
protein)

w genie lizozymu rolę izolatora pełni
sekwencja
SAR A
•
W przejściowej transfekcji (transgen na
plazmidzie) miejsce
SAR A
ulokowane po
obu stronach nie zwiększa ekspresji genu
CAT kierowanej przez sam
pr
omotor ani
pr
omotor-
e
nhancer, jednakże interferuje
we wzajemna interakcję
pr
omotora i
e
nhancera, gdy znajduje się pomiędzy nimi
CAT act.
---
A
---
Pr
CAT-
A
5%
---
E
---
Pr
CAT---- 100%
A
-
E
---
Pr
CAT-
A
-
112%
E
-
A
---
Pr
CAT----
29%
E
------
Pr
CAT----
67%
• W transfekcji stabilnej (transgen zintegrowany z genomem) miejsce
SAR A
zapewnia ekspresję niezależną od miejsca integracji, a poziom
ekspresji odpowiada liczbie zintegrowanych kopii. Bez
SAR A
poziom
ekspresji transgenu zależy od sekwencji flankujących miejsce
integracji.
• SAR A
pełni rolę elementu granicznego dla funkcji enhancera.
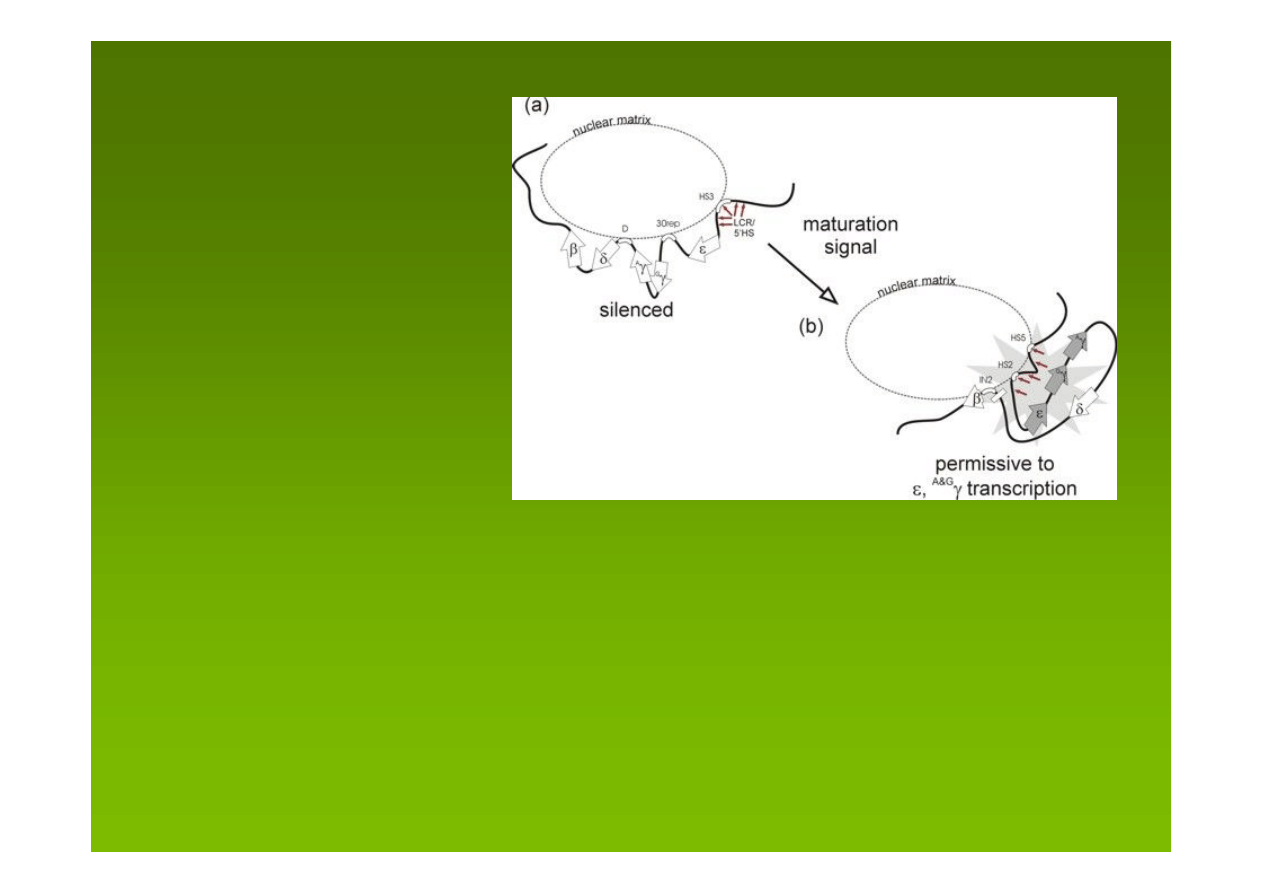
(a) komórki, w których locus β-globiny jest wyciszony – uważa się, że
w hamowaniu ekspresji bierze udział przyczepienie regionu
LCR/5'HS do NM, oddzielenie i wyciszenie tego locus wzmacnia
przyczepienie dwu innych regionów pomiędzy LCRem a genem
β-globiny
(b) komórki z ekspresją genów
ε γ
G
γ
A
– w odpowiedzi na sygnał
wyzwalający ich dojrzewanie uwalniane są z NM regiony
pomiędzy LCRem a genem β-globiny uprzednio związane z NM,
a domena chromatynowa ulega remodelowaniu.
MARy a regulacja
locus β-globiny
locus β-globiny składa się
z genów 5‘ ε γ
G
γ
A
δ β 3’,
które ulegają ekspresji
w sposób rozwojowo
i komórkowo zależny

NM a nowotwory
Białka NM służą jako markery do odróżniania komórek
normalnych i nowotworowych.
Wykazano, że białka NM można wykryć w surowicy i moczu pacjentów
nowotworowych.
W metastatycznych komórkach mysich transformowanych onkogenem
obserwowano duże zmiany kształtu jądra skorelowane ze zmianami
w białkach NM.
NM i czynniki transkrypcyjne
Czynniki transkrypcyjne są zasocjowane z NM.
Uważa się, że NM rekrutuje czynniki transkrypcyjne ułatwiając ich
oddziaływanie z elementami regulatorowymi.
W linii komórek raka sutka MCF-7 receptor estrogenu (czynnik
transkrypcyjny istotny dla ich hormono-zależnej proliferacji)
oraz HET/SAF-B, białko wiążące MAR będące represorem genu
hsp27, są związane
in situ
z sekwencją MAR.
Czynniki transkrypcyjne związane z NM
in situ
związane są też
z MARami, a NM nie jest po prostu magazynem nieaktywnych
czynników/kofaktorów.
http://www.umanitoba.ca/institutes/manitoba_institute_cell_biology/MICB/davie_jim_2.htm
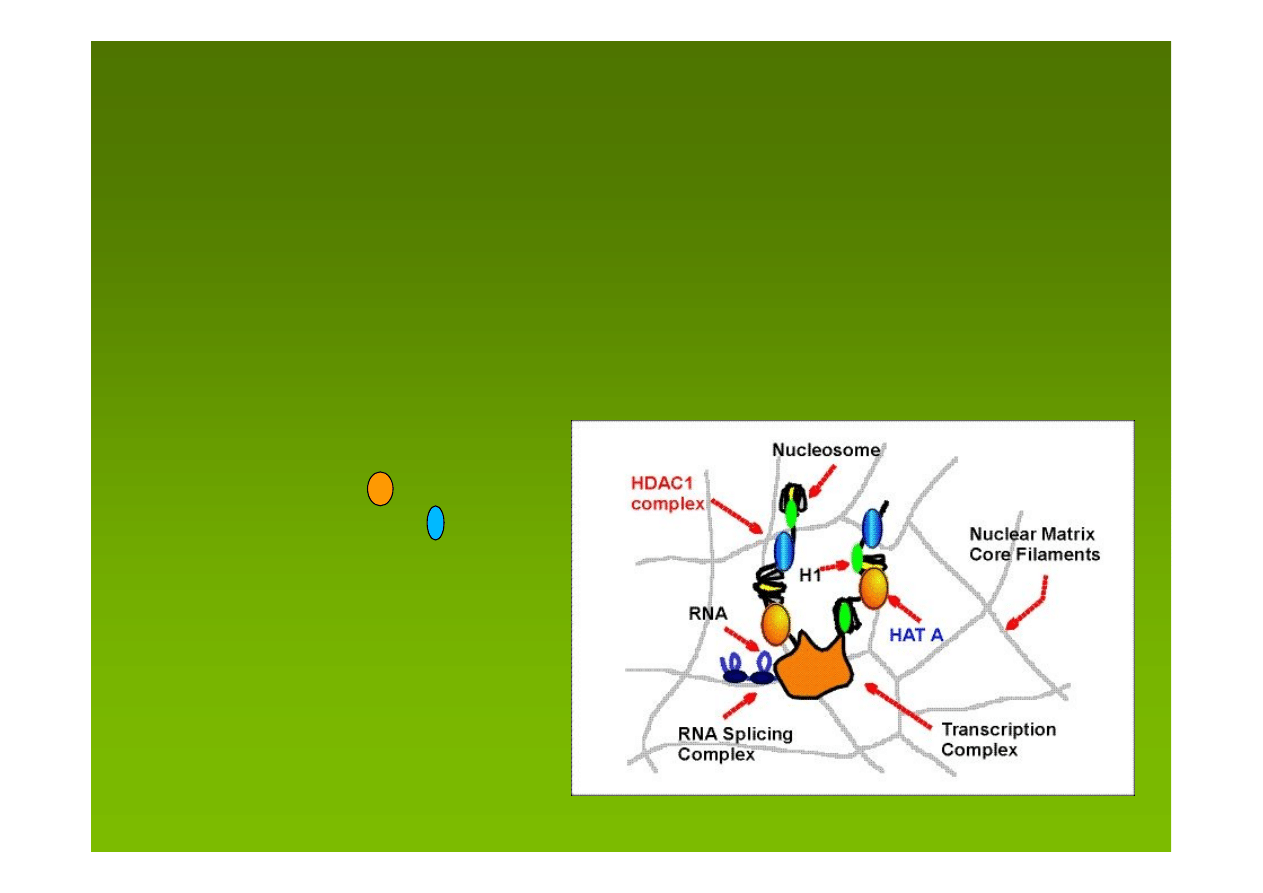
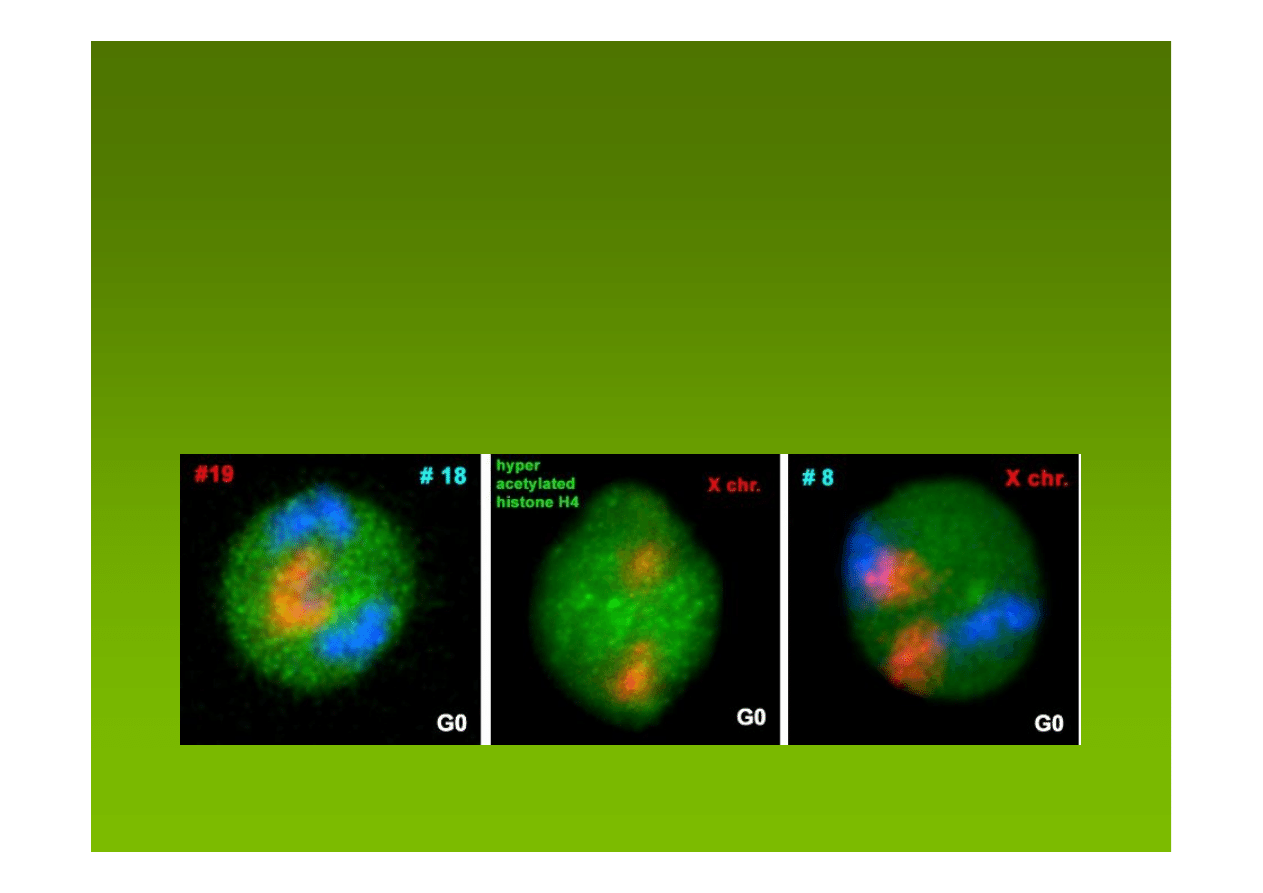
NM a acetylacja histonów
Dynamicznie acetylowane histony są powiązane z
transkrypcyjnie aktywnym DNA. W kurzych komórkach
erytroidalnych najbardziej dynamicznie acetylowane
histony są związane z chromatyną, która prawdopodobnie
jest przyczepiona do NM.
Enzymy katalizujące
acetylację HAT
i
deacetylację HDAC
histonów są związane z NM.
Tylko regiony aktywnie
transkrybowanej chromatyny
są związane z NM poprzez
wiele dynamicznych miejsc
wiązania zapewne dzięki
HAT i HDAC.
http://www.umanitoba.ca/institutes/manitoba_institute_cell_biology/MICB/davie_jim_2.htm
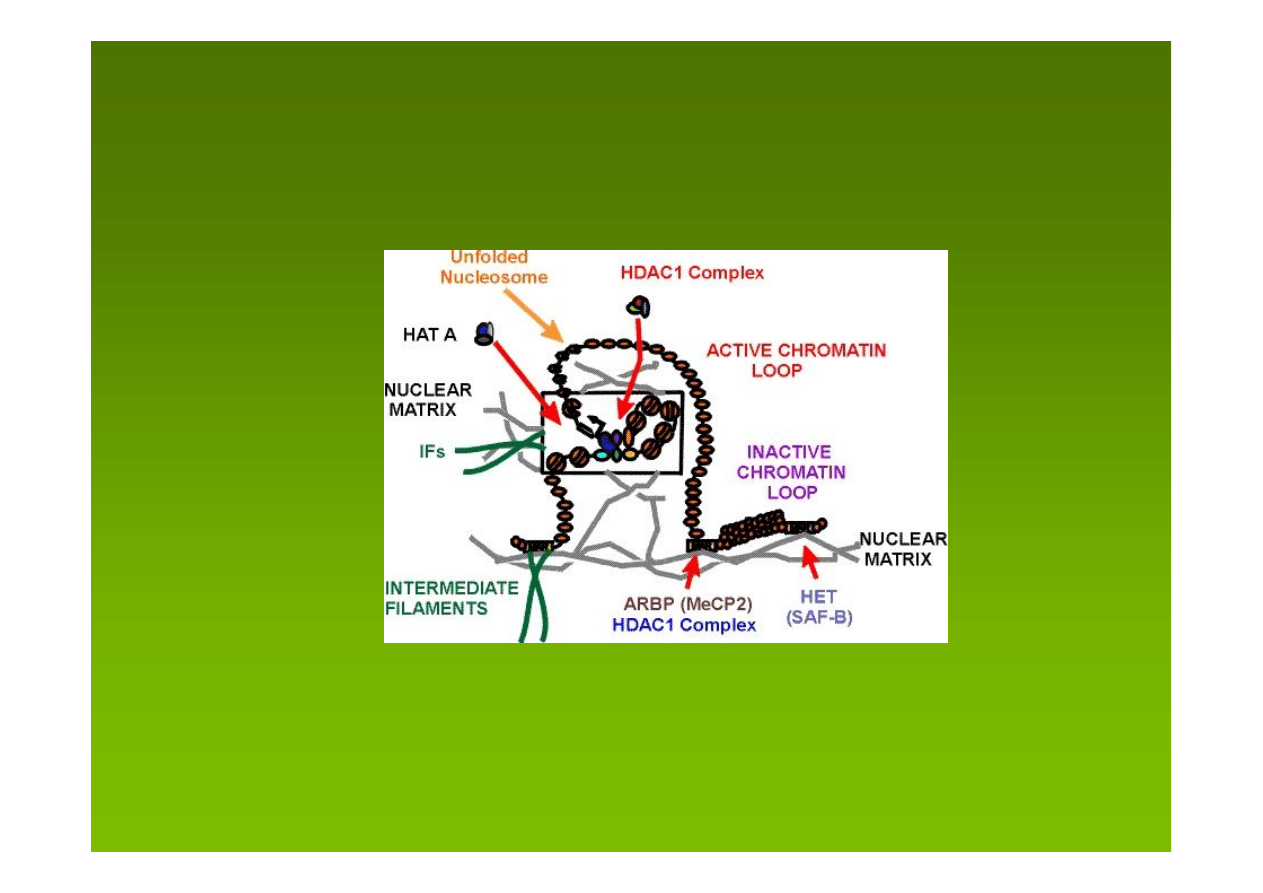
Traktowanie cisplatyną ludzkiej linii komórek raka sutka
sprzęga HDAC1 (enzym deacetylujący histony) z sekwencjami
MAR, co sugeruje, że HDAC1 jest związany z MAR przy
podstawie pętli.
W kurzych erytrocytach, dynamicznie acetylowane histony H3 i H4 są
selektywnie metylowane. Metylacja nie jest zależna od właśnie
zachodzącej acetylacji i
vice versa
. Można przypuszczać, że enzymy
HAT, HDAC i HMT kolokalizują w przestrzeni jądrowej.
http://www.umanitoba.ca/institutes/manitoba_institute_cell_biology/MICB/davie_jim_2.htm
Wyszukiwarka
Podobne podstrony:
biolmol3
BiolMol 10 id 87436 Nieznany
BiolMol 12 id 87437 Nieznany (2)
biolo-molo giełdy, biolmol3
2012 10 26 BiolMol sylabus1 Zb
BIOLOGIA MOLEKULARNA Z BIOTECHNOLOGIí(2), UG, MOLEKUŁY, Biolmol, Biolmol1
BiolMol 8 id 87438 Nieznany
biolo-molo giełdy, biolmol2, 1
Biotechnologia i biologia molekularna1 (1), UG, MOLEKUŁY, Biolmol
biolmol-PCR, far, III rok IV sem, biologia molekularna, notatki
biolmol kartkówki z forum
BiolMol UMP pytania opracowane
biolmol3
ściąga biolmol 2
biolmol 2 termin
więcej podobnych podstron