
METODY MOLEKULARNE I
Katarzyna Stasik
gr. 47

Materiał biologiczny
• Pełna krew obwodowa
• Komórki nabłonka
• Hodowla fibroblastów
• Komórki płynu owodniowego (AFC)
• Komórki kosmówki (CVS)
• Cebulki włosów
• Plamy krwi, nasienia, fragmenty tkanek pobrane metodą biopsji
cienkoigłowej, szpik kostny

Pobieranie materiału genetycznego
Badanie diagnostyczne
1 ml krwi obwodowej pobranej na EDTA, heparynę
10 mg tkanki- bezpośrednia izolacja DNA
10 ml płynu owodniowego hodowla komórek w naczyniach hodowlanych
Włosy, wymazy z jamy ustnej
Do badania PCR:
50 ul krwi
10 ul nasienia
Materiały archiwalne

Przechowywanie próbek
• +4 C (do 48 godzin)
• Materiał zamrożony w -20, -80 lub -195
• Materiał utrwalony w alkoholu
• Materiał umieszczony w buforze do lizy (Tris HCl, SDS, EDTA, ph 8,0)
Materiał suchy (plamy)
• W kopertach w temp pokojowej w suchym miejscu

Badanie prenatalne
Inwazyjne:
• Biopsja kosmówki
• Amniopunkcja
• Kordocenteza
• Fetoskopia
Nieinwazyjne:
• USG + doppler
• Badania przesiewowe oparte na oznaczaniu specyficznych substancji pochodzenia płodowego
obecnych w surowicy krwi matki.
• Badanie komórek i DNA pochodzenia płodowego obecnych w krążeniu matczynym.
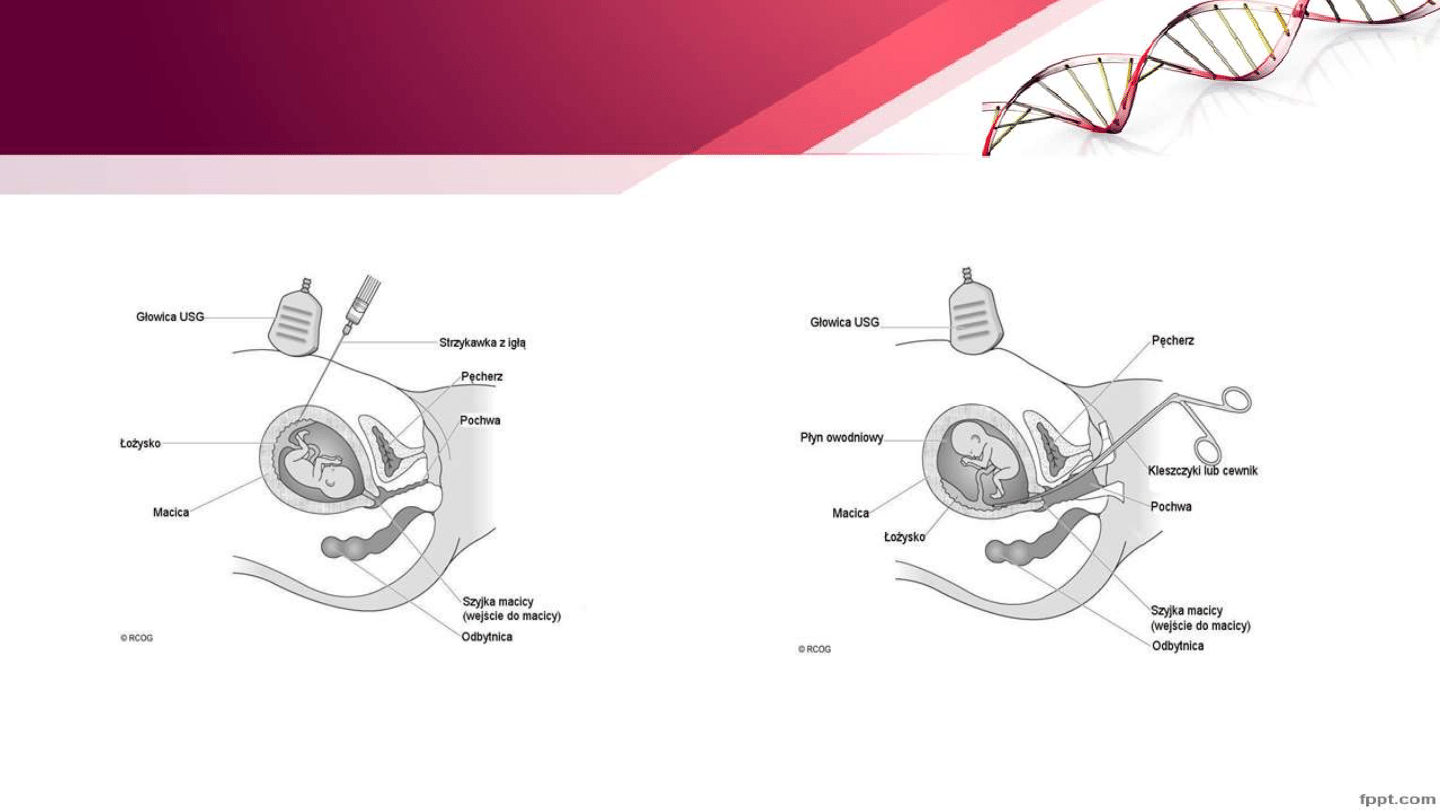
Techniki pobierania materiału do badań
prenatalnych – Biopsja kosmówki
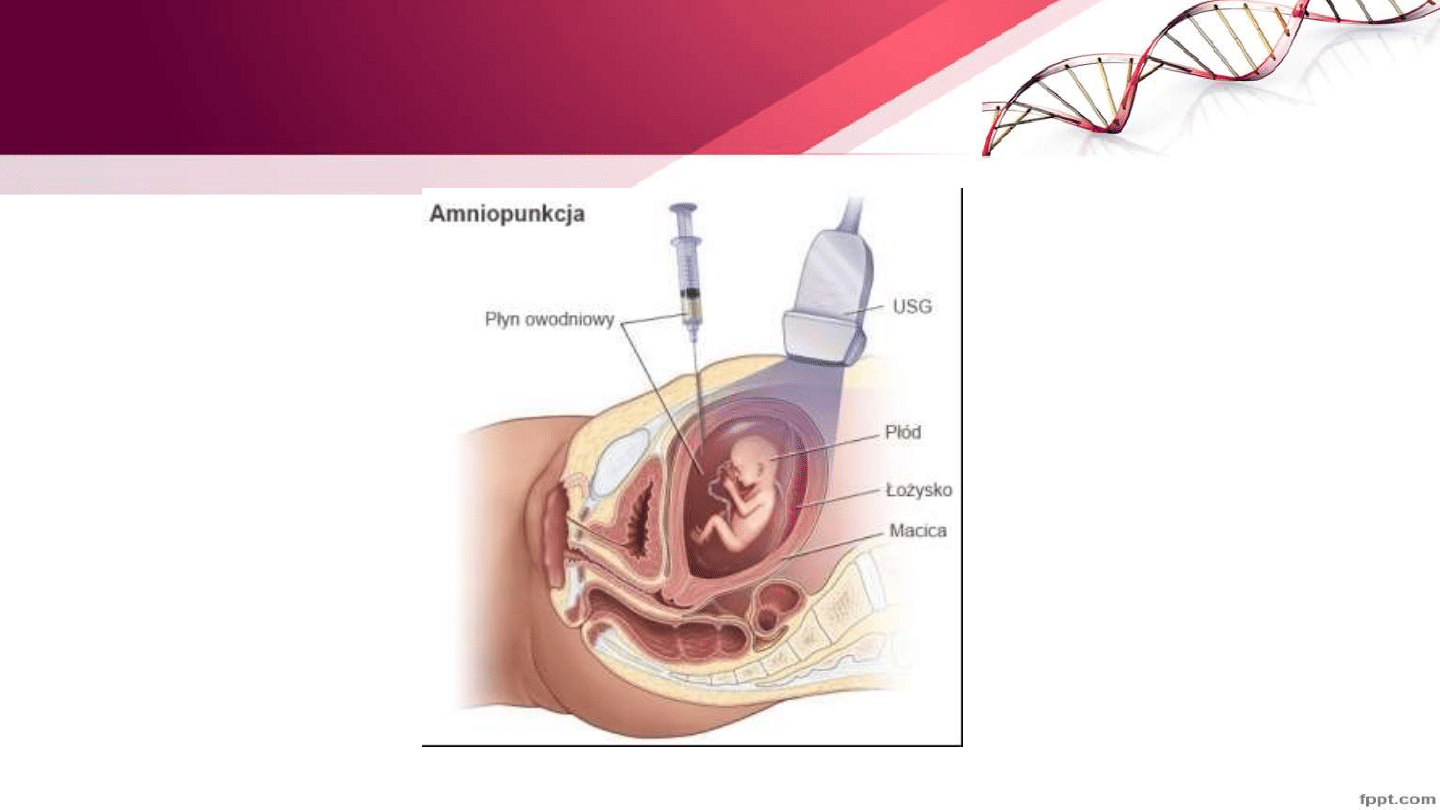
Techniki pobierania materiału do badań
prenatalnych- Amniopunkcja
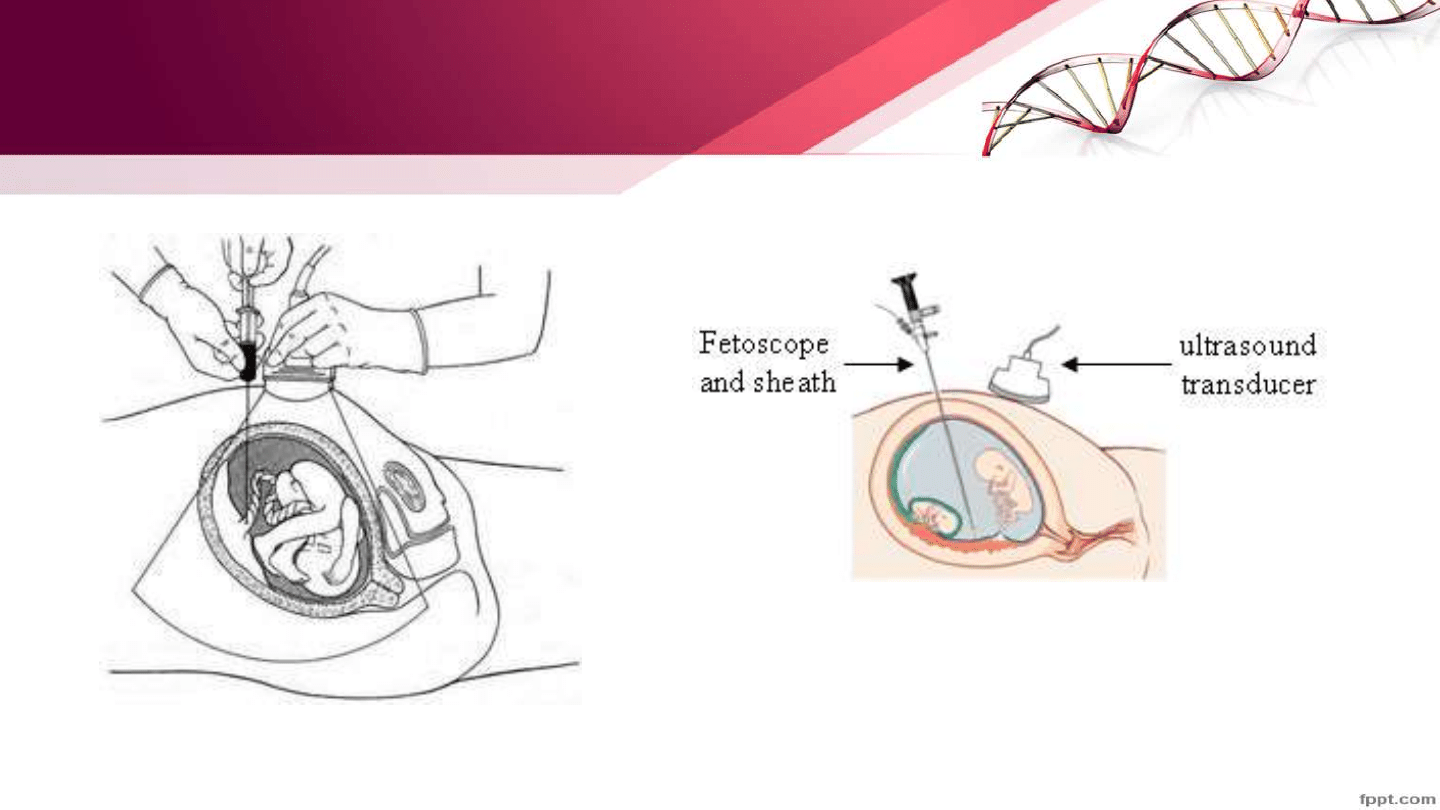
Techniki pobierania materiału do badań
prenatalnych- Kordocenteza i Fetoskopia

Izolacja DNA
• Liza komórki
• Denaturacja i hydroliza białek
• Usunięcie białek
• Zagęszczenie DNA

Odbiałczenie preparatu
• Z zastosowaniem fenolu i chloroformu (najwyższa czystość)
• Wysolenie białek z lizatów komórek
• Po związaniu DNA z nośnikiem, odmyciu zanieczyszczeń i uwolnieniu
DNA. (kolumny chromatograficzne z filtrami jonowymiennymi lub
krzemionkowymi

Izolacja RNA
Metody izolacji w zależności od typu RNA, można przeprowadzić
• Izolację specyficznego RNA w wyniku frakcjonowania całkowitego RNA
komóki
• Bezpośrednią izolację specyficznego RNA ograniczoną do komórek
syntetyzujących określony RNA w większej ilości
• Izolację poli(A) RNA
• Izolację RNA z polirybosomów
• Izolację całkowitego RNA komórki, a następnie uzyskanie określonego RNA w
postaci cDNA w procesie odwrotnej transkrypcji

Zasady izolacji RNA
• Stosowanie silnych związków degradujących białka (chlorowodorek
guanidyny, izotiocyjanian guanidyny) do inaktywacji Rnaz
• Usunięcie RNAz ze sprzętu- DEPC (dietylopirowęglan)
• Usunięcie DNA podczas izolacji RNA (ekstrakcja fenolem o niskim pH;
trawienie Dnazą) można wytrącić RNA w chlorku litu lub wirowanie w
dwustopniowym gradiencie tiocyjanianu guanidyny i chlorku cezu
• Izolacja mRNA z zastosowaniem poliT przyłączonymi do stałego podłoża np. w
kolumnach chromatograficznych lub na kuleczkach magnetycznych
(niezwiązane RNA odmywane, pozostałe poddawane elucji)

Ocena jakościowa i ilościowa wyizolowanych
kwasów nukleinowych
Jakość kwasów nukleinowych ocenia się:
• W elektroforezie analitycznej w żelu
• Metodą spektrofotometryczną pomiarów absorbancji
• Metodą mikromacierzy w połączeniu z elektroforezą mikrokapilarną

Elektroforeza na żelu
• W celu sprawdzenia zarówno jakości, jak i ilości preparatu DNA należy
przygotować 0,8% żel agarozowy, zawierający 0,5 μ/ml bromku etydyny
w buforze 1 x TBE (tj. 10 x TBE: 0,89 M Tris, 0,89 M kwas borowy, 0,02 M
EDTA, pH=8.0), następnie nakłada się 0,5; 1,0; oraz 5,0 μg preparatu o znanym
stężeniu, oraz preparaty do oszacowania (w buforze obciążającym).
• Następnie po przeprowadzonej elektroforezie, ilość i jakość preparatów
ocenia się na transiluminatorze, który emituje światło o długości 312 nm.
• Jeżeli w porównaniu do markera, wielość wyizolowanego DNA migruje
w postaci wartego prążka o wielkości ponad 50 kpz, bez widocznych smug,
o oznacza to, że wyizolowane DNA jest wysokocząsteczkowe

Ocena spektrofotometryczna
• Preparaty do pomiaru absorbancji muszą być wcześniej
rozcieńczone (najczęściej 100 x H2O lub buforem TE). Oznaczenie
wykonuje się za pomocą pomiary fluorescencji
(spektrofotometrem). Po kalibracji spektrofotometru odczytuje
się punktowo wartość absorpcji przy 260, 280 i 320 nm, bądź też
można wykonać widmo pomiarów w zakresie od 200 do 350
nm.

Metoda mikromacierzy
• Podstawą działania mikromacierzy jest, tak jak w tradycyjnej hybrydyzacji
Southerna, komplementarność kwasów nukleinowych. Mikromacierz zawiera
sekwencje komplementarne do badanych sekwencji.
• Próbkę kwasu nukleinowego wyznakowuje się (jednym lub dwoma
znacznikami fluorescencyjnymi) i hybrydyzuje z mikromacierzą.
• Cząsteczki wyznakowanego kwasu nukleinowego wiążą się do
komplementarnych sekwencji.
• Obraz odczytuje się ilościowo (za pomocą lasera lub mikroskopu).
Intensywność sygnału dla poszczególnych sond mikromacierzy jest
proporcjonalna do ilości kwasu nukleinowego o danej sekwencji w próbce.
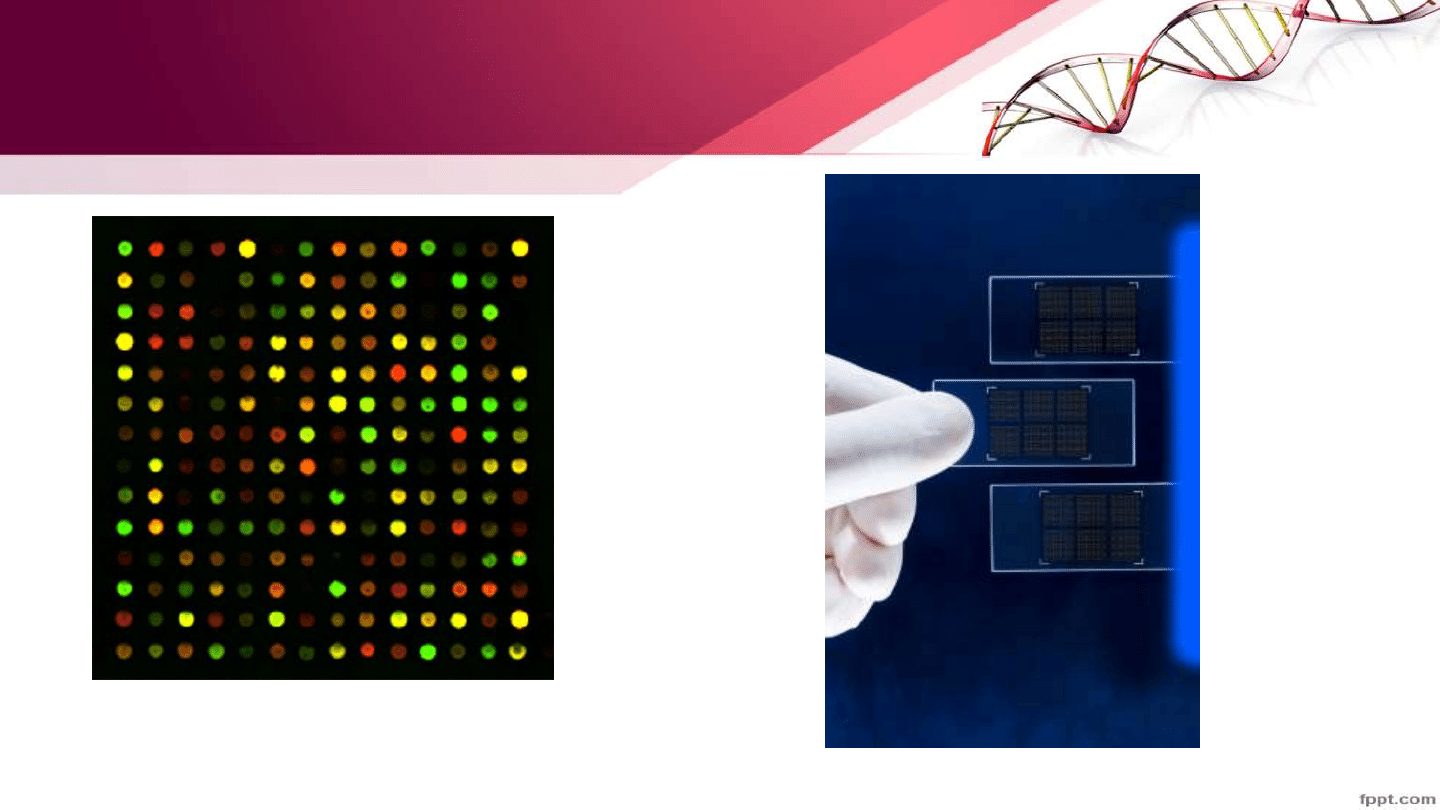
Metoda mikromacierzy

PCR
• Reakcja łańcuchowa polimeryzacji
opracowana została w latach 80-tych przez
Kary'ego Mullisa (Nagroda Nobla (1993 r.)).
• Reakcja ta umożliwia powielanie wybranych
fragmentów DNA za pomocą procesu
enzymatycznego.
• Konieczna jest znajomość sekwencji DNA na
obu końcach regionu, który ma być
skopiowany.
• Namnożony fragment DNA jest
wykorzystywany do analizy lub manipulacji
genetycznych

Skład mieszaniny reakcyjnej PCR
1)matrycowy DNA
2)primery
3)Trifosforany deoksynukleozydów (dNTP) substraty dla polimerazy DNA
4) polimeraza DNA (termostabilna)
5)bufor
6)jony Mg2+ wiązane przez startery, matrycę polimerazę i dNTP
7)Substancje stabilizujące polimerazę

Etapy reakcji PCR
1) Denaturacja- temp 95 C rozplecenie dwuniciowej helisy DNA
2) Hybrydyzacja odcinków starterowych z sekwencjami DNA otaczający
gen namnażany (annealing) 45-60C
3) Wydłużanie (elongacja) synteza DNA przez polimerazę DNA
dobudowującą kolejne nukleotydy do starterów 72 C

RT-PCR
PCR, w którym pierwszy etap jest przeprowadzony przez odwrotną
transkryptazę, a jako matryca służy cząsteczka mRNA.
Podstawowe cechy i zalety Reverse Transcriptase PCR:
• umożliwia utworzenie cDNA na matrycy RNA
• cDNA jest znacznie stabilniejszy niż RNA
• cDNA może być amplifikowany za pomocą PCR

Skład mieszaniny reakcyjnej RT-PCR
1) RNA(wysokiej jakości i wolny od zanieczyszczeń)
2) primery (startery)
3) dNTPs
4) bufor
5) odwrotna transkryptaza
6) polimeraza Taq

• RT-PCR może być użyty do analizy ilościowej (quantitive) lub pół-
ilościowej(semi-quantitative). Analiza pół-ilościowa pozwala nam
określić, czy jeden transkrypt ulegał większej ekspresji niż inny
natomiast ilościowa pozwala nam określić całkowitą ilość danego
transkryptu w badanym materiale.
• Metoda pół-ilościowa może być używana do określenia obecności lub
nieobecności danego transkryptu, określenia jego ilości, klonowania,
tworzenia bibliotek cDNA, konstrukcji sond.

Real Time-PCR
• PCR w czasie rzeczywistym ilościowy pomiar kopii genu, określenie
poziomu ekspresji RNA w tkankach
• Umożliwia monitorowanie zmian stężenia produktu PCR przez pomiar
fluorescencji dzięki stosowaniu barwników sond fluorescencyjnych
• Termocykler + fluorymetr
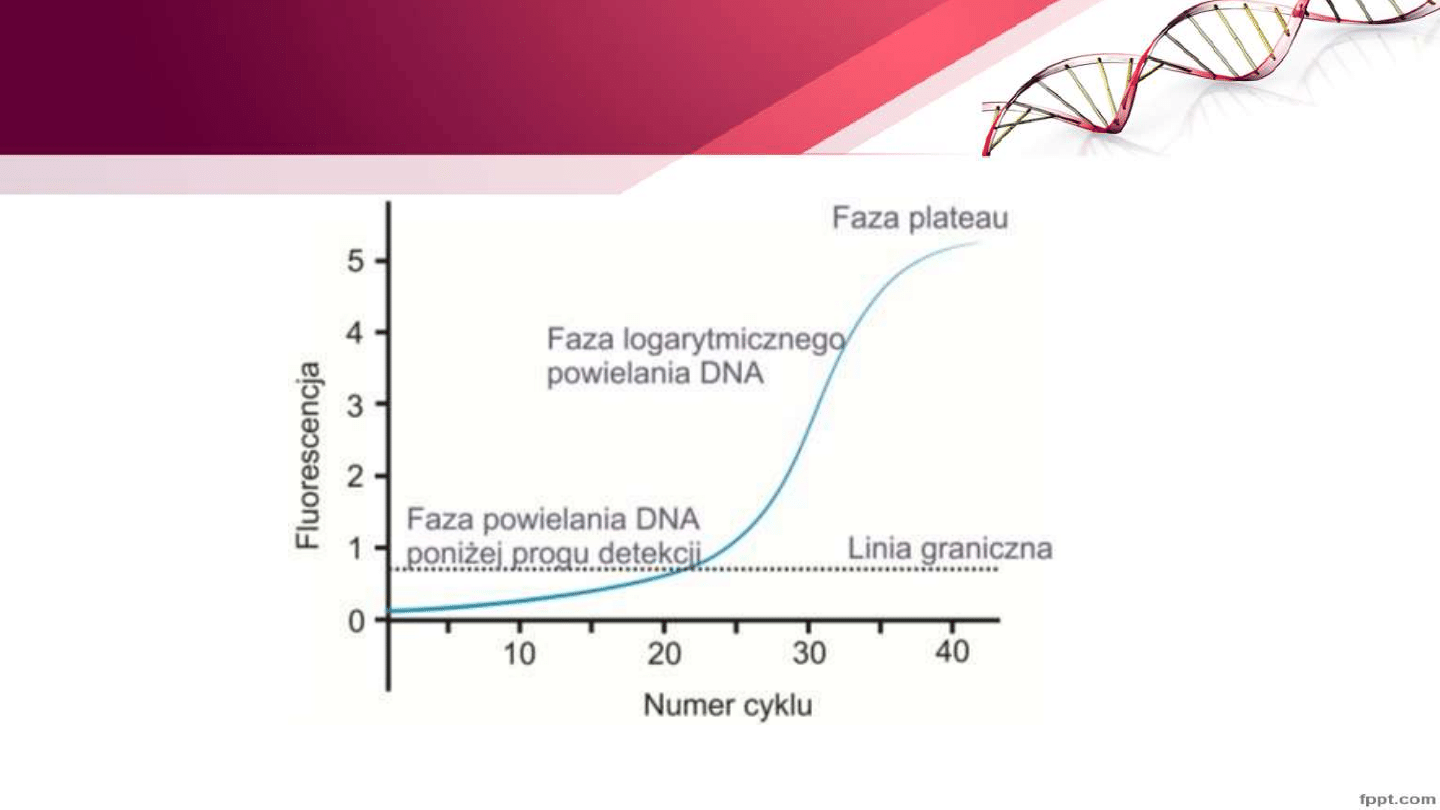
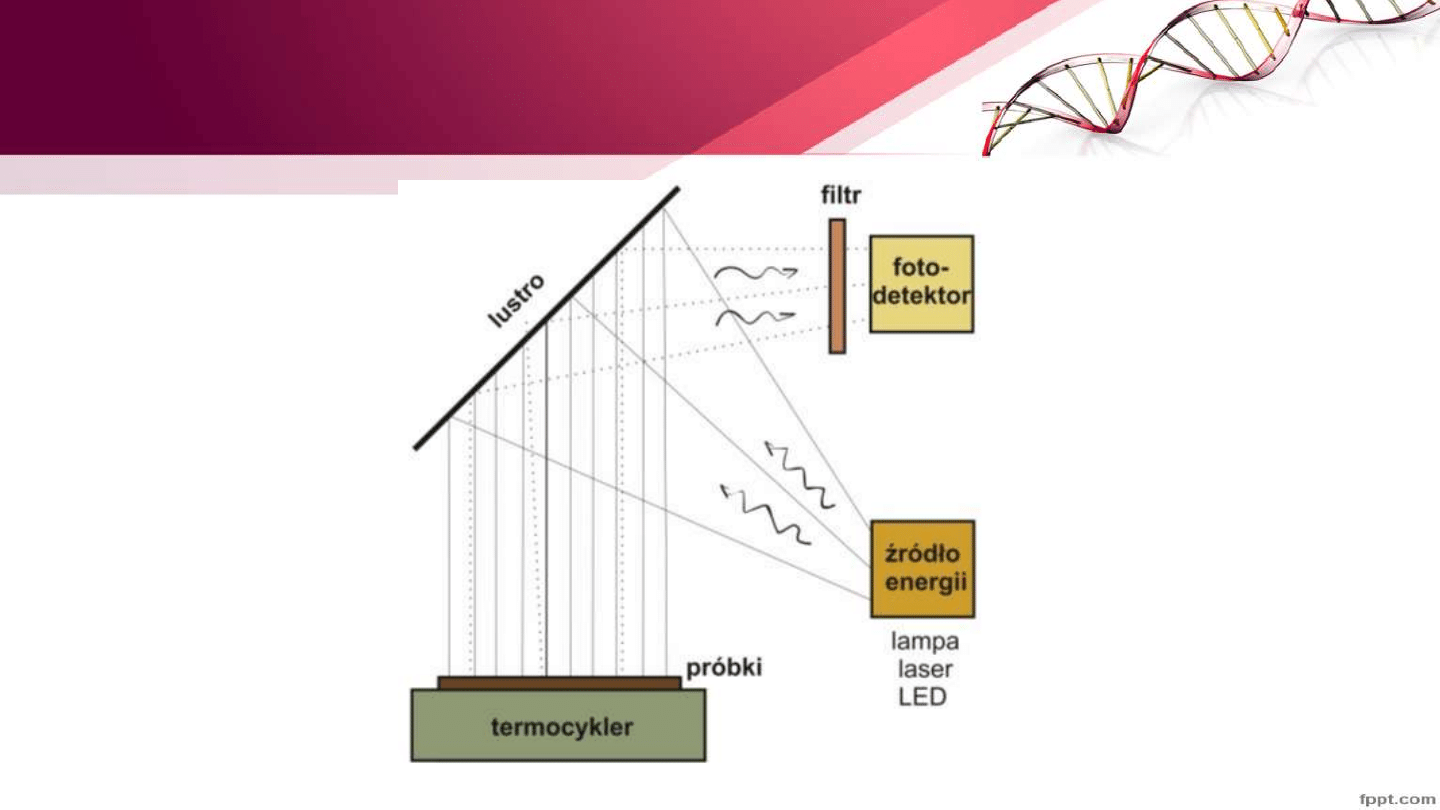
Termocykler do Real Time PCR

PCR multipleks
• Amplifikacja wielu matryc podczas jednej reakcji PCR (wykorzystanie
różnych zestawów primerów (zwiększa ryzyko błędów, krzyżowa
hybrydyzacja)
• Primery powinny mieć podobną kinetykę reakcji, powinny być
specyficzne
• Amplikony powinny różnić się między sobą długością. rozdział na
pojedynczym żelu elektroforetycznym. Wahania te nie mogą być jednak
zbyt duże, gdyż może to uniemożliwić ich
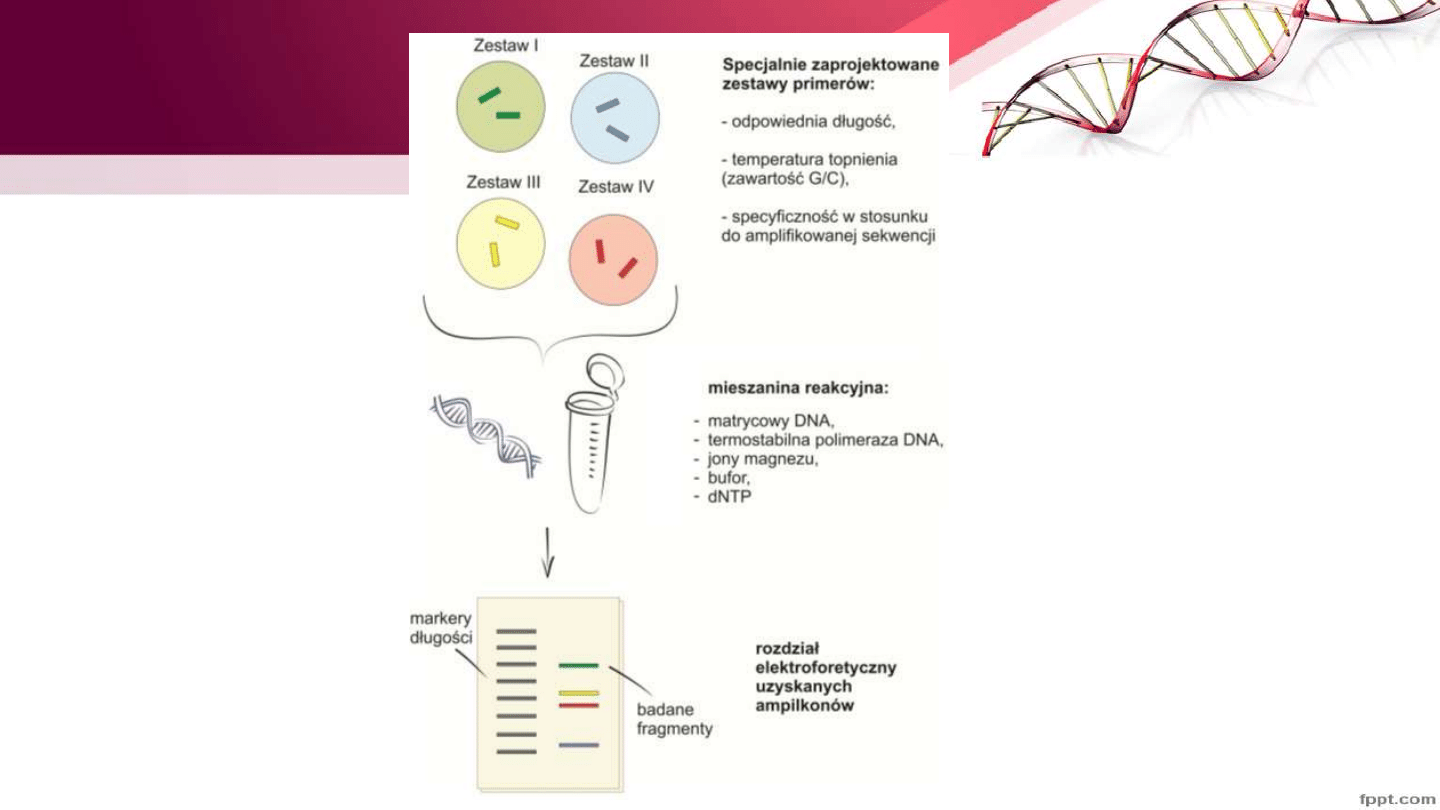

Zastosowanie Multipleks PCR
•
wykrywanie delecji i innych zmian w sekwencji docelowej
•
Polymorphic Repetitive DNA (polimorficzne powtarzalne sekwencje DNA) - Multipleks PCR to
idealna technika do profilowania DNA, czyli identyfikacji poszczególnych osobników. Krótkie tandemowe
powtórzenia (STR ang. Short Tandem Repeats) lub inaczej sekwencje mikrosatelitarne bardzo dobrze nadają
się do techniki multipleks, ze względu na swoje liczne występowanie oraz wysoki polimorfizm. Stosuje się je
m.in. przy wykrywaniu chorób, mapowaniu genów oraz w analizach kryminalistycznych.
•
wykrywanie i charakteryzacja mikroorganizmów grzybów, bakterii , wirusów oraz pasożytów.

DZIĘKUJĘ ZA UWAGĘ
Wyszukiwarka
Podobne podstrony:
10 Metody molekularne cz 2 Repa
10 Metody molekularne cz 2 Repa
10 Metody otrzymywania zwierzat transgenicznychid 10950 ppt
10 Metody otrzymywania sztywnych pianek poliuretanowych
10 Biologia molekularna 5 12 2011
6 Metody zamrażania cz 1
Metodyka szczęścia cz 4
10. Metody oceny sposobu żywienia, licencjat(1)
Metodyka szczęścia cz.2
Wykład 10-Równania nieliniowe cz.1
10 2 metodyka szkolenia2id 10746 ppt
10 1 metodyka szkolenia1id 10717 ppt
Metodyka szczęścia cz 2
Wykład 10, procesy poznawcze cz. II
laboratorium 9 i 10, Metody probabilistyczne i statystyka
10. Metody i techniki organizatorskie I, Organizacja i zarządzanie
Metodyka szczęścia cz 1
więcej podobnych podstron