0000023(1)
gładkie, wysokie AAKK
AK
omszone, karłowate
aabb
gładkie, karłowate
P: AAbb
omszone, wysokie j.i KK
a b
ttktdkic. wysokie
AaKb'
AK
, ab || Ab 0.47 0.47 0.0}
aK
0.03
|
■O |
AK |
ab |
Ab |
aK |
|
0.47 Ali |
AAKK 0,221 |
AaKb 0.221 |
AAKb 0.014 |
AaKK 0.014 |
|
0.47 ab |
AaBb 0.221 |
aabb 0.221 |
Aabb 0.014 |
aaKb 0.014 |
|
0,03 Ab |
AAKb 0,014 |
Aabb 0.014 |
AAbb 0.001 |
AaKb 0.001 |
|
0.03 .t# |
AaKK 0.014 |
aabb 0.014 |
AaKb 0.001 |
aaKK 0.001 |
72.1 % potomstwa 2.9% potomstwa 2.9% potomstwa 22.1% potomstwa
G:
G:
/U>
.i K
gładkie, wysokie AaKb
AI>
. d# II AK . 0.47 0.47 0.03
ab
0.03
|
n |
Ab |
aK |
AK |
ab |
|
0.47 Ab |
AAbb 0.221 |
AaKb 0.221 |
AAKb 0.014 |
Aabb 0.014 |
|
0.47 aK |
AaKb 0.221 |
aaKK 0.221 |
AaKK 0.014 |
aaKb 0.014 |
|
0.03 AK |
AAKb 0.014 |
AaKK 0,014 |
AAKK 0.001 |
AaKb 0.001 |
|
0.03 ab |
Aabb 0.014 |
aaKb 0.014 |
AaKb 0.001 |
aabb 0.001 |
A_K_
Abb
aaB_
aabb
50,1% potomstwa 24.9% potomstwa 24.9% potomstwa 0.1% potomstwa
KRZYŻÓWKA TESTOWA
(zF,)/l.iMł x aabb
KRZYŻÓWKA TESTOWA (z Fj )AaKb X aabb
|
G: |
AK . |
ab || |
Ab . |
aK |
; ab |
G: |
Ab . |
aB II |
AK , |
ab |
; ab |
|
P = |
0.47 |
0,47 |
0.03 |
0.03 |
P ■ |
0.47 |
0.47 |
0.03 |
0.03 | ||
|
AK |
ab |
Ab |
aK |
Ab |
aK |
AK |
ab | ||||
|
17 - |
AaKb |
aabb |
Aabb |
aaKb |
n |
*-v |
Aabb |
aaKb |
AaKb |
aabb |
-3 |
|
r2- |
0.47 |
0.47 |
0.03 |
0.03 |
ab u |
0.47 |
0.47 |
0.03 |
0.03 |
ab n | |
|
47% |
47% |
3% |
3% |
— |
47% |
47% |
3% |
3% |
— |
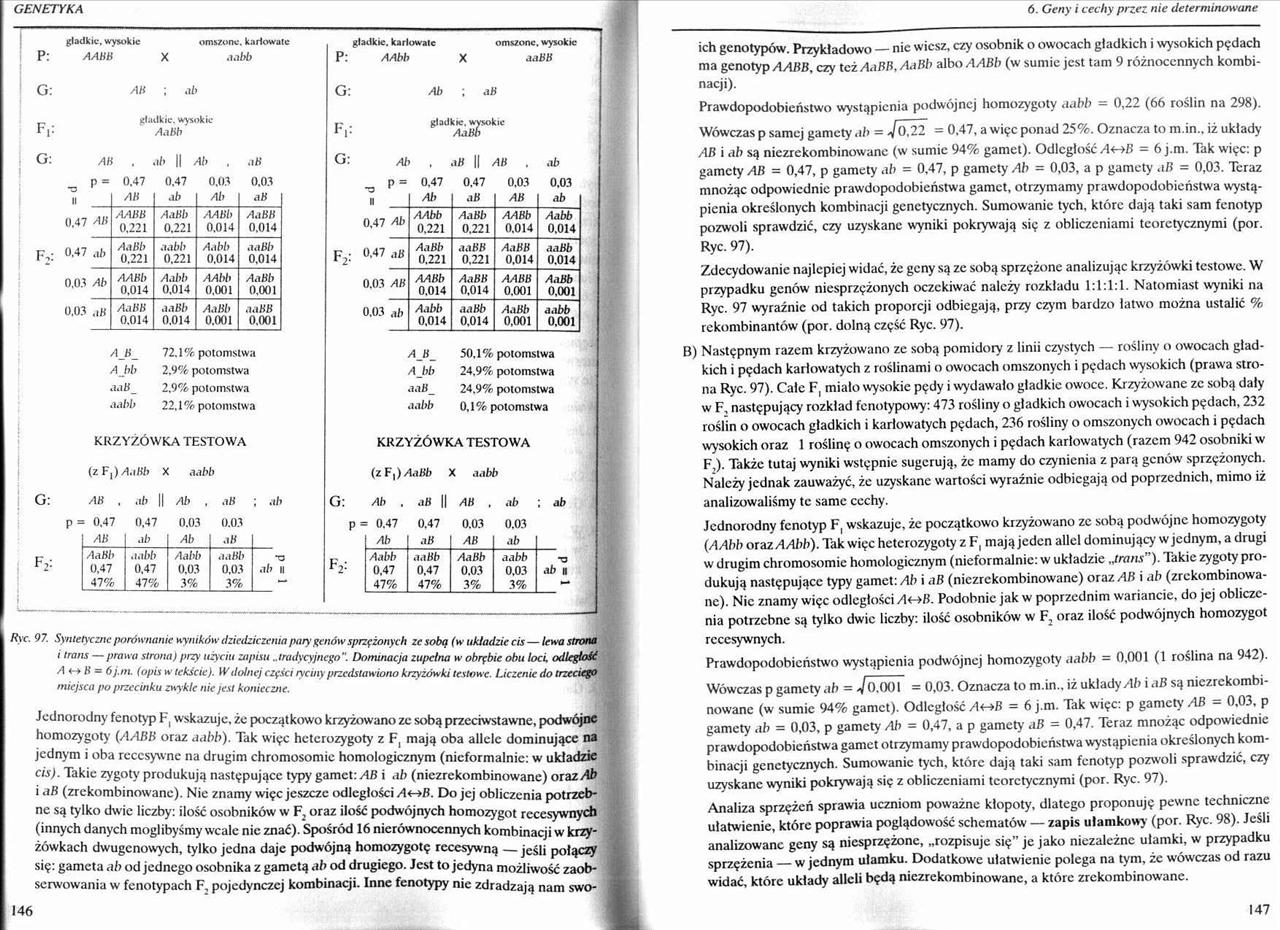
Ryc. 97. Syntetyczne porównanie wyników dziedziczenia pary genów sprzężonych ze sobą (w układzie cis — lewa strona i trans —prawa strona) przy użyciu zapisu ..tradycyjnego". Donunacja zupełna w obrębie obu lock odlegjlośŁ A < *K = 6j.m. (opis w tekście). W dolnej części ryciny przedstawiono krzyżówki testowe. Liczenie do trzeciego miejsca po przecinku zwykle nie jest konieczne.
Jednorodny fenotyp F, wskazuje, że początkowo krzyżowano ze sobą przeciwstawne, podwójne homozygoty (AABB oraz .uibb). Tak więc heterozygoty z F, mają oba alicie dominujące na jednym i oba rcccsywne na drugim chromosomie homologicznym (nieformalnie: w układzie cis). Takie zygoty produkują następujące typy gamet: AB i .i b (niezrekombinowane) oraz Ab i dB (zrekombinowane). Nie znamy więc jeszcze odległości Ac->B. Do jej obliczenia potrzeb ne są tylko dwie liczby: ilość osobników w F, oraz ilość podwójnych homozygot rcccsywnych (innych danych moglibyśmy wcale nic znać). Spośród 16 nierównoccnnych kombinacji w krzyżówkach dwugcnowych, tylko jedna daje podwójną homozygotę rcccsywną — jeśli połączy się: gameta db od jednego osobnika z gametą db od drugiego. Jest to jedyna możliwość zaobserwowania w fenotypach F, pojedynczej kombinacji. Inne fenotypy nie zdradzają nam swoich genotypów. Przykładowo — nic wiesz, czy osobnik o owocach gładkich i wysokich pędach ma genotyp AAKB, czy też AaBB,AdBb albo AABb (w sumie jest tam 9 różnoccnnych kombinacji).
Prawdopodobieństwo wystąpienia podwójnej homozygoty ddbb - 0,22 (66 roślin na 298).
Wówczas p samej gamety db = Jo.22 = 0.47, a więc ponad 25%. Oznacza to m.in.. iż układy AB i db są niczrckombinowanc (w sumie 94% gamet). Odległość/!<->/> = 6 j.m. Tak więc: p gamety AB = 0,47, p gamety db = 0,47, p gamety Ab = 0,03, a p gamety dB = 0,03. Teraz mnożąc odpowiednie prawdopodobieństwa gamet, otrzymamy prawdopodobieństwa wystąpienia określonych kombinacji genetycznych. Sumowanie tych. które dają taki sam fenotyp pozwoli sprawdzić, czy uzyskane wyniki pokrywają się z obliczeniami teoretycznymi (por. Ryc. 97).
Zdecydowanie najlepiej widać, że geny są ze sobą sprzężone analizując krzyżówki testowe. W przypadku genów niesprzężonych oczekiwać należy rozkładu 1:1:1:1. Natomiast wyniki na Ryc. 97 wyraźnie od takich proporcji odbiegają, przy czym bardzo łatwo można ustalić % rckombinantów (por. dolną część Ryc. 97).
B) Następnym razem krzyżowano ze sobą pomidory z linii czystych — rośliny o owocach gładkich i pędach karłowatych z roślinami o owocach omszonych i pędach wysokich (prawa strona Ryc. 97). Całe F, miało wysokie pędy i wydawało gładkie owoce. Krzyżowane ze sobą dały w F, następujący’ rozkład fenotypowy: 473 rośliny o gładkich owocach i wysokich pędach, 232 roślin o owocach gładkich i karłowatych pędach, 236 rośliny o omszonych owocach i pędach wysokich oraz 1 roślinę o owocach omszonych i pędach karłowatych (razem 942 osobniki w F,). Także tutaj wyniki wstępnie sugerują, że mamy do czynienia z parą genów sprzężonych. Należy jednak zauważyć, że uzyskane wartości wyraźnie odbiegają od poprzednich, mimo iż analizowaliśmy te same cechy.
Jednorodny fenotyp F, wskazuje, że początkowo krzyżowano ze sobą podwójne homozygoty (A Abb oraz A Abb). Tak więc heterozygoty z F, mają jeden allel dominujący w jednym, a drugi w drugim chromosomie homologicznym (nieformalnie: w układzie „trans’'). Takie zygoty produkują następujące typy gamet: Ab i dB (niezrekombinowane) oraz AB i ab (zrekombinowa-nc). Nic znamy więc odległości Podobnie jak w poprzednim wariancie, do jej obliczenia potrzebne są tylko dwie liczby: ilość osobników w F, oraz ilość podwójnych homozygot rcccsywnych.
Prawdopodobieństwo wystąpienia podwójnej homozygoty aabb = 0.001 (1 roślina na 942).
Wówczas p gamety ab = V0.001 = 0,03. Oznacza to m.in., iż układy Ab i aB są niczrckombinowanc (w sumie 94% gamet). Odległość A<->B = 6 j.m. Tik więc: p gamety AB = 0.03. p gamety ab = 0,03, p gamety /ti> = 0,47, a p gamet)’ aB = 0,47. Teraz mnożąc odpowiednie prawdopodobieństwa gamet otrzy mamy prawdopodobieństwa wystąpienia określonych kombinacji genetycznych. Sumowanie tych, które dają taki sam fenotyp pozwoli sprawdzić, czy uzyskane wyniki pokrywają się z obliczeniami teoretycznymi (por. Ryc. 97).
Analiza sprzężeń sprawia uczniom poważne kłopoty, dlatego proponuję pewne techniczne ułatwienie, które poprawia poglądowość schematów — zapis ułamkowy (por. Ryc. 98). Jeśli analizowane geny są niesprzężonc, „rozpisuje się” je jako niezależne ułamki, w przypadku sprzężenia — w jednym ułamku. Dodatkowe ułatwienie polega na tym, że wówczas od razu widać, które układy alleli będą niezrekombinowane, a które zrekombinowane.
147
Wyszukiwarka
Podobne podstrony:
0000020(1) GENETYKA genu nieallcliczncgo naci drugim — cpistnzę (cpistazję). W opisywanym przykładzi
0000020(2) GENETYKA niektóre: geny dominujące wykazują zróżnicowaną penetrację We wszystkich opisywa
0000021(2) GENETYKA być może zbyt długi okres zahamowania powoduje (daje czas na?) kumulowanie się b
0000021 3 GENETYKA (5) KOD GENETYCZNY JEST KOLINEARNY Matryca złożona jest z kolejnych trójek ułożon
0000022(2) GENETYKA PODSUMOWANIE: 1. Badania genetyczne człowieka są kłopotliwe z
0000022 3 GENETYKA ncgo. Często więc poznanie istoty formowania danej cechy lub cech jest bardzo tru
0000023 2 GENETYKA GENETYKA OH * A C C wolny koniec 3 -OH.do którego przyłączany jest aminokwas koni
0000024(1) GENETYKA P: AB Y ab P. -A x a b P: Ab x aB AB A ab AB
0000024 3 GENETYKA przez chwilę zastanowisz się, zwrócisz zapewne uwagę, że rozpoznawanie matrycy pr
0000025(2) GENETYKA 1. W takiej populacji prawdopodobieństwo skojarzenia się z jakimkolwiek osobniki
0000025 2 GENETYKA ;rejon promruorowy! : W** rejoo crarukrjbow any ; mmŁryp-J <}> DNA ilUKfon
0000026(2) GENETYKA gli określić jc także dla drugiego. Znając prawdopodobieństwo (praktycznie częst
0000026 3 GENETYKA Elongacja łańcucha mRNA przebiega zasadniczo podobnie jak u bakterii. Z kolei ter
0000027(2) GENETYKA W MAŁYCH POPUL/iCJACH WYSTĘPUJE ZJAWISKO DRYFTU GENETYCZNEGO Dryft genetyczny (m
0000027 3 GENETYKA3. 2. 2. Translacja Zacznijmy od przypomnienia, że dla określenia budowy przestrze
0000028(2) genetyka12. Znaczenie genetyki ZNACZENIE GENETYKI SYSTEMATYCZNIE ROŚNIE Człowiek juz od b
0000028 3 GENETYKA Mg-‘ AUGGAAGGAAGU Ryc 32 A. Translacja — etap cjacji (opis w tekście) Polega to n
0000029(1) GENETYKA 1. Aktywność potowych (już zsyntetyzowanych) białek: 2.
0000029(2) GENETYKA KAJAK). Określenie sekwencja palindromowa bierze się siad. ze ułożenie nukJeotyd
więcej podobnych podstron